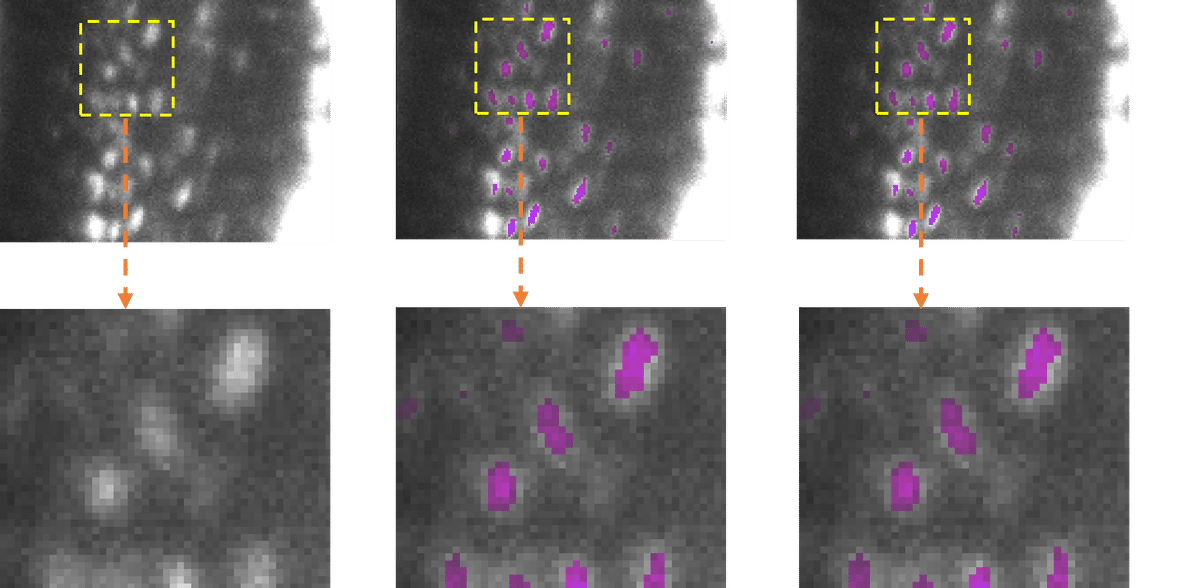
Analyse von Mikroskopaufnahmen: Neue Open-Source-Software macht KI-Modelle leichter & grüner
Keine speziellen Kenntnisse notwendig
künstliche Intelligenz (KI) hat sich als unverzichtbare Komponente der Auswertung mikroskopischer Daten etabliert. Doch während die KI-Modelle immer besser und komplexer werden, steigt auch der Rechenaufwand – und damit einhergehend der Energieverbrauch. Forschende des Leibniz-Instituts für Analytische Wissenschaften (ISAS) und der Universität Peking haben deswegen eine kostenfreie Kompressionssoftware entwickelt, mit der Wissenschaftler:innen bereits bestehende Bioimaging-KI-Modelle schneller und mit wesentlich geringerem Energieaufwand ausführen können. Ihre nutzerfreundliche Toolbox namens EfficientBioAI (Open Source) haben die Forschenden nun im Fachjournal Nature Methods vorgestellt.
Moderne Mikroskopieverfahren produzieren eine Vielzahl hochauflösender Bilder, einzelne Datensätze können tausende davon umfassen. Um die Datenmengen zuverlässig analysieren zu können, nutzen Wissenschaftler:innen häufig KI-gestützte Software. Mit immer komplexeren KI-Modellen kann sich die Latenzzeit (Verarbeitungszeit) für Bilder jedoch deutlich erhöhen. „Eine hohe Netzwerklatenz, beispielsweise bei besonders großen Bildern, führt zu einer höheren Rechenleistung und schließlich zu einem gesteigerten Energieverbrauch“, sagt Dr. Jianxu Chen, Leiter der Nachwuchsgruppe AMBIOM – Analysis of Microscopic BIOMedical Images am ISAS.
Neue Anwendungsgebiete für eine bekannte Technik
Um hohe Latenzen bei der Bildanalyse zu vermeiden, insbesondere bei Geräten mit begrenzter Rechenleistung, verwenden die Forschenden komplexe Algorithmen und komprimieren damit die KI-Modelle. Das heißt, sie reduzieren die Menge der Berechnungen in den Modellen, während diese eine vergleichbare Vorhersagegenauigkeit beibehalten. „Modellkomprimierung ist eine Technik, die bei der digitalen Bildverarbeitung, der sogenannten Computer Vision, und der KI weit verbreitet ist, um Modelle leichter und grüner zu machen“, erklärt Chen. Dabei kombinieren Forschende verschiedene Strategien, um den Speicherverbrauch zu reduzieren, die Modellinferenz, also den „Denkprozess“ des Modells zu beschleunigen – und somit Energie zu sparen. Zum Einsatz kommt beispielsweise Pruning, das überflüssige Knoten aus dem neuronalen Netzwerk entfernt. „In der Bioimaging-Gemeinschaft sind diese Techniken häufig unbekannt. Daher wollten wir eine gebrauchsfertige und einfache Lösung entwickeln, um sie auf gängige KI-Tools beim Bioimaging anzuwenden“, sagt Yu Zhou, Erstautor der Publikation und Doktorand bei AMBIOM.
Bis zu ca. 81 Prozent Energieeinsparung
Um ihre neue Toolbox auf die Probe zu stellen, haben die Forschenden um Chen ihre Software an mehreren realen Anwendungen getestet. Bei unterschiedlicher Hardware und verschiedenen Bioimaging-Analyseaufgaben konnten die Komprimierungstechniken die Latenzzeit erheblich verringern und den Energieverbrauch zwischen 12,5 bis 80,6 Prozent senken. „Unsere Versuche haben gezeigt, dass EfficientBioAI die Effizienz neuronaler Netzwerke bei Bioimaging-Analysen deutlich erhöhen kann, ohne die Genauigkeit der Modelle einzuschränken“, resümiert Chen. Die Energieeinsparungen verdeutlicht er am Beispiel des allgemein verwendeten CellPose-Modells: Würden eintausend Nutzer:innen die Toolbox nutzen, um das Modell zu komprimieren und auf den Jump-Target-ORF-Datensatz (etwa eine Million Mikroskopbilder von Zellen) anwenden, könnten sie Energie einsparen, die circa den Emissionen einer Autofahrt von etwa 7.300 Meilen (ca. 11.750 Kilometer) entsprechen.
Keine speziellen Kenntnisse notwendig
Die Autor:innen legen viel Wert darauf, EfficientBioAI möglichst vielen Wissenschaftler:innen in der biomedizinischen Forschung zugänglich zu machen. Forschende können die Software installieren und nahtlos in bereits existierende PyTorch-Bibliotheken (Open-Source-Programmbibliothek für die Programmiersprache Python) integrieren. Für einige sehr verbreitete Modelle wie etwa Cellpose, können Forschende die Software nutzen, ohne selbst etwas am Code ändern zu müssen. Für spezifische Änderungswünsche stellt die Gruppe außerdem mehrere Demos und Tutorials zur Verfügung. Mit nur ein paar geänderten Code-Zeilen lässt sich die Toolbox dann auch für angepasste KI-Modelle verwenden.
Originalveröffentlichung
Meistgelesene News
Originalveröffentlichung
Yu Zhou, Jiajun Cao, Justin Sonneck, Sweta Banerjee, Stefanie Dörr, Anika Grüneboom, Kristina Lorenz, Shanghang Zhang, Jianxu Chen; "EfficientBioAI: making bioimaging AI models efficient in energy and latency"; Nature Methods, 2024-1-24
Themen
Organisationen
Weiterführender Link
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

Limsophy LIMS von AAC Infotray
Optimieren Sie Ihre Laborprozesse mit Limsophy LIMS
Nahtlose Integration und Prozessoptimierung in der Labordatenverwaltung
GUS-OS Suite von GUS
Ganzheitliche ERP-Lösung für Unternehmen der Prozessindustrie
Integrieren Sie alle Abteilungen für nahtlose Zusammenarbeit

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.
Meistgelesene News
Weitere News von unseren anderen Portalen
Zuletzt betrachtete Inhalte

Wie COVID-19 das Immunsystem verändert - Bisher unbekannten Effekt auf das Immunsystem entdeckt: Nach COVID-19 anfälliger für Sekundärinfektionen?
HELLP-Syndrom
Neuer Sensor misst Arsengehalt im Trinkwasser - Ausgründungsprojekt startet 2010
Röntgenaugen blicken tiefer in tödliche Krankheitserreger
Intrinsische_Aktivität
Glycolipide