Die Ausbreitung von Krebs durch ein Computerfenster sehen
Computermodell ermöglicht die Simulation zellulärer Interaktionen über noch nie dagewesene Entfernungen im menschlichen Blutgefäßsystem
Biomedizintechniker der Duke University haben die Fähigkeiten eines Rechenmodells, das die Bewegung einzelner Krebszellen über große Entfernungen im gesamten menschlichen Körper simuliert, erheblich verbessert.
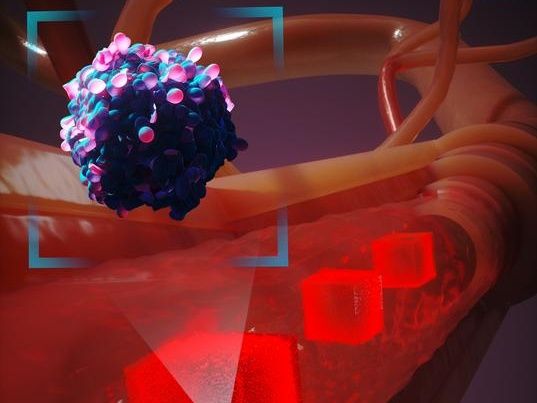
Eine künstlerische Darstellung, wie ein neues Computermodellierungssystem eine Krebszelle auf ihrem Weg durch den menschlichen Körper simuliert. Um Millionen von zellulären Interaktionen zu berücksichtigen, werden nur detaillierte Simulationen von Zellen in der unmittelbaren Umgebung der Krebszelle erstellt, während sie im Blutkreislauf zirkuliert.
Duke University
Der Ansatz mit der Bezeichnung "Adaptive Physics Refinement (APR)" erfasst detaillierte zelluläre Interaktionen und deren Auswirkungen auf die zelluläre Flugbahn und bietet unschätzbare Einblicke in die Wanderungen von metastasierenden Krebszellen.
"Krebszellen in unserem Blutkreislauf werden dadurch beeinflusst, dass sie an roten Blutkörperchen in der Nähe anstoßen und sich um diese herum bewegen, sowie durch andere zelluläre Interaktionen", sagt Aristoteles Martin, Doktorand im Labor von Amanda Randles am Duke Biomedical Engineering. "Aber es ist unmöglich, die Bewegung aller roten Blutkörperchen in allen Blutgefäßen des Körpers zu simulieren, also mussten wir einen Weg finden, diese rechnerischen Einschränkungen zu umgehen.
Die Forschungsergebnisse wurden am 15. November 2023 auf der International Conference for High Performance Computing, Networking, Storage, and Analysis (SC23) vorgestellt. Diese Konferenz ist das wichtigste internationale Treffen im Bereich des Hochleistungsrechnens und wird von ACM und IEEE-CS gesponsert.
Die Entschlüsselung der Dynamik, mit der sich Krebszellen durch die Blutgefäße des Körpers bewegen, ist nach wie vor ein kritisches und komplexes Thema in der Krebsforschung, das für die Früherkennung und eine mögliche gezielte Behandlung entscheidend ist. Die Untersuchung dieser Vorgänge am lebenden Patienten ist jedoch nicht möglich und erfordert stattdessen fortschrittliche Computermodelle zur Simulation der Dynamik von Krebszellen.
Amanda Randles, Alfred Winborne und Victoria Stover Mordecai Associate Professor für Biomedizinische Wissenschaften an der Duke University, entwickelt und verbessert seit über einem Jahrzehnt Berechnungsmethoden zur Erforschung dieser grundlegenden Prozesse. Einer ihrer bemerkenswerten Beiträge ist HARVEY, ein hoch skalierbares Hämodynamik-Simulationspaket, das für den Betrieb auf den modernsten Supercomputern der Welt entwickelt wurde.
Aber auch Supercomputer haben ihre Grenzen.
Um die Flugbahn einer einzelnen Krebszelle zu berechnen, müssen die Modelle ihre mikroskopischen Wechselwirkungen mit den umgebenden roten Blutkörperchen erfassen. Der menschliche Körper besteht jedoch aus etwa 25 Billionen roten Blutkörperchen und fünf Litern Blut. Mit den größten Supercomputern, die heute zur Verfügung stehen, können moderne Modelle nur einen Bereich nachbilden, der ein Prozent dieses Volumens in zellulärer Auflösung enthält - ein begrenzter Bereich, der immer noch mehrere hundert Millionen rote Blutkörperchen umfasst.
Um dieses Problem zu umgehen, hat ein großes Team mit Mitarbeitern des Lawrence Livermore National Laboratory (LLNL) und des Oak Ridge National Laboratory (ORNL) unter der Leitung von Sayan Roychowdhury, einem ehemaligen Doktoranden im Randles-Labor, einen neuen Ansatz gewählt. Durch die Erweiterung des bestehenden Algorithmus des Labors um die Interaktion mit Millionen benachbarter roter Blutkörperchen erstellt APR ein hochauflösendes Fenster, das die Zelle von Interesse auf ihrem Weg durch das Gefäßsystem verfolgt.
Ein Teil des Tricks, damit dieser Ansatz funktioniert, ist die Kopplung des sich bewegenden Fensters mit einer Simulation des gesamten Gefäßbereichs, wobei das Blut als Massenflüssigkeit modelliert wird. Dieser Ansatz ist vergleichbar mit der Simulation des Verhaltens eines Spielzeugboots, das auf einem Fluss treibt. Der einfachste Ansatz wäre, die gesamte Wasserstraße von Anfang bis Ende mit der höchsten Auflösung zu modellieren. In einem solchen Szenario würde jedoch der größte Teil der Rechenressourcen genau die physikalischen Phänomene erfassen, die weit entfernt vom Boot auftreten, was bestenfalls unpraktisch wäre.
Stattdessen ist es viel effizienter, die Region in der Nähe des Bootes so genau wie möglich zu modellieren, während der Rest des Stroms mit einer geringeren Auflösung modelliert wird. Wenn sich das Boot komplexen Merkmalen wie Felsen, Strudeln und Stromschnellen nähert, würde das Modell diese genau erfassen, sobald sie nahe genug sind, um einen messbaren Einfluss auf die Flugbahn des Bootes zu haben. Das Ergebnis ist eine genaue Simulation, die weitaus effizienter ist und es der Simulation ermöglicht, viel längere Flussabschnitte zu erkunden.
"Die meisten Zellen, die wir jemals auf einmal simuliert haben, sind 580 Millionen", sagt Samreen Mahmud, Doktorandin im Randles-Labor. "Unser Ziel war es, die Fenstergröße zu maximieren, um zu sehen, wie viele Zellen wir mit einem Supercomputer der Spitzenklasse erfassen können. Anschließend haben wir uns darauf konzentriert, die Rechenkosten zu senken und die Methode effizient in die Cloud zu verlagern."
Die Effizienz des Algorithmus bei der Durchführung groß angelegter Simulationen wurde in der Studie durch die Simulation des Transports einer Krebszelle über einen Zentimeter mit nur einem einzigen Knoten auf Amazons Web Services (AWS) für 500 Stunden gezeigt. Durch den Einsatz von Adaptive Physics Refinement konnte der Bedarf an Rechenspeicher drastisch von Petabytes auf handlichere Gigabytes reduziert werden.
Die Ergebnisse, so die Forscher, könnten für andere Labors, die Krebs erforschen oder biomedizinische Geräte entwickeln, eine entscheidende Rolle spielen. Sie könnten beispielsweise Forschern helfen, die mechanische und physikalische Seite der Krebsmetastasierung zu verstehen, indem sie Faktoren isolieren, die in einem Versuchsaufbau nur schwer oder gar nicht zu erfassen wären. Es stellt auch einen bedeutenden Sprung in den Fähigkeiten des Hochleistungsrechnens dar und erleichtert die praktische Anwendung der Modellierung großer Mengen roter Blutkörperchen mit begrenzter Kapazität.
Für die Zukunft plant das Team, seine Modellierungssoftware um weitere Funktionen zu erweitern, z. B. um die Adhäsion zwischen Zellen und Veränderungen im Zellverhalten in der Nähe von Blutgefäßwänden. Sie wollen auch untersuchen, wie sich Cluster von Krebszellen durch das Gefäßsystem bewegen, da klinische Studien gezeigt haben, dass die Bewegung in Gruppen das Potenzial von metastatischen Zellen zur Bildung neuer Tumore erhöht.
"Wir hoffen, dass Methoden wie APR dazu beitragen werden, die Computermodellierung auf zellulärer Ebene zu demokratisieren, indem sie das Spielfeld ebnen", sagte Randles, "Wir wollen es Forschern ohne Zugang zu den größten Supercomputern der Welt ermöglichen, Berechnungsansätze zur Untersuchung der Krebsdynamik zu nutzen."
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.
Originalveröffentlichung
Sayan Roychowdhury, Samreen T. Mahmud, Aristotle Martin, Peter Balogh, Daniel F. Puleri, John Gounley, Erik W. Draeger, Amanda Randles; "Enhancing Adaptive Physics Refinement Simulations Through the Addition of Realistic Red Blood Cell Counts"; Proceedings of the International Conference for High Performance Computing, Networking, Storage and Analysis, 2023-11-11