Wie sich Krebsgene selbständig machen
Studie wirft Licht auf die mysteriöse Evolution von DNA-Ringen
Tumore verhalten sich manchmal eigenartig: Sie wachsen außergewöhnlich stark oder werden plötzlich gegen ein Krebsmedikament resistent. Dieses Verhalten lässt sich häufig darauf zurückführen, dass sich Krebsgene aus den Chromosomen der Zelle herauslösen und in Ringform „selbständig machen“. Wenig ist bisher darüber bekannt, wie genau diese DNA-Ringe entstehen und wie sie sich im Verlauf des Tumorwachstums weiterentwickeln. Mit einer neuen Methode hat ein internationales Forschungsteam unter Leitung der Charité – Universitätsmedizin Berlin und des Max Delbrück Centers diesen Weg jetzt bei dem Neuroblastom nachgezeichnet. Die Ergebnisse sind im Fachmagazin Nature Genetics veröffentlicht.
Sie gelten als eine der größten Herausforderungen in der Krebsforschung: DNA-Ringe – also kleine Erbgut-Schlaufen, die zu Hunderten abseits der Chromosomen im Zellkern schwimmen. Bereits seit 1965 bekannt, stellen sie Forschende noch immer vor viele Fragen. Wo kommen all diese Ringe her? Welche Funktion haben sie? Wie wirken sie sich auf die Zelle und den Organismus aus? Klar ist: Nahezu ein Drittel aller Tumore bei Kindern und Erwachsenen tragen in ihren Zellen DNA-Ringe – und diese sind fast immer besonders aggressiv. Auch wenn ein Tumor gegen ein zuvor wirksames Medikament resistent wird, ist das oft auf ringförmige DNA zurückzuführen. Mit der Erforschung dieser speziellen Form der Erbinformation verbinden Wissenschaftler:innen weltweit deshalb die Hoffnung auf neue Therapieansätze gegen Krebs. Allerdings: Nicht immer wirkt sich die „extrachromosomale zirkuläre DNA“ negativ auf das Krebswachstum aus. Manche Ringe scheinen auch harmlos zu sein.
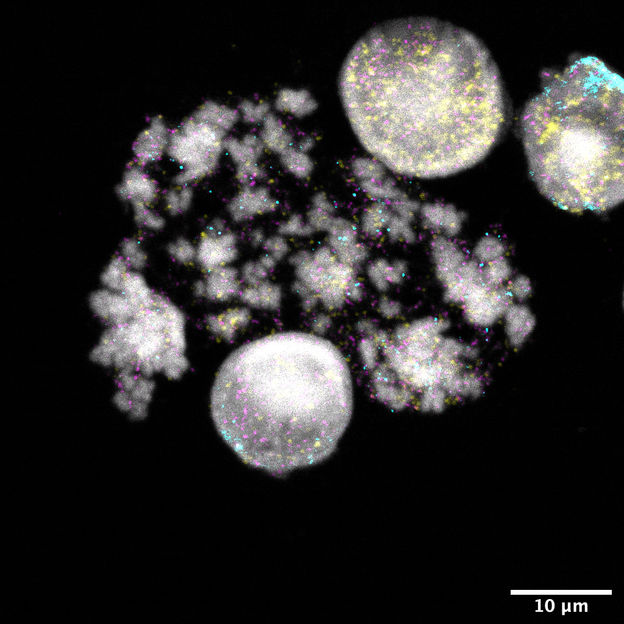
Kerne und Chromosomen von Neuroblastomzellen. DNA-Ringe sind gelb, türkis oder magenta angefärbt. Jede Farbe signalisiert unterschiedliche Krebsgene.
Rocío Chamorro González, Charité
„Um die gefährlichen von den harmlosen DNA-Ringen zu unterscheiden und ihre Evolution innerhalb des Tumors nachvollziehen zu können, muss man sich das Gewebe Zelle für Zelle anschauen“, erklärt der Leiter der Studie Prof. Dr. Anton Henssen. Der Mediziner ist an der Klinik für Pädiatrie mit Schwerpunkt Onkologie und Hämatologie der Charité tätig und forscht am Experimental and Clinical Research Center (ECRC), einer gemeinsamen Einrichtung der Charité und des Max Delbrück Centers. Zusammen mit seinem Team hat er jetzt eine Technologie entwickelt, die für jede einzelne Zelle den genetischen Code der vorhandenen DNA-Ringe auslesen kann. Sie gibt gleichzeitig Auskunft darüber, welche Gene darauf aktiv sind. „So können wir einfach auszählen, wie viele Zellen des Tumors einen spezifischen Ring beherbergen“, sagt Prof. Henssen. „Sind es wenige, ist der Ring nicht besonders relevant für das Krebswachstum. Sind es viele, verleiht er einer Tumorzelle offenbar einen Selektionsvorteil.“
Welche DNA-Ringe treiben das Tumorwachstum an?
Die neue Methode nutzten die Wissenschaftler:innen zunächst, um eine Bestandsaufnahme aller DNA-Ringe bei kultivierten Neuroblastomzellen zu machen. Das Neuroblastom ist eine Krebserkrankung, die vor allem sehr junge Kinder betrifft und als besonders bösartig gilt. Das Ergebnis der Untersuchungen: Keine Krebszelle ist wie die andere – während in einer 100 DNA-Ringe schwimmen, können es in der nächsten 2.000 sein. Auch sind die Ringe sehr unterschiedlich groß: Die Winzlinge unter ihnen bestehen nur aus 30, die Riesen aus über einer Million genetischen Bausteinen.
„Die großen DNA-Ringe sind beladen mit Krebsgenen, die ursprünglich aus den Chromosomen der Zelle stammen“, erklärt Rocío Chamorro González. Sie ist die Erstautorin der Studie und forscht ebenfalls an der Klinik für Pädiatrie mit Schwerpunkt Onkologie und Hämatologie der Charité sowie am ECRC. „Durch die Ringform umgehen sie die klassischen Gesetze der Genetik – und werden ein Stück weit autonom. Diese Krebsgene haben sich sozusagen selbständig gemacht. Welche Konsequenzen das hat, beginnen wir gerade erst zu verstehen. In unserer Studie haben wir die großen DNA-Ringe in vielen Neuroblastomzellen gefunden, sie treiben das Zellwachstum also offenbar an. Die kleinen Ringe haben wir nur vereinzelt entdeckt, sie haben für die Krebszellen wohl keine große Relevanz.“
Die Evolution eines unabhängigen Krebsgens
Um nachzuvollziehen, wie ein „autonomes Krebsgen“ eigentlich entsteht und sich innerhalb eines Tumors weiterentwickelt, analysierte die Forschungsgruppe im zweiten Schritt beispielhaft das Neuroblastom in jungen Patient:innen – und zwar Zelle für Zelle. Die Ergebnisse legen nahe, dass sich zu Beginn des Tumorwachstums in diesem Fall zunächst das bekannte Krebsgen MYCN aus seinem Heimat-Chromosom herauslöste und einen Ring bildete. Anschließend verschmolzen zwei dieser Ringe zu einem größeren, der wiederum einen kürzeren und dann einen längeren Abschnitt verlor. „Erst der letzte Ring scheint einen Wachstumsvorteil mit sich gebracht zu haben, weil nur er in vielen Zellen des Neuroblastoms zu finden ist“, sagt Prof. Henssen. „Das zeigt, dass sich das Krebsgen durch diese Vorgänge nicht nur selbständig gemacht, sondern auch immer weiter ‚verbessert‘ hat.“
Ein solcher Einblick in die Evolution von DNA-Ringen innerhalb eines Tumors wäre ohne die neu entwickelte Methode nicht möglich gewesen. Das Forschungsteam wird sie nun nutzen, um bei weiteren Krebsfällen die Entwicklungsschritte zu rekonstruieren. So wollen die Wissenschaftler:innen künftig noch besser in der Lage sein, die gefährlichen von den harmlosen DNA-Ringen zu unterscheiden. „Unsere Hoffnung ist, dass wir in Zukunft durch einen Blick auf die DNA-Ringe im individuellen Fall erkennen können, ob der Tumor besonders aggressiv ist oder nicht“, sagt Prof. Henssen. „Dann könnten wir die Therapie daran anpassen. Die Vorhersagekraft von spezifischen DNA-Ringen zu testen, ist deshalb unser nächstes Forschungsziel.“





















































