Protein in Schwarzarbeit macht Krankenhauskeim erst gefährlich
Forschungsteam findet möglichen Ansatzpunkt für neue Therapien bei Infektionen mit Pseudomonas aeruginosa
Das Bakterium Pseudomonas aeruginosa ist ein weit verbreiteter gefährlicher Krankenhauskeim. Er befällt Atemwege und Lunge und ist von Natur aus gegen zahlreiche Antibiotika resistent. Um künftig besser gegen ihn vorgehen zu können, wird zum Beispiel nach sogenannten Pathoblockern gesucht. Dabei steht nicht wie bei einer antibiotischen Behandlung das Abtöten des Erregers im Fokus, sondern das gezielte Ausschalten oder Abschwächen seiner krankmachenden Wirkung. Bei einer Infektion mit Pseudomonas aeruginosa ist es unter anderem das blaugrünliche Stoffwechselprodukt Pyocyanin, das zur Entstehung entzündlicher Prozesse beiträgt und gewebeschädigend wirkt. Könnte man seine Herstellung mittels eines Pathoblockers verhindern, wäre eine Infektion weniger schwerwiegend. Doch dafür müssen zunächst die genauen molekularen Mechanismen verstanden werden, die innerhalb der Bakterienzelle für die Herstellung von Pyocyanin verantwortlich sind. Dem Forschungsteam um Prof. Wulf Blankenfeldt, Leiter der Abteilung „Struktur und Funktion der Proteine“ am Helmholtz-Zentrum für Infektionsforschung (HZI) in Braunschweig, ist es nun mithilfe moderner proteinbiochemischer Analyseverfahren gelungen, das Zusammenspiel von Proteinen, die für die Produktion von Pyocyanin notwendig sind, im Detail weiter aufzudecken. Dabei hat er die zentrale Funktion eines Proteins namens PqsE als „Schwarzarbeiter“ aufklären können. Die Studie ist in der aktuellen Ausgabe des Fachmagazins Nature Communications erschienen.
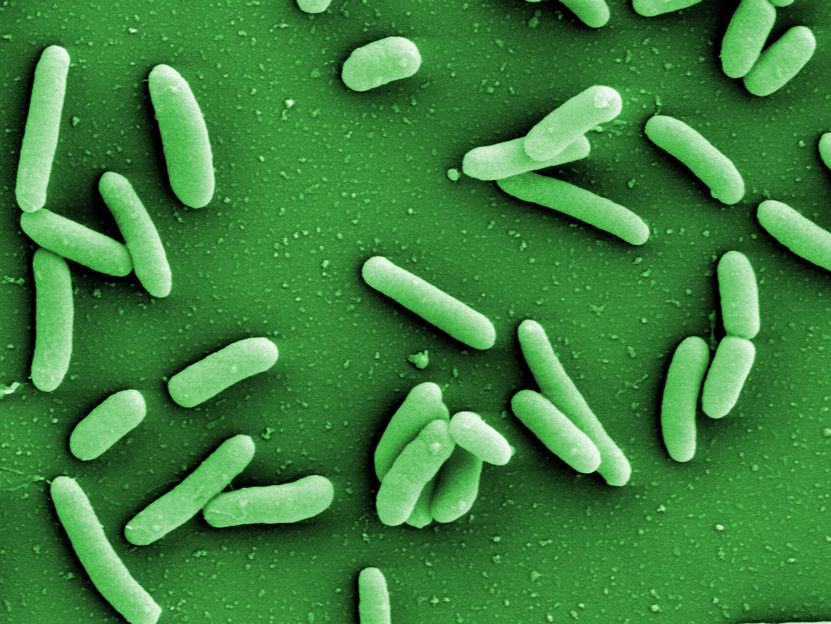
Elektronenmikroskopische Aufnahme von Pseudomonas aeruginosa
HZI/Manfred Rohde
Wulf Blankenfeldt erklärt im Interview, was das Bakterium Pseudomonas aeruginosa so gefährlich macht, warum ihn die Funktion von PqsE über zwei Jahrzehnte nicht losgelassen hat – und wie er die Schwarzarbeit dieses Proteins zur Anzeige bringen konnte.
Herr Professor Blankenfeldt, wo kann man sich mit Pseudomonas aeruginosa infizieren und für wen ist eine Infektion besonders gefährlich?
Wulf Blankenfeldt: Mit diesem weit verbreiteten bakteriellen Erreger kann man sich nahezu überall infizieren. Er mag feuchte Umgebung – und da gibt es eben ziemlich viele Orte, wo er vorkommen kann: in der Natur etwa in Böden oder Gewässern. Aber er fühlt sich auch in Leitungswasser, Waschbecken, Spülmaschine, Dusche oder Toilette wohl, wo man sich bei nicht hundertprozentigen Hygienemaßnahmen leicht infizieren kann. Und er ist auch von Mensch zu Mensch übertragbar. Für Personen mit geschwächtem Immunsystem oder schweren Atemwegserkrankungen wie Mukoviszidose oder chronisch obstruktiver Lungenerkrankung (COPD) kann eine Infektion tatsächlich sehr gefährlich und mitunter lebensbedrohlich sein. Als Krankenhauskeim hat es Pseudomonas aeruginosa daher leider zu trauriger Berühmtheit gebracht.
Warum wirken bei ihm viele Antibiotika nicht?
Pseudomonas aeruginosa kann Fremdstoffe wie Medikamente gezielt aus seinem Zellinneren ausschleusen und gehört außerdem zu den Bakterien, die einen sogenannten Biofilm ausbilden. Die Bakterien sind dann von einer Art Schleimschicht umgeben, die ihnen Schutz bietet – unter anderem eben leider auch vor Antibiotika. Daher ist Pseudomonas aeruginosa von Natur aus gegen eine große Zahl von Antibiotika resistent und schwer zu bekämpfen. Die Entwicklung effektiver Pathoblocker, die die Schlagkraft des Erregers eindämmen oder ausschalten könnten, wäre hier tatsächlich ein Segen.
Günstig wäre es, das gewebeschädigende Stoffwechselprodukt Pyocyanin auszuschalten, das der Erreger herstellt. Welche Rolle spielen dabei die in Ihrer Studie untersuchten Proteine?
Pyocyanin wird erst gebildet, wenn die für die Herstellung erforderlichen Gene abgelesen werden. Und dieser Ableseprozess wird durch ein Zusammenspiel verschiedener Proteine kontrolliert. Im Zentrum steht dabei ein Protein mit dem Kurznamen RhlR. Bekannt war bereits, dass RhlR die Genablesung erst startet, wenn zuvor das Signalmolekül C4-HSL den Startschuss gibt und an RhlR bindet. Doch ob und vor allen Dingen wie in dieser Signalkaskade womöglich noch weitere Proteine ihre Finger mit im Spiel haben, war bisher nicht richtig verstanden.
Aber Sie hatten schon einen Verdächtigen im Visier…
(lacht) Genau – sogar einen Langzeitverdächtigen! Bereits vor rund 20 Jahren haben mein Team und ich angefangen, uns mit einem Protein namens PqsE zu beschäftigen, das mit der Erzeugung von Pyocyanin in Verbindung gebracht wurde. Wir konnten damals unter anderem seine Kristallstruktur aufklären. Und vor etwa fünf Jahren konnten wir dann in Laboruntersuchungen erstmals Hinweise dafür gewinnen, dass das eigentlich recht instabile Protein RhlR durch Bindung an PqsE stabilisiert wird. Dass PqsE damit womöglich auch direkt in die Aktivierung der Pyocyanin-Gene involviert sein könnte, war für mich seither naheliegend – aber ich konnte es ihm nicht nachweisen.
In Ihrer aktuellen Studie in Nature Communications ist es Ihnen nun gelungen!
Ja, endlich! Und zwar auch mithilfe hochmoderner computergestützter Methoden, die erst vor kurzer Zeit entwickelt wurden. Früher wäre das so gar nicht möglich gewesen. Wir haben am Computer mit einem Programm aus Israel ein künstliches RhlR-Protein entworfen, das von sich aus stabil sein und gleichzeitig seine volle Funktion bei der Genablesung behalten sollte. Den DNA-Bauplan dafür haben wir dann von einer Firma herstellen lassen und anschließend das künstliche Protein bei uns untersucht. In unserer Studie haben wir getestet, ob das stabilisierte, synthetische RhlR auch in Abwesenheit von PqsE in der Lage ist, die Genablesung zur Herstellung von Pyocyanin zu starten. Und siehe da: Genau das war der Fall! In Vergleichsversuchen dagegen konnte das natürliche RhlR in Abwesenheit von PqsE die Gene nicht ablesen, und es wurde kein Pyocyanin gebildet. Damit konnten wir zeigen, dass für den Ableseprozess ein stabiles RhlR notwendig ist – und dass dafür das Protein PqsE verantwortlich ist, indem es sich mit RhlR zu einem Komplex zusammentut.
PqsE hat Sie und Ihre internationalen Forschungskollegen zuvor aber ziemlich aufs Glatteis geführt...
Ja, das stimmt! PqsE wurde aufgrund seiner in Laborversuchen nachgewiesenen Eigenschaften als Enzym – das sind Proteine, die (bio)chemische Prozesse beschleunigen – in die Gruppe der sogenannten Thioesterasen eingeteilt. Daher vermutete man eigentlich, dass seine Fähigkeiten als Enzym auch im Zusammenhang mit Pyocyanin in irgendeiner Weise zum Tragen kommen. Doch die stabilisierende Wirkung, die PqsE auf RhlR ausübt, hat überhaupt nichts mit seiner Fähigkeit als Enzym zu tun. PqsE hat uns da tatsächlich ziemlich lange aufs Glatteis geführt. Doch seinen Schwarzarbeiterjob als Stabilisator von RhlR konnten wir nun zum Glück zur Anzeige bringen – und können ihn künftig vielleicht sogar unterbinden.
Wie geht es nun weiter – werden die Proteine von Pseudomonas aeruginosa Sie weiterhin beschäftigen?
Auf jeden Fall. Sie haben mich zwanzig Jahre nicht losgelassen, und ich schätze, es werden noch viele weitere Jahre werden – und jetzt wird es ja auch erst richtig spannend. Mit modernen Methoden wie der Proteinkristallographie konnten wir in unserer Studie ja auch den RhlR-PqsE-Proteinkomplex in 3D darstellen. Das ist eine hervorragende Grundlage, um gemeinsam mit unseren Kolleginnen und Kollegen am HZI nach Wirkstoffen zu fahnden, die diese Interaktion zielgerichtet stören und als Pathoblocker eingesetzt werden könnten. Wir arbeiten bereits an einem Screeningverfahren und hoffen, dass wir mit unserer zukünftigen Forschung weiter dazu beitragen können, Ansätze für Therapien zu finden, die bei Pseudomonas-Infektionen erfolgreich eingesetzt werden können.
Originalveröffentlichung
Sebastian Roman Borgert, Steffi Henke, Florian Witzgall, Stefan Schmelz, Susanne zur Lage, Sven-Kevin Hotop, Steffi Stephen, Dennis Lübken, Jonas Krüger, Nicolas Oswaldo Gomez, Marco van Ham, Lothar Jänsch, Markus Kalesse, Andreas Pich, Mark Brönstrup, Susanne Häussler & Wulf Blankenfeldt: Moonlighting chaperone activity of the enzyme PqsE contributes to RhlR-controlled virulence of Pseudomonas aeruginosa. Nature Communications 2022