Eine neue Methode zur Untersuchung der Ribosomenfunktion
UIC-Wissenschaftler verwenden native chemische Ligation, um Peptide an tRNAs zu binden
In winzigen Zellmaschinen, den so genannten Ribosomen, werden Ketten von genetischem Material, die so genannten Boten-RNAs (mRNAs), mit den entsprechenden Transfer-RNAs (tRNAs) zusammengebracht, um Sequenzen von Aminosäuren zu erzeugen, die das Ribosom als Proteine verlassen. Unfertige Proteine werden als naszierende Ketten bezeichnet und bleiben am Ribosom hängen.
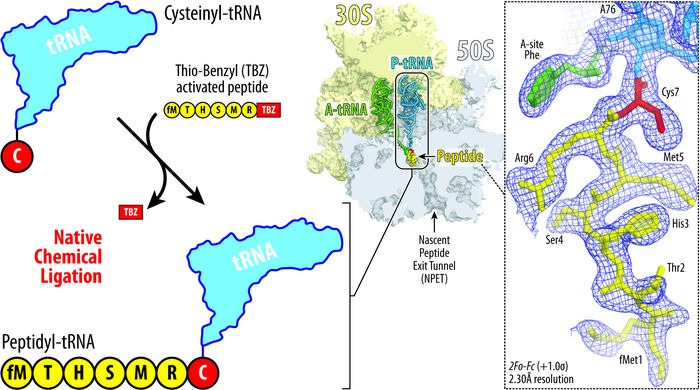
Die Abbildung zeigt das Prinzip des von Syroegin et al. entwickelten nativen chemischen Ligationsansatzes. Durch Hinzufügen der Aminosäure Cystein (rot) zur tRNA (blau, oben links) kann die tRNA mit einem Peptid (gelb, unten links) verschmelzen. Die resultierende Ribosomenstruktur (Mitte) und die eingefangenen Elektronendichtekarten für die Peptidyl-tRNA innerhalb des Ribosoms (rechts) wurden durch Röntgenkristallographie in den UIC-Experimenten gewonnen.
Syroegin, et al.
Wissenschaftler wissen, dass einige dieser naszierenden Ketten die Aktivität des Ribosoms regulieren können und dass die naszierenden Ketten manchmal mit Antibiotika interferieren können - von denen viele auf die bakterielle Ribosomaktivität abzielen. Die Wissenschaftler wissen nicht, warum dies geschieht, vor allem, weil es schwierig ist, die Wechselwirkungen zwischen Ribosom, Peptid und Medikament sichtbar zu machen, solange die unfertigen Proteine noch an das Ribosom gebunden sind.
Nun haben Wissenschaftler der University of Illinois Chicago als erste eine Methode zur stabilen Bindung von Peptiden an tRNAs vorgestellt, die es ihnen ermöglicht hat, neue grundlegende Erkenntnisse über die Funktion von Ribosomen zu gewinnen, indem sie die Strukturen von Ribosomen auf atomarer Ebene und die Formen, die diese Peptide innerhalb des Ribosoms annehmen, bestimmt haben.
"Die Herausforderung bestand darin, die Struktur des Ribosoms und des Ausgangstunnels in Gegenwart der naszierenden Peptide aus der Nähe zu betrachten, da das Ribosom in der Natur sehr schnell ist, um Bilder aufzunehmen oder Experimente durchzuführen", sagte Yury Polikanov, außerordentlicher Professor in der Abteilung für Biowissenschaften am College of Liberal Arts and Sciences. "Bis zum Aufkommen dieser neuen Methode war es uns im Grunde genommen verwehrt, zu sehen, was im aktiven Bereich des Ribosoms in diesem kritischen Moment passiert".
Polikanov und sein Kollege Egor Syroegin, ein Doktorand der Biowissenschaften an der UIC, verwendeten eine Methode, die als native chemische Ligation bezeichnet wird, um maßgeschneiderte Peptide mit der tRNA zu verschmelzen und eine so genannte Peptidyl-tRNA zu erhalten.
"tRNA-Moleküle zu erhalten, die mit Peptiden verbunden sind, ähnlich denen im Ribosom während der Proteinsynthese, ist seit fast zwei Jahrzehnten ein Traum vieler Forscher auf diesem Gebiet", sagte Polikanov. "Dies war eine große Herausforderung, da es keine Enzyme gibt, die Peptide direkt an eine tRNA binden können.
"Die Methode wird schon seit langem in der Chemie verwendet, aber sie wurde noch nie auf diese Weise angewendet. Im Grunde ahmen wir die Natur nach, und mit unserer fortgeschrittenen Erfahrung in der Bildgebung sehen wir jetzt, wie die Natur in hoher Auflösung funktioniert", so Syroegin.
Mit diesem neuen Ansatz ermittelten Polikanov und Syroegin eine Reihe von hochauflösenden Strukturen des Ribosoms, das Peptidyl-tRNAs unterschiedlicher Länge trägt.
Die detaillierte Analyse dieser Strukturen bietet neue und überraschende Einblicke in den Mechanismus des katalytischen Zentrums des Ribosoms und beantwortet mehrere seit langem bestehende grundlegende Fragen auf dem Gebiet der Ribosomen, so Polikanov.
"Wir haben gesehen, dass verschiedene Peptide je nach Sequenz unterschiedliche Formen oder Falten innerhalb des ribosomalen Tunnels bilden können, und wir können verschiedene Peptide mit unterschiedlichen Sequenzen synthetisieren und dann ihre Form aufgrund der hohen Auflösung unserer Strukturen sehr genau verfolgen", sagte Syroegin. Jetzt können wir mit großer Sicherheit sagen, dass diese Peptide mit dieser Sequenz diese Form haben" oder dass ein anderes Peptid eine andere Form hat". Das ist wichtig, denn die Faltung des Peptids im Anfangsstadium bestimmt, ob Medikamente das Ribosom aufhalten oder nicht."
"Diese Methode eröffnet zahllose Möglichkeiten für strukturelle und funktionelle Studien, die darauf abzielen, die Mechanismen der Ribosomenfunktion sowie sequenzspezifische Ribosomenstillstände, die durch bestimmte Antibiotika ausgelöst werden, zu verstehen", so Polikanov.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.
Originalveröffentlichung
"Insights into the ribosome function from the structures of non-arrested ribosome–nascent chain complexes"; Nature Chemistry.