Komplexe Muster: Eine Brücke vom Großen ins Kleine schlagen
Ein neue Theorie ermöglicht die Simulation komplexer Musterbildung in biologischen Systemen über unterschiedliche räumliche und zeitliche Skalen
Für viele lebenswichtige Prozesse wie Zellteilung, Zellmigration oder die Entwicklung von Organen ist die räumlich und zeitlich korrekte Bildung biologischer Muster essenziell. Um diese Prozesse zu verstehen, gilt es nicht in erster Linie zu erklären, wie sich Muster aus einem homogenen Anfangszustand heraus ausbilden, sondern vielmehr wie sich einfache Muster in immer komplexer werdende Muster verwandeln. Die Mechanismen dieser komplexen Selbstorganisation auf unterschiedlichen räumlichen und zeitlichen Skalen aufzuklären, ist eine zentrale wissenschaftliche Herausforderung. Mit „coarse-graining“ – also „vergröbernden“ – Verfahren können solche multiskaligen Systeme vereinfacht werden, sodass sie mit einem reduzierten Modell auf großen Längen-und Zeitskalen beschrieben werden können. „Der Preis den man durch coarse-graining allerdings bezahlt ist, dass wesentliche Informationen über die Muster auf den kleinen Skalen (wie z.B. der Mustertyp) verloren gehen. Diese Muster spielen aber eine entscheidende Rolle in biologischen Systemen, weil sie beispielsweise wichtige zelluläre Prozesse steuern“, erklärt Laeschkir Würthner, Mitarbeiter im Team des LMU-Physikers Prof. Erwin Frey und Erstautor einer neuen Studie, die dieses Manko überwindet. Freys Team hat in Kooperation mit der Arbeitsgruppe von Prof. Cees Dekker (TU Delft) für sogenannte masseerhaltende Reaktions-Diffusions-Systeme einen neuen coarse-graining Ansatz entwickelt, bei dem die großskalige Analyse der Gesamtdichten der beteiligten Teilchen die Vorhersage von Mustern auf kleinen Skalen ermöglicht.

Prof. Dr. Erwin Frey
LMU / v.zingn
Numerische Berechnungen in Minuten statt Monaten
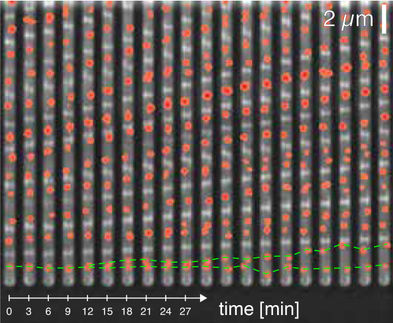
Die Wissenschaftlerinnen und Wissenschaftler veranschaulichten das Potenzial ihres Ansatzes anhand des Min-Protein-Systems, einem wichtigen Modell für die biologische Musterbildung. Das Bakterium E. coli legt mithilfe verschiedener in der Zelle zirkulierender Min-Proteine fest, an welcher Stelle die Zellteilung erfolgt. Entscheidend ist dabei, dass die beteiligten Proteine je nach ihrer Lokalisation in der Zelle und chemischen Zustand unterschiedlich häufig sind, also eine Vielzahl unterschiedlicher Dichten aufweisen. „Uns ist es nun gelungen, die Komplexität dieses Systems zu reduzieren, indem wir eine Theorie entwickelt haben, die lediglich auf den Gesamtdichten der Proteine basiert, um die Dynamik der Musterbildung vollständig widerzuspiegeln“, sagt Frey. „Dies ist eine enorme Reduktion. Die numerischen Berechnungen dauern dadurch nur Minuten anstatt Monate.“
Theoretische Vorhersagen des Modells, nach denen die Verteilung der Proteine von der Geometrie der Umgebung abhängt, konnten die Forschenden auch experimentell bestätigen, indem sie das Min-Proteinsystem in einer In-vitro-Durchflusszelle nachbauten: Dabei zeigten sich dieselben Proteinmuster wie in der Simulation. „Eine solche Rekonstruktion von Informationen auf kleiner Längenskala aus einer reduzierten Dynamik auf makroskopischer Ebene eröffnet neue Wege für ein besseres Verständnis von komplexen Multiskalensystemen, die in einem breiten Spektrum von physikalischen Systemen vorkommen“, sagt Frey.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

Antibody Stabilizer von CANDOR Bioscience
Protein- und Antikörperstabilisierung leicht gemacht
Langzeitlagerung ohne Einfrieren – Einfache Anwendung, zuverlässiger Schutz

DynaPro NanoStar II von Wyatt Technology
NanoStar II: DLS und SLS mit Touch-Bedienung
Größe, Partikelkonzentration und mehr für Proteine, Viren und andere Biomoleküle

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.
Meistgelesene News
Weitere News von unseren anderen Portalen
Zuletzt betrachtete Inhalte
Herbert A. Fritsche, Ph.D., tritt Health Discovery Corporation als Senior Vice President und Chief Science Officer bei
Immunhistochemie
Senologie

Effizientes Recyclingverfahren für Seltene Erden durch Biolaugung und Bioakkumulation - Großes Potenzial bei der Rückgewinnung von Wertstoffen aus Elektronikschrott: Bis zu 85% Rückgewinnung Seltener Erden im Labor erzielt
Nanomaterialien auf der Spur - Forscher ebnen neue Wege zur realistischen Risikobewertung von Nanomaterialien in der Umwelt
Nissl-Färbung