Künstliche Intelligenz sagt die regulatorische Rolle und die 3D-Struktur der DNA voraus
Algorithmen zur Sequenzmodellierung könnten neue Wege zur Bekämpfung von durch genetische Mutationen verursachten Krankheiten eröffnen
Neu entwickelte Programme der künstlichen Intelligenz (KI) haben die Rolle der regulatorischen Elemente und der dreidimensionalen (3D) Struktur der DNA allein auf der Grundlage ihrer Rohsequenz genau vorhergesagt, so zwei aktuelle Studien in Nature Genetics. Diese Werkzeuge könnten schließlich ein neues Licht darauf werfen, wie genetische Mutationen zu Krankheiten führen, und könnten zu einem neuen Verständnis darüber führen, wie die genetische Sequenz die räumliche Organisation und Funktion der chromosomalen DNA im Zellkern beeinflusst, sagte Studienautor Jian Zhou, Ph.D., Assistenzprofessor in der Lyda Hill-Abteilung für Bioinformatik an der UTSW.
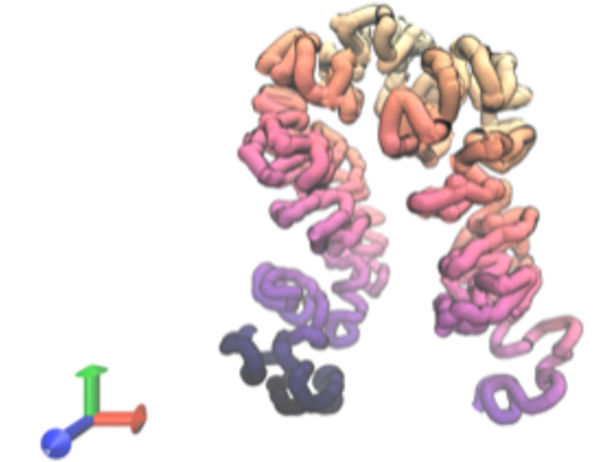
Vorhersage der 3D-Struktur eines Abschnitts der menschlichen genomischen DNA
UT Southwestern Medical Center
"Zusammengenommen liefern diese beiden Programme ein vollständigeres Bild davon, wie Veränderungen in der DNA-Sequenz, selbst in nicht kodierenden Regionen, dramatische Auswirkungen auf ihre räumliche Organisation und Funktion haben können", so Dr. Zhou, Mitglied des Harold C. Simmons Comprehensive Cancer Center, Lupe Murchison Foundation Scholar in Medical Research und Cancer Prevention and Research Institute of Texas (CPRIT) Scholar.
Nur etwa 1 % der menschlichen DNA kodiert Anweisungen für die Herstellung von Proteinen. Die Forschung der letzten Jahrzehnte hat gezeigt, dass ein großer Teil des verbleibenden nicht codierenden genetischen Materials regulatorische Elemente enthält - wie Promotoren, Enhancer, Silencer und Insulatoren -, die steuern, wie die codierende DNA ausgedrückt wird. Wie die Sequenz die Funktionen der meisten dieser regulatorischen Elemente steuert, ist nicht gut verstanden, erklärte Dr. Zhou.
Um diese regulatorischen Komponenten besser zu verstehen, entwickelten er und seine Kollegen von der Princeton University und dem Flatiron Institute ein Deep-Learning-Modell mit dem Namen Sei, das diese Schnipsel nichtcodierender DNA genau in 40 "Sequenzklassen" oder Aufgaben einteilt - zum Beispiel als Verstärker für die Genaktivität von Stammzellen oder Gehirnzellen. Diese 40 Sequenzklassen, die anhand von fast 22 000 Datensätzen aus früheren Studien zur Genomregulierung entwickelt wurden, decken mehr als 97 % des menschlichen Genoms ab. Darüber hinaus kann Sei jede Sequenz nach ihrer vorausgesagten Aktivität in jeder der 40 Sequenzklassen bewerten und vorhersagen, wie sich Mutationen auf diese Aktivitäten auswirken.
Durch die Anwendung von Sei auf humangenetische Daten waren die Forscher in der Lage, die regulatorische Architektur von 47 Merkmalen und Krankheiten zu charakterisieren, die in der UK Biobank-Datenbank erfasst sind, und zu erklären, wie Mutationen in regulatorischen Elementen spezifische Pathologien verursachen. Solche Fähigkeiten können dazu beitragen, ein systematischeres Verständnis dafür zu gewinnen, wie genomische Sequenzveränderungen mit Krankheiten und anderen Merkmalen verbunden sind. Die Ergebnisse wurden diesen Monat veröffentlicht.
Im Mai berichtete Dr. Zhou über die Entwicklung eines anderen Tools namens Orca, das die 3D-Architektur der DNA in Chromosomen auf der Grundlage ihrer Sequenz vorhersagt. Unter Verwendung vorhandener Datensätze von DNA-Sequenzen und Strukturdaten aus früheren Studien, die die Faltungen, Drehungen und Wendungen des Moleküls aufzeigten, trainierte Dr. Zhou das Modell, um Verbindungen herzustellen, und bewertete die Fähigkeit des Modells, die Struktur auf verschiedenen Längenskalen vorherzusagen.
Die Ergebnisse zeigten, dass Orca sowohl kleine als auch große DNA-Strukturen auf der Grundlage ihrer Sequenzen mit hoher Genauigkeit vorhersagen konnte, auch für Sequenzen, die Mutationen tragen, die mit verschiedenen Gesundheitszuständen in Verbindung gebracht werden, darunter eine Form von Leukämie und Missbildungen der Gliedmaßen. Orca ermöglichte es den Forschern auch, neue Hypothesen darüber aufzustellen, wie die DNA-Sequenz ihre lokale und großräumige 3D-Struktur kontrolliert.
Dr. Zhou sagte, dass er und seine Kollegen planen, Sei und Orca, die beide auf Webservern und als Open-Source-Code öffentlich zugänglich sind, zu nutzen, um die Rolle genetischer Mutationen bei der Verursachung der molekularen und physischen Manifestationen von Krankheiten weiter zu erforschen - eine Forschung, die schließlich zu neuen Möglichkeiten der Behandlung dieser Erkrankungen führen könnte.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.