Das „Selenosom“: Choreografie einer Umprogrammierung
Kryo-Elektronenmikroskopie macht zelluläre Strukturen sichtbar
Einem Team unter Leitung von Forschenden der Charité – Universitätsmedizin Berlin ist es gelungen, einen grundlegenden Vorgang der Molekularbiologie aufzuklären: den Einbau der sogenannten 21. Aminosäure Selenocystein in sogenannte Selenoproteine. Diese besonderen Eiweiß-Konstrukte sind für Säugetiere, Menschen, aber auch einige Mikroorganismen lebensnotwendig. Wie sie im Körper entstehen und zusammengefügt werden, war bislang unbekannt. Im Fachmagazin Science beschreiben die Autoren erstmals im Detail, wie eine spezielle Bindungstasche im Zusammenspiel mit verschiedenen weiteren Faktoren diesen Vorgang ermöglicht.
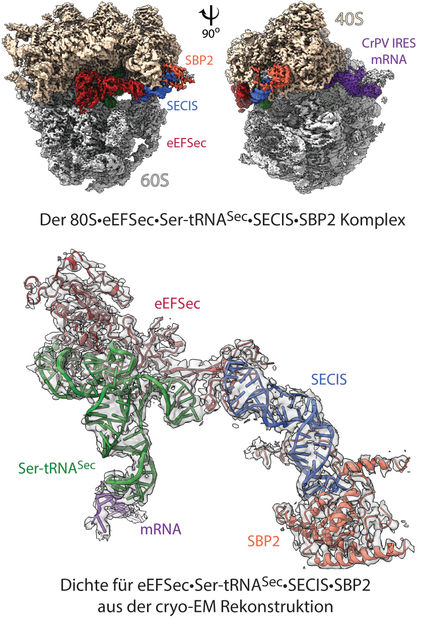
Struktur des „Selenosoms“: dreidimensionale Kryo-EM-Rekonstruktion des ribosomalen Komplexes mit einer Auflösung von 2,8 Ångström (oben). Zu sehen sind die ribosomalen 40S- und 60S-Untereinheiten, gekennzeichnet sind die Bindungselemente (Liganden): mRNA sowie zusätzlich beitragende Faktoren. Im Modell (unten) zeigt die hervorgehobene Dichte für Faktoren eEFSec•Ser-tRNASec•SECIS•SBP2, wie sich die einzelnen Elemente am 80S Ribosom anordnen und die Umprogrammierung zustande kommt.
© Charité | C. M. T. Spahn
Selenoproteine sind eine ungewöhnliche Gruppe von Proteinen, die noch gar nicht so lang bekannt ist. Man geht von bis zu 50 dieser Eiweiße aus, nur ein Teil von ihnen ist bisher erforscht. Sie alle sind komplex aufgebaut und enthalten im Zentrum mindestens einen Teil der namensgebenden Aminosäure Selenocystein (Sec). Selenoproteine übernehmen wichtige Schutz- und Abwehrfunktionen in der Zelle und im menschlichen Körper. Vor allem fungieren sie als sogenannte Oxidoreduktasen, also Vermittler zentraler chemischer Reaktionen, und als Schilddrüsenhormone. Es wird auch vermutet, dass Selenoproteine zum Schutz vor Tumoren beitragen, da sie das Element Selen tragen und oxidativem Stress schnell entgegenwirken können.
Doch wie kommen diese besonderen Eiweiße zustande? Wie funktioniert die molekulare Choreografie beim Einbau von Selenocystein während des Zusammenfügens der Proteine, der Proteinbiosynthese? Und wie genau sieht die Struktur des „Selenosoms“ aus, jenem Komplex, der sich bildet, um Selenoproteine herzustellen? Das Team um Prof. Dr. Christian Spahn, Direktor des Instituts für Medizinische Physik und Biophysik, und Dr. Tarek Hilal, Institut für Chemie und Biochemie der Freien Universität Berlin, konnte gemeinsam mit Partnern am Max-Planck-Institut für Molekulare Genetik, an der University of Illinois, Chicago und der Rutgers-Robert Wood Johnson Medical School, New Jersey, mittels hochauflösender, dreidimensionaler Kryo-Elektronenmikroskopie, diesen fundamentalen molekularbiologischen Vorgang strukturell und mechanistisch nachvollziehen.
Ribosomen, die Proteinfabriken der Zelle, stellen Proteine normalerweise streng nach den von den messenger-RNAs (mRNAs) gelieferten Bauplänen her. Der universelle genetische Code definiert anhand bestimmter Sequenzen, bestehend aus jeweils drei variierenden Basen, den mRNA-Triplett-Codonen, welche Aminosäure an welcher Stelle eines Proteins eingebaut wird. Selenoproteine haben allerdings einen speziellen Aufbau, sodass die 20 Standardaminosäuren bei ihnen nicht ausreichen. Sie enthalten an bestimmten Stellen die sogenannte 21. Aminosäure, das Selenocystein (Sec).
Da für den Einbau von Selenocystein kein eigenes Codon, also keine verschlüsselnde Basensequenz existiert, entsteht beim Bau von Selenoproteinen im ribosomalen Komplex eine grundlegende Besonderheit. Durch eine gesonderte Signalsequenz in der mRNA, das sogenannte SECIS-Element (SElenoCystein-Insertionssequenz), wird das Ribosom umprogrammiert und die genetische Information quasi überschrieben. Ein Codon, das normalerweise einen Kettenabbruch und damit die Fertigstellung des Proteins programmiert (UGA-Stopcodon), wird an den gewünschten Stellen zu einem neuen Codon, und zwar dem Codon zum Einbau von Selenocystein. Dieser Rekodierungsvorgang erfordert neben dem SECIS-Element zusätzlich eine besondere, mit Selenocystein beladene Transfer-RNA (tRNASec) und zusätzliche, spezialisierte Translationsfaktoren. „Zwar sind die beteiligten Player seit einigen Jahren bekannt, dennoch blieb bislang ein Rätsel, wie sie genau funktionieren und wie sie zusammenwirken“, so Prof. Spahn. „Vor allem, wie im Einzelnen das SECIS-Element wirkt, war mysteriös, denn es befindet sich in der linearen Sequenz der mRNA nicht in direkter Nachbarschaft zum umprogrammierten UGA-Stopcodon, sondern am Ende, viele hundert Nukleotid-Bausteine entfernt.“
Um den molekularen Mechanismus aufzuklären, hat das Forschungsteam den ribosomalen Komplex, der sich bildet, um das UGA-Stopcodon zu rekodieren, das „Selenosom“, im Labor nachempfunden. Das hochauflösende Bildgebungsverfahren der Kryo-Elektronenmikroskopie ermöglicht eine dreidimensionale Darstellung des winzigen Konstrukts und somit strukturelle Untersuchungen. „Anhand der abgebildeten Strukturen konnten wir aufklären, wie die beteiligten Faktoren mit dem Ribosom interagieren und wie sie genau zusammenwirken, um das Ribosom umzuprogrammieren“, erklärt Prof. Spahn. „Wir konnten insbesondere zeigen, dass die mRNA eine große Schleife bildet, so dass das UGA-Stopcodon und das SECIS-Element gleichzeitig am Ribosom gebunden sind. Das SECIS-Element wird dabei in einer bislang unbekannten Bindetasche am Ribosom verankert und kann dann, im Ribosomen-gebundenen Zustand, unterstützt durch Translationsfaktoren den Selenocystein-Einbau begünstigen.“ Diese Struktur und die Funktionsweise des „Selenosoms“ haben das Forschungsteam überrascht und waren so nicht vorherzusehen. Denn der Vorgang verläuft bei Säugetieren und beim Menschen deutlich anders als der Einbau von Selenocystein in Bakterien, der zuvor schon bekannt war. Der nun beschriebene Komplex zeigt beispielhaft, wie Signalstrukturen am hinteren Ende einer mRNA, also außerhalb des kodierenden Bereiches, mit dem Ribosom interagieren können, um dieses zu regulieren.
Den ersten Schritt beim Einbau von Selenocystein konnten die Forschenden somit aufklären. Die nachfolgenden Schritte sind noch immer unklar und sollen in weiteren Arbeiten strukturell untersucht werden. Studien wie diese tragen dazu bei, Funktion und Bedeutung des lebenswichtigen Spurenelements Selen in der normalen Physiologie und beim Entstehen von Krankheiten wie Diabetes oder Krebs besser zu verstehen.





















































