Wie eine Bodenmikrobe die künstliche Photosynthese ankurbeln könnte
Das Geheimnis ist ein Enzym, das mit den Reaktionsbestandteilen "jongliert"
Pflanzen sind für ihre Existenz auf einen Prozess angewiesen, der als Kohlenstofffixierung bezeichnet wird - die Umwandlung von Kohlendioxid aus der Luft in kohlenstoffreiche Biomoleküle. Das ist der Sinn der Photosynthese und ein Eckpfeiler des riesigen, ineinandergreifenden Systems, das den Kohlenstoffkreislauf durch Pflanzen, Tiere, Mikroben und die Atmosphäre aufrechterhält, um das Leben auf der Erde zu erhalten.
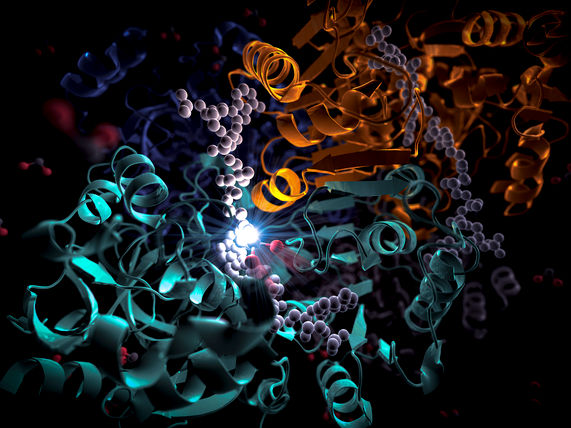
Ein Reaktionssubstrat (lila Molekül in der Mitte) schmiegt sich in eine Tasche des Enzyms ECR (orange und türkis), das eine Reaktion katalysiert, bei der Kohlendioxid (rosa Kugeln) in Biomoleküle umgewandelt wird, die von Bodenbakterien benötigt werden. Eine neue Studie zeigt, wie ECR diese Reaktion 20 Mal schneller durchführt als das entsprechende Enzym in der Photosynthese. Die Wissenschaftler hoffen, diese Enzymfamilie zu optimieren, um Produkte aus CO2 herzustellen.
Greg Stewart, SLAC National Accelerator Laboratory
Aber die Meister der Kohlenstoffbindung sind nicht die Pflanzen, sondern die Bodenbakterien. Einige bakterielle Enzyme führen einen Schlüsselschritt bei der Kohlenstofffixierung 20-mal schneller aus als Pflanzenenzyme. Wenn man herausfindet, wie sie dies tun, könnte dies Wissenschaftlern helfen, Formen der künstlichen Photosynthese zu entwickeln, um das Treibhausgas in Kraftstoffe, Düngemittel, Antibiotika und andere Produkte umzuwandeln.
Jetzt hat ein Team von Forschern des SLAC National Accelerator Laboratory des Energieministeriums, der Stanford University, des Max-Planck-Instituts für terrestrische Mikrobiologie in Deutschland, des Joint Genome Institute (JGI) des DOE und der Universität Concepción in Chile herausgefunden, wie ein bakterielles Enzym - eine molekulare Maschine, die chemische Reaktionen ermöglicht - auf Touren kommt, um dieses Kunststück zu vollbringen.
Anstatt Kohlendioxidmoleküle zu ergreifen und sie einzeln an Biomoleküle zu binden, besteht dieses Enzym aus Molekülpaaren, die synchron arbeiten, wie die Hände eines Jongleurs, der gleichzeitig Bälle wirft und fängt, um die Arbeit schneller zu erledigen. Ein Mitglied jedes Enzympaars öffnet sich weit, um eine Reihe von Reaktionsbestandteilen zu fangen, während sich das andere über den gefangenen Bestandteilen schließt und die kohlenstoffbindende Reaktion durchführt; dann tauschen sie in einem kontinuierlichen Zyklus die Rollen.
Das Team entdeckte, dass ein einziger molekularer "Klebstoff" jedes Enzympaar zusammenhält, so dass sie sich abwechselnd auf koordinierte Weise öffnen und schließen können, während eine Drehbewegung dazu beiträgt, Zutaten und Endprodukte in und aus den Taschen zu befördern, in denen die Reaktionen stattfinden. Wenn sowohl Klebstoff als auch Drehung vorhanden sind, läuft die kohlenstoffbindende Reaktion 100-mal schneller ab als ohne sie.
"Dieses bakterielle Enzym ist der effizienteste Kohlenstofffixierer, den wir kennen, und wir haben eine saubere Erklärung dafür gefunden, was es tun kann", sagte Soichi Wakatsuki, Professor am SLAC und in Stanford und einer der Hauptverantwortlichen für die Studie, die diese Woche in ACS Central Science veröffentlicht wurde.
"Einige der Enzyme in dieser Familie arbeiten langsam, aber auf eine sehr spezifische Weise, um nur ein einziges Produkt herzustellen", sagte er. "Andere sind viel schneller und können chemische Bausteine für alle Arten von Produkten herstellen. Jetzt, da wir den Mechanismus kennen, können wir Enzyme entwickeln, die die besten Eigenschaften beider Ansätze kombinieren und mit allen möglichen Ausgangsstoffen sehr schnell arbeiten.
Die Natur verbessern
Das von dem Team untersuchte Enzym gehört zu einer Familie, die Enoyl-CoA-Carboxylasen/Reduktasen oder ECRs genannt wird. Es stammt von Bodenbakterien namens Kitasatospora setae, die neben ihrer Fähigkeit, Kohlenstoff zu binden, auch Antibiotika produzieren können.
Wakatsuki erfuhr von dieser Enzymfamilie vor einem halben Dutzend Jahren durch Tobias Erb vom Max-Planck-Institut für terrestrische Mikrobiologie in Deutschland und Yasuo Yoshikuni vom JGI. Erbs Forschungsteam hatte an der Entwicklung von Bioreaktoren für die künstliche Photosynthese gearbeitet, um Kohlendioxid (CO2) aus der Atmosphäre in alle möglichen Produkte umzuwandeln.
So wichtig die Photosynthese für das Leben auf der Erde auch ist, sagte Erb, sie ist nicht sehr effizient. Wie alle Dinge, die im Laufe der Äonen durch die Evolution geformt wurden, ist sie nur so gut, wie sie sein muss. Sie ist das Ergebnis einer langsamen Entwicklung, die auf früheren Entwicklungen aufbaut, aber nie etwas völlig Neues von Grund auf erfindet.
Außerdem sei der Schritt in der natürlichen Photosynthese, bei demCO2 aus der Luft gebunden wird und der von einem Enzym namens Rubisco abhängt, ein Engpass, der die gesamte Kette der photosynthetischen Reaktionen verzögert. Die Verwendung von schnellen ECR-Enzymen für diesen Schritt und die Entwicklung von Enzymen, die diesen Schritt noch schneller ausführen, könnte einen großen Effizienzschub bringen.
"Wir versuchen nicht, eine Kopie der Photosynthese zu erstellen", erklärte Erb. "Wir wollen einen Prozess entwickeln, der viel effizienter ist, indem wir unser technisches Verständnis nutzen, um die Konzepte der Natur nachzubilden. Diese 'Photosynthese 2.0' könnte in lebenden oder synthetischen Systemen wie künstlichen Chloroplasten - in Öl suspendierte Wassertröpfchen - ablaufen."
Porträts eines Enzyms
Wakatsuki und seine Gruppe hatten ein verwandtes System untersucht, die Stickstofffixierung, bei der Stickstoff aus der Atmosphäre in Verbindungen umgewandelt wird, die Lebewesen benötigen. Er war fasziniert von der Frage, warum die ECR-Enzyme so schnell sind, und begann, mit der Gruppe von Erb zusammenzuarbeiten, um Antworten zu finden.
Hasan DeMirci, ein wissenschaftlicher Mitarbeiter in Wakatsukis Gruppe, der jetzt Assistenzprofessor an der Koc-Universität und Forscher am Stanford PULSE Institute ist, leitete die Bemühungen am SLAC mit Hilfe eines halben Dutzend SLAC-Sommerpraktikanten, die er betreute. "Wir bilden jedes Jahr sechs oder sieben von ihnen aus, und sie waren furchtlos", sagte er. "Sie kamen mit offenem Geist, bereit zu lernen, und sie haben Erstaunliches geleistet".
Das SLAC-Team stellte Proben des ECR-Enzyms her und kristallisierte sie zur Untersuchung mit Röntgenstrahlen an der Advanced Photon Source im Argonne National Laboratory des DOE. Die Röntgenstrahlen enthüllten die molekulare Struktur des Enzyms - die Anordnung seines atomaren Gerüsts - sowohl für sich allein als auch in Verbindung mit einem kleinen Hilfsmolekül, das seine Arbeit erleichtert.
Weitere Röntgenstudien an der Stanford Synchrotron Radiation Lightsource (SSRL) des SLAC zeigten, wie sich die Struktur des Enzyms veränderte, wenn es an ein Substrat gebunden war, eine Art molekulare Werkbank, die die Zutaten für die Kohlenstoffbindungsreaktion zusammenstellt und die Reaktion antreibt.
Schließlich führte ein Forscherteam der Linac Coherent Light Source (LCLS) des SLAC genauere Untersuchungen des Enzyms und seines Substrats am japanischen Freie-Elektronen-Röntgenlaser SACLA durch. Die Wahl eines Röntgenlasers war wichtig, weil er es ermöglichte, das Verhalten des Enzyms bei Raumtemperatur - näher an seiner natürlichen Umgebung - fast ohne Strahlenschäden zu untersuchen.
In der Zwischenzeit führten die Gruppe von Erb in Deutschland und die Gruppe von Professor Esteban Vöhringer-Martinez an der Universität von Concepción in Chile detaillierte biochemische Studien und umfangreiche dynamische Simulationen durch, um die von Wakatsuki und seinem Team gesammelten Strukturdaten zu verstehen.
Die Simulationen ergaben, dass das Öffnen und Schließen der beiden Teile des Enzyms nicht nur mit molekularem Klebstoff verbunden ist, sondern auch mit Drehbewegungen um die zentrale Achse jedes Enzympaars, so Wakatsuki.
"Diese Drehung ist fast wie eine Rochade, die ein fertiges Produkt herausdrücken oder einen neuen Satz von Zutaten in die Tasche ziehen kann, in der die Reaktion stattfindet", sagte er. Durch die Verdrehung und Synchronisation der Enzympaare können sie zusammen 100 Mal pro Sekunde Kohlenstoff binden.
Die ECR-Enzymfamilie umfasst auch einen vielseitigeren Zweig, der mit vielen verschiedenen Arten von Biomolekülen interagieren kann, um eine Vielzahl von Produkten herzustellen. Da sie jedoch nicht durch einen molekularen Klebstoff zusammengehalten werden, können sie ihre Bewegungen nicht koordinieren und arbeiten daher viel langsamer.
"Wenn wir die Geschwindigkeit dieser hochentwickelten Reaktionen zur Herstellung neuer Biomoleküle erhöhen können", so Wakatsuki, "wäre das ein bedeutender Sprung in diesem Bereich.
Von statischen Aufnahmen zu Flüssigkeitsfilmen
Bisher haben die Experimente statische Schnappschüsse des Enzyms, der Reaktionsbestandteile und der Endprodukte in verschiedenen Konfigurationen ergeben.
"Unser Traumexperiment", so Wakatsuki, "wäre es, alle Zutaten zu kombinieren, während sie in den Strahlengang des Röntgenlasers fließen, damit wir die Reaktion in Echtzeit beobachten können."
Das Team hat das bei SACLA tatsächlich versucht, aber es hat nicht funktioniert. "DieCO2-Moleküle sind wirklich klein und bewegen sich so schnell, dass es schwierig ist, den Moment zu erwischen, in dem sie sich an das Substrat anlagern", sagte er. "Außerdem ist der Röntgenlaserstrahl so stark, dass wir die Bestandteile nicht lange genug in ihm halten konnten, damit die Reaktion stattfinden konnte. Wenn wir stark darauf drückten, gelang es uns, die Kristalle zu zerbrechen."
Ein bevorstehendes Hochenergie-Upgrade für LCLS wird dieses Problem wahrscheinlich lösen, fügte er hinzu, mit Pulsen, die viel häufiger eintreffen - eine Million Mal pro Sekunde - und individuell auf die ideale Stärke für jede Probe eingestellt werden können.
Wakatsuki sagte, dass sein Team weiterhin mit Erb's Gruppe zusammenarbeitet, und dass es mit der LCLS-Probentransportgruppe und mit Forschern an den SLAC-Stanford Einrichtungen für kryogene Elektronenmikroskopie (Kryo-EM) zusammenarbeitet, um einen Weg zu finden, diesen Ansatz zu verwirklichen.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.