Nanotechnologie ermöglicht Visualisierung von RNA-Strukturen mit nahezu atomarer Auflösung
Kombination von Nukleinsäure-Nanotechnologie und Kryo-EM ermöglicht beispiellose Einblicke in die Strukturen großer und kleiner RNAs
Wir leben in einer Welt, die von der RNA, dem ebenso wichtigen Geschwisterchen des Genmoleküls DNA, geschaffen und gesteuert wird. Evolutionsbiologen gehen sogar davon aus, dass die RNA bereits vor dem Auftreten der DNA und der von ihr kodierten Proteine existierte und sich selbst reproduzierte. Die Wissenschaft hat herausgefunden, dass weniger als 3% des menschlichen Genoms in Boten-RNA-Moleküle (mRNA) umgeschrieben werden, die wiederum in Proteine übersetzt werden. Im Gegensatz dazu werden 82% des Genoms in RNA-Moleküle mit anderen Funktionen umgeschrieben, von denen viele noch immer rätselhaft sind.
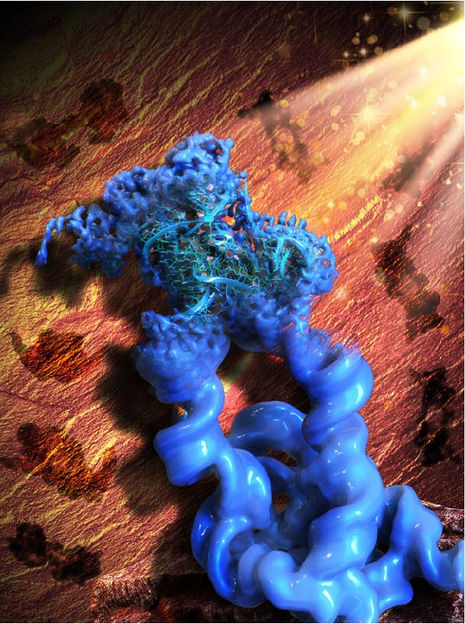
Diese Illustration ist von der paläolithischen Felsmalerei in der Höhle von Lascaux inspiriert und steht für das Akronym unserer Methode, ROCK. Bildlich gesprochen sind die Muster der Felsmalerei im Hintergrund (braun) die 2D-Projektionen des konstruierten dimeren Konstrukts des Tetrahymena-Introns der Gruppe I, während das Hauptobjekt im Vordergrund (blau) die rekonstruierte 3D-Kryo-EM-Karte des Dimers ist, mit einem Monomer im Fokus und verfeinert auf die hohe Auflösung, die es den Mitarbeitern ermöglichte, ein atomares Modell der RNA zu erstellen.
Wyss Institute at Harvard University
Um zu verstehen, was ein einzelnes RNA-Molekül tut, muss seine 3D-Struktur auf der Ebene der einzelnen Atome und molekularen Bindungen entschlüsselt werden, aus denen es besteht. Forscher haben DNA- und Proteinmoleküle routinemäßig untersucht, indem sie sie in regelmäßig gepackte Kristalle verwandelt haben, die mit einem Röntgenstrahl (Röntgenkristallografie) oder Radiowellen (Kernspinresonanz) untersucht werden können. Diese Techniken können jedoch bei RNA-Molekülen nicht annähernd mit der gleichen Effektivität angewandt werden, da sie aufgrund ihrer molekularen Zusammensetzung und strukturellen Flexibilität nicht so leicht Kristalle bilden können.
Nun hat eine Forschungskooperation unter der Leitung von Peng Yin, Ph.D., Mitglied der Wyss Core Faculty am Wyss Institute for Biologically Inspired Engineering an der Harvard University, und Maofu Liao, Ph.D., an der Harvard Medical School (HMS), einen grundlegend neuen Ansatz zur Strukturuntersuchung von RNA-Molekülen vorgestellt. ROCK, wie es genannt wird, verwendet eine RNA-Nanotechnik, die es ermöglicht, mehrere identische RNA-Moleküle zu einer hoch organisierten Struktur zusammenzusetzen, was die Flexibilität der einzelnen RNA-Moleküle deutlich verringert und ihr Molekulargewicht vervielfacht. Anhand bekannter Modell-RNAs mit unterschiedlichen Größen und Funktionen zeigte das Team, dass ihre Methode die strukturelle Analyse der enthaltenen RNA-Untereinheiten mit einer Technik ermöglicht, die als Kryo-Elektronenmikroskopie (Kryo-EM) bekannt ist. Über diesen Fortschritt wird in Nature Methods berichtet.
"ROCK sprengt die derzeitigen Grenzen der RNA-Strukturforschung und ermöglicht es, 3D-Strukturen von RNA-Molekülen zu entschlüsseln, die mit bestehenden Methoden nur schwer oder gar nicht zugänglich sind, und das bei nahezu atomarer Auflösung", so Yin, der zusammen mit Liao die Studie leitete. "Wir erwarten, dass dieser Fortschritt viele Bereiche der Grundlagenforschung und der Arzneimittelentwicklung beleben wird, einschließlich des aufkeimenden Bereichs der RNA-Therapeutika". Yin ist auch Leiter der Molekularen Robotik-Initiative des Wyss-Instituts und Professor in der Abteilung für Systembiologie an der HMS.
Die Kontrolle über RNA gewinnen
Yins Team am Wyss-Institut hat verschiedene Ansätze entwickelt, die es DNA- und RNA-Molekülen ermöglichen, sich auf der Grundlage unterschiedlicher Prinzipien und Anforderungen selbst zu großen Strukturen zusammenzusetzen, darunter DNA-Bausteine und DNA-Origami. Sie stellten die Hypothese auf, dass solche Strategien auch dazu verwendet werden könnten, natürlich vorkommende RNA-Moleküle zu hochgradig geordneten zirkulären Komplexen zusammenzusetzen, in denen ihre Freiheit, sich zu biegen und zu bewegen, stark eingeschränkt ist, indem sie gezielt miteinander verbunden werden. Viele RNAs falten sich auf komplexe und doch vorhersehbare Weise, wobei kleine Segmente durch Basenpaare miteinander verbunden werden. Das Ergebnis ist oft ein stabiler "Kern" und "Stamm-Schleifen", die sich in die Peripherie ausdehnen.
"In unserem Ansatz bauen wir 'Kissing Loops' ein, die verschiedene periphere Stammschleifen, die zu zwei Kopien einer identischen RNA gehören, auf eine Art und Weise verbinden, die es ermöglicht, einen insgesamt stabilisierten Ring zu bilden, der mehrere Kopien der interessierenden RNA enthält", so Di Liu, Ph.D., einer der beiden Erstautoren und Postdoctoral Fellow in Yins Gruppe. "Wir spekulierten, dass diese Ringe höherer Ordnung mit hoher Auflösung durch Kryo-EM analysiert werden könnten, das bereits mit erstem Erfolg auf RNA-Moleküle angewendet wurde."
Stabilisierte RNA abbilden
Bei der Kryo-EM werden viele einzelne Partikel bei kryogenen Temperaturen schockgefroren, um weitere Bewegungen zu verhindern, und dann mit einem Elektronenmikroskop und der Hilfe von Rechenalgorithmen visualisiert, die die verschiedenen Aspekte der 2D-Oberflächenprojektionen eines Partikels vergleichen und seine 3D-Architektur rekonstruieren. Peng und Liu arbeiteten mit Liao und seinem ehemaligen Doktoranden François Thélot, dem anderen Erstautor der Studie, zusammen. Liao hat mit seiner Gruppe wichtige Beiträge zu dem sich rasch entwickelnden Bereich der Kryo-EM und der experimentellen und rechnerischen Analyse einzelner Partikel geleistet, die von bestimmten Proteinen gebildet werden.
"Kryo-EM hat große Vorteile gegenüber herkömmlichen Methoden, um hochauflösende Details biologischer Moleküle wie Proteine, DNAs und RNAs zu sehen, aber die geringe Größe und die Bewegungstendenz der meisten RNAs verhindern eine erfolgreiche Bestimmung von RNA-Strukturen. Unsere neuartige Methode zum Zusammensetzen von RNA-Multimeren löst diese beiden Probleme gleichzeitig, indem sie die Größe der RNA erhöht und ihre Bewegung verringert", sagte Liao, der auch außerordentlicher Professor für Zellbiologie an der HMS ist. "Unser Ansatz hat die Tür zu einer schnellen Strukturbestimmung vieler RNAs durch Kryo-EM geöffnet." Die Integration von RNA-Nanotechnologie und Kryo-EM-Ansätzen veranlasste das Team, seine Methode "RNA oligomerization-enabled cryo-EM via installing kissing loops" (ROCK) zu nennen.
Um den Grundsatzbeweis für ROCK zu erbringen, konzentrierte sich das Team auf eine große Intron-RNA aus Tetrahymena, einem einzelligen Organismus, und eine kleine Intron-RNA aus Azoarcus, einem stickstofffixierenden Bakterium, sowie auf den so genannten FMN-Riboswitch. Intron-RNAs sind nicht-kodierende RNA-Sequenzen, die in die Sequenzen frisch transkribierter RNAs eingestreut sind und herausgespleißt" werden müssen, damit die reife RNA entstehen kann. Der FMN-Riboschalter findet sich in bakteriellen RNAs, die an der Biosynthese von Flavin-Metaboliten aus Vitamin B2 beteiligt sind. Bei der Bindung eines dieser Metaboliten, des Flavinmononukleotids (FMN), schaltet es seine 3D-Konformation um und unterdrückt die Synthese der Mutter-RNA.
"Der Zusammenbau des Tetrahymena-Introns der Gruppe I zu einer ringförmigen Struktur machte die Proben homogener und ermöglichte den Einsatz von Berechnungswerkzeugen, die die Symmetrie der zusammengesetzten Struktur nutzen. Unser Datensatz ist zwar relativ bescheiden, aber die Vorteile von ROCK haben es uns ermöglicht, die Struktur in einer noch nie dagewesenen Auflösung aufzulösen", so Thélot. "Der Kern der RNA ist mit 2,85 Å aufgelöst [ein Ångström ist ein Zehnmilliardenmeter und die von Strukturbiologen bevorzugte Maßeinheit], wodurch detaillierte Merkmale der Nukleotidbasen und des Zuckergerüsts sichtbar werden. Ich glaube nicht, dass wir ohne ROCK so weit gekommen wären - oder zumindest nicht ohne erheblich mehr Ressourcen."
Die Kryo-EM ist auch in der Lage, Moleküle in verschiedenen Zuständen zu erfassen, wenn sie zum Beispiel ihre 3D-Konformation als Teil ihrer Funktion ändern. Durch die Anwendung von ROCK auf die Azoarcus-Intron-RNA und den FMN-Riboschalter gelang es dem Team, die verschiedenen Konformationen zu identifizieren, die das Azoarcus-Intron während des Selbstspleißprozesses durchläuft, und die relative Konformationssteifigkeit der Ligandenbindungsstelle des FMN-Riboschalters aufzudecken.
"Diese Studie von Peng Yin und seinen Mitarbeitern zeigt auf elegante Weise, wie die RNA-Nanotechnologie als Beschleuniger für den Fortschritt anderer Disziplinen dienen kann. Die Möglichkeit, die Strukturen vieler natürlich vorkommender RNA-Moleküle zu visualisieren und zu verstehen, könnte enorme Auswirkungen auf unser Verständnis vieler biologischer und pathologischer Prozesse in verschiedenen Zelltypen, Geweben und Organismen haben und sogar neue Ansätze für die Entwicklung von Medikamenten ermöglichen", sagte Wyss-Gründungsdirektor Donald Ingber, M.D., Ph.D., der auch Judah-Folkman-Professor für Gefäßbiologie an der Harvard Medical School und am Boston Children's Hospital sowie Professor für Bioengineering an der Harvard John A. Paulson School of Engineering and Applied Sciences ist.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.
























































