Deep Learning erträumt neue Proteinstrukturen
Forscher zeigen, dass ein neuronales Netz, das ausschließlich für die Vorhersage von Proteinformen trainiert wurde, auch neue Formen erzeugen kann
So wie mit Hilfe künstlicher Intelligenz überzeugende Bilder von Katzen erstellt werden können, lassen sich jetzt mit ähnlichen Werkzeugen neue Proteine herstellen. In einem Bericht in Nature beschreibt ein Team, dem Forscher der University of Washington, des Rensselaer Polytechnic Institute und der Harvard University angehören, die Entwicklung eines neuronalen Netzwerks, das Proteine mit neuen, stabilen Strukturen "halluziniert".
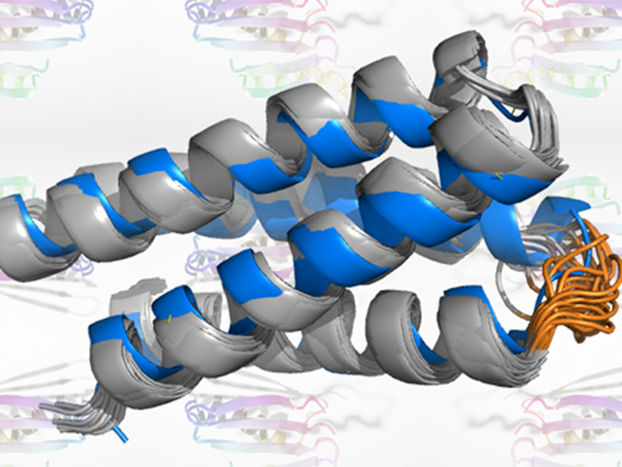
Ein neuronales Netz "halluzinierte" Proteine, die synthetisiert wurden, um ihre Struktur zu bestätigen.
Rensselaer Polytechnic Institute
"Das Potenzial, brandneue Proteine zu halluzinieren, die bestimmte Biomoleküle binden oder die gewünschten aktiven Stellen von Enzymen bilden, ist sehr aufregend", sagte Gaetano Montelione, Professor für Chemie und chemische Biologie an der Rensselaer University, wo synthetisierte Versionen von "halluzinierten" Proteinen, die von einem neuronalen Netzwerk erfunden wurden, analysiert wurden.
Proteine sind fadenförmige Moleküle, die in jeder Zelle vorkommen und sich spontan zu komplizierten dreidimensionalen Formen falten. Diese gefalteten Formen sind der Schlüssel zu fast allen Prozessen in der Biologie, einschließlich Zellentwicklung, DNA-Reparatur und Stoffwechsel. Die Komplexität der Proteinformen macht es jedoch schwierig, sie zu untersuchen. Biochemiker verwenden häufig Computer, um vorherzusagen, wie sich Proteinstränge oder -sequenzen falten könnten. In den letzten Jahren haben Techniken der künstlichen Intelligenz, wie neuronale Netze und Deep Learning, die Genauigkeit dieser Arbeit revolutioniert.
"Für dieses Projekt haben wir völlig zufällige Proteinsequenzen erstellt und so lange Mutationen in sie eingebracht, bis unser neuronales Netzwerk vorhersagte, dass sie sich zu stabilen Strukturen falten würden", sagt Co-Autor Ivan Anishchenko, ein Postdoktorand im Baker-Labor am Institut für Proteindesign der University of Washington School of Medicine. "Wir haben die Software zu keinem Zeitpunkt auf ein bestimmtes Ergebnis ausgerichtet - diese neuen Proteine sind einfach das, was sich der Computer ausdenkt."
In Zukunft sollte es nach Ansicht des Teams möglich sein, die künstliche Intelligenz so zu steuern, dass sie neue Proteine mit nützlichen Eigenschaften erzeugt. "Wir möchten Deep Learning nutzen, um Proteine mit Funktionen zu entwerfen, einschließlich proteinbasierter Medikamente, Enzyme und so weiter", so Co-Autor Sam Pellock, ein Postdoktorand im Baker-Labor.
Das Forschungsteam generierte 2.000 neue Proteinsequenzen, deren Faltung vorhergesagt wurde. Über 100 von ihnen wurden im Labor hergestellt und untersucht. Detaillierte Analysen von drei dieser Proteine bestätigten, dass die vom Computer vorhergesagten Formen im Labor tatsächlich realisiert wurden.
"Unsere Lösungs-NMR-Studien zeigen zusammen mit den Röntgenkristallstrukturen, die vom Team der University of Washington bestimmt wurden, die bemerkenswerte Genauigkeit der Proteindesigns, die mit dem Halluzinationsansatz erstellt wurden", sagte Mitautorin Theresa Ramelot, eine leitende Wissenschaftlerin im Montelione-Labor innerhalb des Rensselaer Center for Biotechnology and Interdisciplinary Studies.
Montelione merkt an: "Der Halluzinationsansatz baut auf früheren Beobachtungen auf, die wir zusammen mit dem Baker-Labor gemacht haben und die zeigen, dass die Proteinstrukturvorhersage mit Deep Learning sogar für eine einzelne Proteinsequenz recht genau sein kann, ohne auf Kontaktvorhersagen zurückzugreifen, die normalerweise durch die Analyse vieler evolutionär verwandter Proteinsequenzen erhalten werden."
"Dieser Ansatz vereinfacht das Design von Proteinen erheblich", sagte der Erstautor David Baker, der 2021 mit dem Breakthrough Prize in Life Sciences ausgezeichnet wurde. "Früher musste man, um ein neues Protein mit einer bestimmten Form zu entwerfen, zunächst sorgfältig verwandte Strukturen in der Natur studieren, um eine Reihe von Regeln aufzustellen, die dann im Designprozess angewendet wurden. Für jede neue Art von Faltung wurden neue Regeln benötigt. Durch den Einsatz eines Deep-Learning-Netzwerks, das bereits allgemeine Prinzipien der Proteinstruktur erfasst, brauchen wir keine faltungsspezifischen Regeln mehr und können uns direkt auf die funktionalen Teile eines Proteins konzentrieren."
"Die Erforschung der Frage, wie diese Strategie am besten für spezifische Anwendungen genutzt werden kann, ist jetzt ein aktiver Forschungsbereich, und hier erwarte ich die nächsten Durchbrüche", so Baker.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.