Mit künstlicher Intelligenz zu neuen Naturstoffen
Neues Verfahren kann bislang unbekannte kleine Moleküle schnell und sicher identifizieren
Sie sind entzündungshemmend, können Krankheitskeime abwehren oder unterbinden sogar das Wachstum von Krebszellen – Wirkstoffe aus der Natur. Mehr als ein Drittel aller heute verfügbaren Medikamente basieren auf solchen sekundären Naturstoffen, wie sie in zahlreichen Pflanzen, Bakterien und Pilzen vorkommen. Den reichhaltigen Medizinschrank der Natur nutzbar zu machen und neue Naturstoffe zu identifizieren, ist jedoch zeit-, kosten- und arbeitsintensiv. Ein Team der Bioinformatik an der Friedrich-Schiller-Universität Jena hat jetzt ein Verfahren entwickelt, mit dem sich kleine Wirkstoff-Moleküle sehr viel schneller und einfacher identifizieren lassen als bisher. Ihr Verfahren COSMIC (Confidence Of Small Molecule IdentifiCations) stellen die Forschenden in der aktuellen Ausgabe der Fachzeitschrift Nature Biotechnology vor.
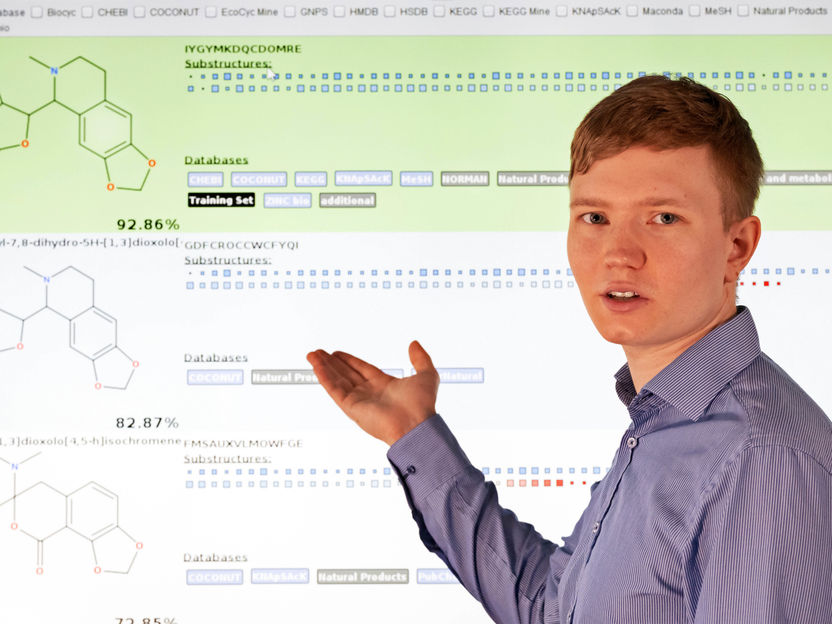
Martin Hoffmann von der Universität Jena präsentiert COSMIC (Confidence Of Small Molecule IdentifiCations), ein Verfahren mit dem sich kleine Wirkstoff-Moleküle sehr viel schneller und einfacher identifizieren lassen als bisher.
Jens Meyer/Uni Jena
Millionen von Strukturdaten bisher nicht entschlüsselt
Will man wissen, welche Substanzen eine biologische Probe wie etwa ein Pflanzenextrakt enthält, wird diese mittels Massenspektrometrie analysiert. Dabei werden die Moleküle in Fragmente zerlegt und deren Masse bestimmt. „Die von uns entwickelte Molekülsuchmaschine CSI:FingerID erlaubt es gezielt nach Molekülstrukturen zu suchen, die zu diesen Fragmenten passen“, beschreibt Prof. Dr. Sebastian Böcker von der Universität Jena das Vorgehen. „Ob diese Suche erfolgreich ist – das Suchergebnis also die korrekte Struktur wiedergibt – können wir so nicht entscheiden“.
Mittlerweile liegen riesige Datensammlungen mit Milliarden von Massenspektrometriedaten aus Millionen von Analysen biologischer Proben vor, wovon der weitaus größte Teil in ihrer Struktur nicht identifiziert sind. Genau hier kommt nun COSMIC zum Einsatz und ermöglicht es, für einen großen Teil dieser bisher unidentifizierten Moleküle automatisch Strukturen zu entschlüsseln. „Dabei nutzen wir Verfahren des maschinellen Lernens“, erläutert Martin Hoffmann, der Erstautor der vorgelegten Publikation. „Zunächst wird das Massenspektrum der untersuchten Probe mit den verfügbaren Strukturdaten abgeglichen.“ Als Ergebnis erhält man – wie in einer Google-Suche – eine mehr oder weniger umfangreiche Liste möglicher Treffer. „Unsere Methode gibt nun an, wie sicher es ist, dass der gefundene Treffer an erster Stelle tatsächlich die Struktur ist, nach der wir suchen“, veranschaulicht Hoffmann. Dafür ermittelt COSMIC einen Score, der die Qualität des vorgeschlagenen Treffers bewertet und daraus ableitet, ob er richtig oder falsch ist.
Neue Gallensäuren entdeckt
Wie gut ihre Methode tatsächlich funktioniert, konnten Böcker und sein Team in Kooperation mit Kolleginnen und Kollegen der University of California in San Diego demonstrieren. Sie haben dafür Massenspektrometriedaten des Verdauungssystems von Mäusen untersucht und darin nach bislang unbekannten Gallensäuren gesucht. Dafür wurden über 28.000 theoretisch mögliche Gallensäurestrukturen konstruiert und diese mit den Messdaten aus dem Mikrobiom der Mäuse abgeglichen. Die anschließende Analyse mit COSMIC lieferte insgesamt elf neue, bislang völlig unbekannte Gallensäurestrukturen. Zwei davon konnten inzwischen anhand gezielt synthetisierter Referenzproben bestätigt werden.
„Das zeigt zum einen, dass unsere Methode zuverlässig funktioniert“, unterstreicht Sebastian Böcker. Zum anderen biete COSMIC die Chance, die Suche nach neuen interessanten Substanzen erheblich zu beschleunigen, da sich das Screening vollautomatisch, ohne personellen Aufwand und in kurzer Zeit durchführen lasse. Er erwartet, dass in den kommenden Jahren auf diese Weise Tausende neuer Molekülstrukturen aufgeklärt werden können.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft
Meistgelesene News
Weitere News von unseren anderen Portalen
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Massenspektrometrie
Die Massenspektrometrie ermöglicht es uns, Moleküle aufzuspüren, zu identifizieren und ihre Struktur zu enthüllen. Ob in der Chemie, Biochemie oder Forensik – Massenspektrometrie eröffnet uns ungeahnte Einblicke in die Zusammensetzung unserer Welt. Tauchen Sie ein in die faszinierende Welt der Massenspektrometrie!
Themenwelt Massenspektrometrie
Die Massenspektrometrie ermöglicht es uns, Moleküle aufzuspüren, zu identifizieren und ihre Struktur zu enthüllen. Ob in der Chemie, Biochemie oder Forensik – Massenspektrometrie eröffnet uns ungeahnte Einblicke in die Zusammensetzung unserer Welt. Tauchen Sie ein in die faszinierende Welt der Massenspektrometrie!