Membranproteine von Bakterien und Menschen zeigen überraschende Gemeinsamkeiten
Forschungskooperation deckt Struktur und Funktion von PspA auf und vermutet evolutionäre Abstammung des Bakterienproteins von der ESCRT-Proteinfamilie
Die Zellen einfacher Organismen, wie Bakterien, sind ebenso wie menschliche Zellen von einer Zellmembran umgeben. Diese Membran übernimmt vielfältige Aufgaben, unter anderem schützt sie die Zelle vor Stress. Teams der Johannes Gutenberg-Universität Mainz (JGU) und des Forschungszentrums Jülich haben unter Beteiligung der Heinrich-Heine-Universität Düsseldorf (HHU) nun in einem Kooperationsprojekt herausgefunden, dass ein Membranprotein, welches in Bakterien vorkommt, eine ähnliche Struktur und Funktion aufweist wie Proteine, die beim Menschen für den Umbau oder Wiederaufbau der Zellmembran sorgen. Bisher war ein Zusammenhang zwischen den beiden Proteingruppen nicht bekannt.
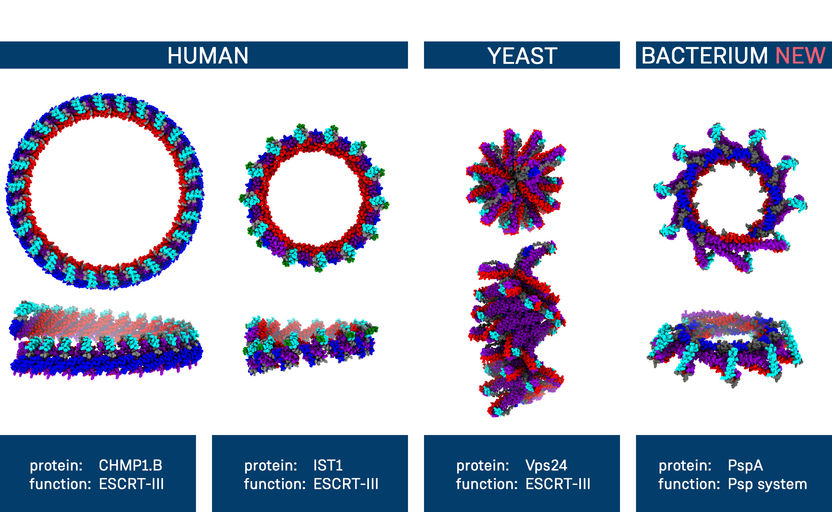
Strukturelle Gemeinsamkeiten zwischen verschiedenen Membranproteinen
Copyright: Benedikt Junglas, Dirk Schneider, Carsten Sachse
Wichtiger Akteur der bakteriellen Stressreaktion
Das Phagen-Schock-Protein-System, kurz als Psp-System bezeichnet, wurde vor etwa 30 Jahren in Bakterien entdeckt. Damals wurde es als eine Antwort von Escherichia coli-Bakterien auf eine Infektion mit speziellen Viren, sogenannten Bakteriophagen, angesehen. Später wurde klar, dass seine Funktion beim Schutz der Zellmembran über die spezifische Reaktion auf eine Bakteriophagen-Infektion hinausgeht. Auch osmotischer Stress, Hitze, Zellgifte oder Fehlstellen in der Membranhülle können eine Psp-System-Antwort auslösen.
„Wir wissen heute, dass das Psp-System bei vielen Arten von Membranstress aktiviert wird. Viele molekulare Details sind jedoch noch rätselhaft“, erklärt Prof. Dr. Dirk Schneider, Leiter der Arbeitsgruppe Membranbiochemie an der JGU. „Darum haben wir begonnen, uns Proteine des Psp-Systems genauer anzusehen.“ Mit seinem Team hatte er vor Kurzem bereits aufgedeckt, wie der Psp-Vertreter IM30 einen Schutzschild teppichartig auf der Zellmembran ausbreitet, um damit Membranstress abzufedern.
In ihrer neuen Arbeit haben die Wissenschaftler den Hauptakteur des Psp-Systems, das Phagen-Schock-Protein A (PspA), genauer unter die Lupe genommen. Mithilfe der Kryoelektronenmikroskopie wurde sichtbar, wie PspA lange, spiralförmige Röhrchen bildet, die im innenliegenden Hohlraum eine Biomembran einschließen können. Die hochaufgelösten Bilder zeigen nun erstmals, wie PspA einzelne Membranen lokal auflöst und danach zu größeren Einheiten umgestaltet oder auch die Bildung neuer Membranstrukturen vermittelt.
„PspA-Bausteine können tausendfach aneinandergelegt werden und riesige spiralförmige Strukturen bilden. Sie sind daher ein ideales Untersuchungsobjekt für unsere kryoelektronenmikroskopische Strukturanalyse“, so Prof. Dr. Carsten Sachse vom Forschungszentrum Jülich und der Heinrich-Heine-Universität Düsseldorf. Die Untersuchungen konnten zusammen mit Dr. Benedikt Junglas, ehemaliger Mainzer Doktorand von Dirk Schneider, in Jülich vorgenommen werden. Das Forschungszentrum Jülich betreibt am Ernst Ruska-Centrum für Mikroskopie und Spektroskopie mit Elektronen einige der leistungsstärksten Elektronenmikroskope Europas, seit Kurzem auch Kryomikroskope für die Untersuchung von schockgefrorenen biologischen Proben.
PspA gestaltet Membranen um
Die Struktur eines Proteins ist außerordentlich wichtig für seine Funktion, und ein Defekt in der Struktur kann die Funktion eines Proteins beeinträchtigen. „Unter dem Mikroskop wurde uns klar, dass PspA eine ähnliche Struktur besitzt wie ESCRT-III-Proteine, an denen unser Labor bereits gearbeitet hatte – für uns eine völlige Überraschung, die zeigt, wie wichtig es ist, Proteinstrukturen im Detail aufzuklären“, sagt Sachse. „Genetisch haben sich die beiden Proteingruppen nach Milliarden von Jahren so weit voneinander entfernt, dass die Gemeinsamkeiten nur noch anhand der 3-D-Struktur nachweisbar sind.“
ESCRT-III-Proteine kommen in allen Lebewesen mit einem echten Zellkern vor, also auch in menschlichen Zellen. Hier sind die ESCRT-III-Proteinkomplexe an dem Umbau und der Reparatur der Zellmembran beteiligt, spielen aber auch eine zentrale Rolle bei einer Reihe von weiteren Zellprozessen. Bei Bakterien kannte man bisher keine Proteine aus der ESCRT-III-Familie. „PspA gehört somit in die gleiche Gruppe von Proteinen wie ESCRT-III. Die beiden übernehmen an der Membran im Innern der Zellen ähnliche Aufgaben“, fasst Schneider zusammen.
„Aufgrund der ähnlichen strukturellen und funktionalen Eigenschaften von PspA und den eukaryotischen ESCRT-III-Proteinen haben wir PspA hier als ein bakterielles Mitglied der evolutionär konservierten ESCRT-III-Superfamilie von membran-umgestaltenden Proteinen identifiziert“, schreiben die Autoren in ihrem Beitrag für die Zeitschrift Cell.