Replikationszyklus von SARS-COV-2 in 3D
Untersuchung wie SARS-CoV-2 die Maschinerien der Wirtszellen übernimmt wird dabei helfen therapeutische Strategien zu entwickeln
Im Zuge der weltweiten Corona-Pandemie versuchen Wissenschaftler nicht nur Impfstoffe und Medikamente gegen die Krankheit zu finden, sondern auch kontinuierlich mehr über das Virus selbst zu erfahren. „Inzwischen können wir damit rechnen, dass das Coronavirus saisonal wird,“ erklärt Ralf Bartenschlager, Professor in der Abteilung für Infektionskrankheiten der Universität Heidelberg. „Es ist daher dringend notwendig, sowohl prophylaktische als auch therapeutische Strategien gegen dieses Virus zu entwickeln und umzusetzen.“ In einer neuen Studie führte Bartenschlager daher mit Unterstützung des Schwab-Teams am Europäischen Laboratorium für Molekularbiologie in Heidelberg und unter Verwendung der Elektronenmikroskopie-Anlage des EMBL eine detaillierte bildgebende Analyse durch, um festzustellen, wie das Virus infizierte Zellen umprogrammiert.
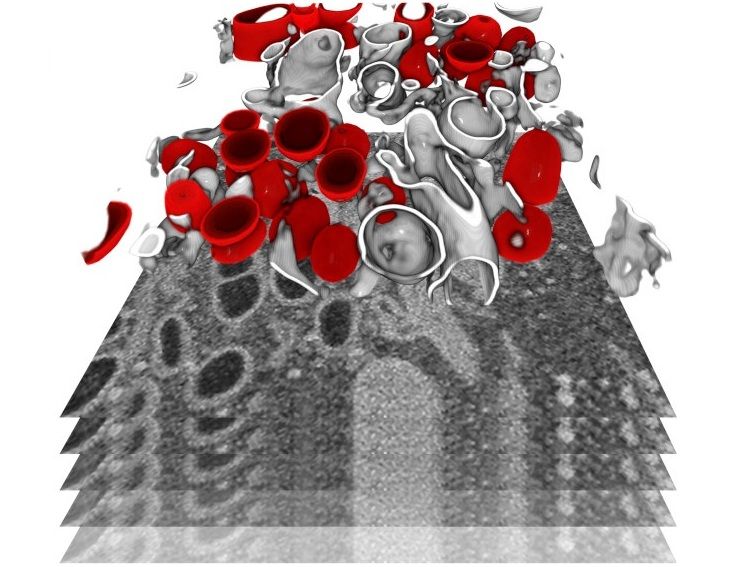
Infizierte Zellen wurden mittels fokussierter Ionenstrahl-Rasterelektronenmikroskopie abgebildet, einer leistungsstarken Technik, um die Organisation einer Zelle auf subzellulärer Ebene in 3D darzustellen. In diesem Bild wurde ein Subvolumen einer Zelle segmentiert, um membrangebundene Organellen (in grau) und Doppelmembran-Vesikel (in rot) darzustellen – ein virusspezifisches Kompartiment, in dem das virale Genom in großen Mengen repliziert wird.
Julian Hennies/EMBL
Zellen, die durch das Virus infiziert werden, sterben innerhalb von nur 24 bis 48 Stunden ab. Dies deutet darauf hin, dass das Virus die menschliche Zelle in einer Weise schädigt, dass diese umprogrammiert und gezwungen wird, Virenpartikel zu produzieren. Die Identifizierung dieser morphologischen Veränderungen innerhalb einer Zelle war daher das Hauptziel des Projekts. „Um Medikamente zu entwickeln, welche die virale Replikation unterdrücken und damit auch die Folgen der Infektion und des virusinduzierten Zelltods, ist es entscheidend, die biologischen Mechanismen, die den Replikationszyklus des Virus antreiben, besser zu verstehen,“ erklärt Bartenschlager. Deshalb nutzte das Team die bildgebenden Einrichtungen des EMBL und modernste bildgebende Verfahren, um die 3D-Architektur von SARS-CoV-2 sowie die virusbedingten Veränderungen der infizierten Zellen zu bestimmen.
Das Team war in der Lage, 3D-Rekonstruktionen von ganzen Zellen und den subzellulären Kompartimenten zu erstellen. „Wir liefern kritische Einblicke in virusinduzierte Strukturveränderungen in den untersuchten menschlichen Zellen,“ erklärt Bartenschlager. Die Bilder zeigten eine offensichtliche und massive Veränderung in den Endomembransystemen der infizierten Zellen – ein System, das der Zelle dazu dient, verschiedene Abteilungen und Orte zu definieren. Das Virus verändert Membranen auf eine Art und Weise, dass es seine eigenen Replikationsorganellen produzieren kann. Dies sind Mini-Replikationsgefäße, in denen das virale Genom enorm vervielfältigt wird. Dazu benötigt das Virus die Oberfläche von Membranen, und das Virus holt sich diese Membranen von den Zellen. Es baut sie so um, dass eine Organelle entsteht, die ein sehr charakteristisches Aussehen hat. Die Wissenschaftler beschreiben es als eine massive Ansammlung von Blasen: zwei Membranschichten, die einen großen Ballon bilden. Innerhalb dieser Ballons – die ein sehr abgeschirmtes Behältnis bilden – werden die viralen Genome repliziert und vermehrt.
Diese auffälligen Veränderungen zeigen sich bereits wenige Stunden nach der Infektion in den infizierten Zellen. „Wir sehen, wie und wo sich das Virus innerhalb der Zelle vermehrt und wie es seine Wirtszellen manipuliert, um nach der Vermehrung freigesetzt zu werden,“ sagte Yannick Schwab. Über die Entstehung und Entwicklung der Auswirkungen, die das Coronavirus im menschlichen Körper hervorruft, war bisher wenig bekannt. Dazu gehören fehlende Kenntnisse über den Mechanismus, wie die Infektion zum Absterben der infizierten Zellen führt. Mit den jetzt vorhandenen Informationen kann die Entwicklung von Therapien, die die Schwere der Krankheit verringern, beschleunigt werden.
Das Team sorgte dafür, dass die gesammelten Informationen und insbesondere die beispiellose Sammlung von 3D-Strukturinformationen von virusinduzierten Substrukturen von jedermann genutzt werden können. „Ich glaube, dass wir damit einen Präzedenzfall dafür schaffen, dass wir alle Daten, die wir produziert haben, mit der wissenschaftlichen Gemeinschaft teilen. Sie stellen eine beeindruckende Ressource für Wissenschaftler dar,“ betont Yannick Schwab. “Auf diese Weise können wir die weltweiten Bemühungen unterstützen, zu untersuchen, wie SARS-CoV-2 mit seinem Wirt interagiert. Das Team hofft, dass die gesammelten Informationen zur Entwicklung antiviraler Medikamente beitragen werden.
Dem Team ist es trotz der schwierigen Umstände gelungen, die Studie in unglaublich kurzer Zeit zu erstellen. „Die halbe Welt – und natürlich auch Heidelberg – war im Lockdown, und wir mussten fast täglich improvisieren, um uns an die Situation anzupassen. Ob am EMBL oder von zu Hause aus, alle waren extrem motiviert und haben großzügig ihre Zeit und ihr Expertenwissen zur Verfügung gestellt,“ betont Schwab stolz. „Die Geschwindigkeit, mit der wir arbeiteten, und die Menge an Daten, die produziert wurde, ist bemerkenswert.“






















































