Synthetische antivirale Proteine hemmen SARS-CoV-2 im Labor
Computer-designte Miniproteine wirken als potente, stabile antivirale Medikamente, die die Coronavirus-Infektion kultivierter menschlicher Zellen blockieren
Am Computer entworfene kleine Proteine schützen jetzt nachweislich im Labor gewachsene menschliche Zellen vor SARS-CoV-2, dem Coronavirus, das Covid-19 verursacht.
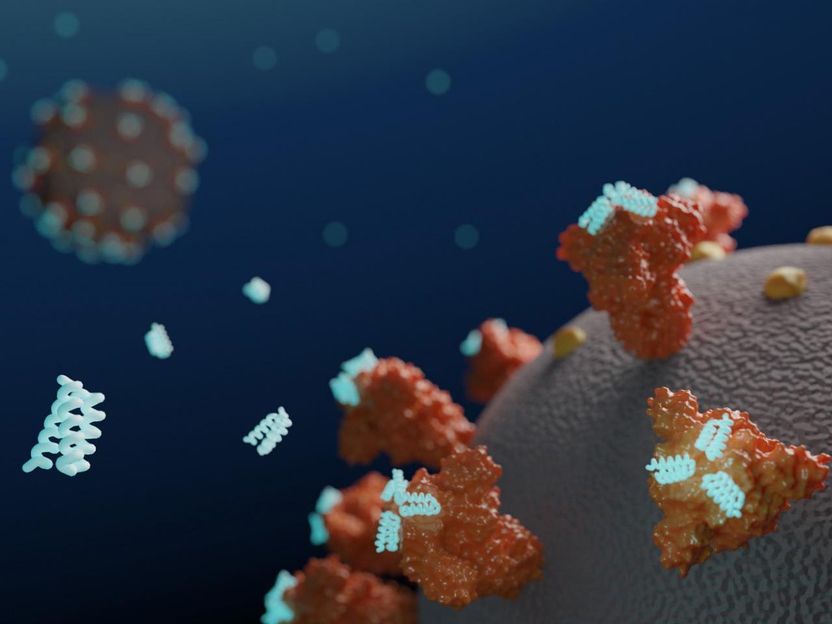
Eine Vorstellung der Künstler, wie kleine synthetische Proteine, die von Grund auf am Computer entworfen wurden, eine antivirale Wirkung haben können, indem sie mit Spike-Proteinen an SARS-CoV-2 binden. Die Bindung greift in den Infektionsmechanismus des Virus ein und versucht, es daran zu hindern, sich an Zellen anzulagern und in diese einzudringen.
UW Medicine Institute for Protein Design
In den Experimenten konkurrierte der führende antivirale Kandidat mit dem Namen LCB1 in seiner Schutzwirkung mit den bekanntesten SARS-CoV-2 neutralisierenden Antikörpern. LCB1 wird derzeit bei Nagetieren evaluiert.
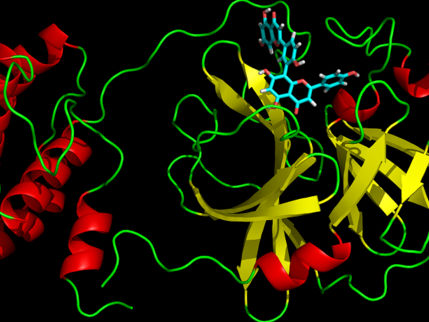
Coronaviren sind mit so genannten Spike-Proteinen besetzt. Diese heften sich an menschliche Zellen, damit das Virus eindringen und sie infizieren kann. Die Entwicklung von Medikamenten, die in diesen Eindringmechanismus eingreifen, könnte zur Behandlung oder sogar zur Prävention der Infektion führen.
Forscher des Institute for Protein Design an der University of Washington School of Medicine verwendeten Computer, um neue Proteine zu entwickeln, die eng an das SARS-CoV-2-Spike-Protein binden und es daran hindern, Zellen zu infizieren.
Ab Januar wurden mehr als zwei Millionen Kandidaten für Spike-bindende Proteine am Computer entworfen. Anschließend wurden über 118.000 hergestellt und im Labor getestet.
"Obwohl noch umfangreiche klinische Tests erforderlich sind, glauben wir, dass die besten dieser computergenerierten antiviralen Medikamente recht vielversprechend sind", sagte Hauptautorin Longxing Cao, eine Postdoc-Stipendiatin am Institut für Proteindesign.
"Sie scheinen eine SARS-CoV-2-Infektion mindestens ebenso gut zu blockieren wie monoklonale Antikörper, sind aber viel einfacher herzustellen und weitaus stabiler, so dass eine Kühlung möglicherweise überflüssig wird", fügte er hinzu.
Die Forscher schufen antivirale Proteine durch zwei Ansätze. Erstens wurde ein Segment des ACE2-Rezeptors, an den SARS-CoV-2 auf natürliche Weise auf der Oberfläche menschlicher Zellen bindet, in eine Reihe von kleinen Proteingerüsten eingebaut.
Zweitens wurden vollständig synthetische Proteine von Grund auf neu entwickelt. Mit der letztgenannten Methode wurden die wirksamsten antiviralen Medikamente hergestellt, darunter LCB1, das pro Masse etwa sechsmal wirksamer ist als die wirksamsten monoklonalen Antikörper, über die bisher berichtet wurde.
An dieser Arbeit arbeiteten Wissenschaftler der University of Washington School of Medicine in Seattle und der Washington University School of Medicine in St. Louis zusammen.
"Unser Erfolg bei der Entwicklung hochaffiner antiviraler Proteine von Grund auf ist ein weiterer Beweis dafür, dass computergestütztes Proteindesign zur Herstellung vielversprechender Arzneimittelkandidaten verwendet werden kann", sagte der leitende Autor und Howard Hughes Medical Institute Investigator David Baker, Professor für Biochemie an der UW School of Medicine und Leiter des Instituts für Proteindesign. Im Jahr 2019 hielt Baker einen TED-Vortrag darüber, wie Proteindesign eingesetzt werden könnte, um Viren zu stoppen.
Um zu bestätigen, dass sich die neuen antiviralen Proteine wie beabsichtigt an das Spike-Protein des Coronavirus anlagerten, sammelte das Team Momentaufnahmen der beiden Moleküle, die mit Hilfe der Kryo-Elektronenmikroskopie interagieren. Diese Experimente wurden von Forschern in den Labors von David Veesler, Assistenzprofessor für Biochemie an der UW School of Medicine, und Michael S. Diamond, dem Herbert S. Gasser-Professor in der Abteilung für Infektionskrankheiten an der Washington University School of Medicine in St. Louis, durchgeführt.
"Die hyperstabilen Minibinder bieten vielversprechende Ansatzpunkte für neue SARS-CoV-2-Therapeutika", schrieb das Antivirus-Forschungsteam in ihrem Vorabdruck zur Studie, "und veranschaulichen die Leistungsfähigkeit des rechnergestützten Proteindesigns zur raschen Generierung potenzieller therapeutischer Kandidaten gegen pandemische Bedrohungen".
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.