Neue Software spürt krankmachende Genveränderungen auf
Software findet „Nadel im Heuhaufen“ z.B. bei der Diagnose von seltenen Erkrankungen
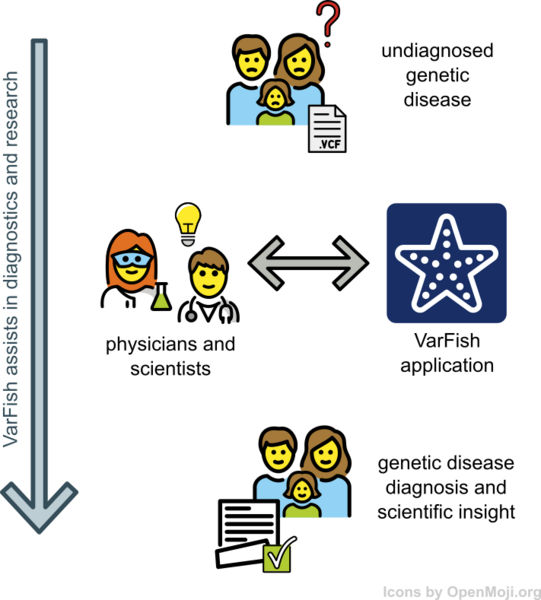
Oft ist es ein einziger Fehler im Erbgut, der eine schwere Erkrankung zur Folge hat. Doch weil zwei gesunde Menschen schon etwa drei Millionen Unterschiede in ihrem Erbgut aufweisen, ist es gar nicht so leicht, die eine, krank machende Abweichung zu entdecken und von einer harmlosen Genvariante zu unterscheiden. Wissenschaftler der Core Unit Bioinformatics des Berlin Institute of Health (BIH) haben nun gemeinsam mit Kollegen von der Charité – Universitätsmedizin Berlin und des Max-Delbrück-Centrums (MDC) in der Helmholtzgemeinschaft eine Software entwickelt, die dabei hilft, die „Nadel im Heuhaufen“ zu finden. Besonders wichtig ist dies bei der Diagnose von sogenannten „Seltenen Erkrankungen“. Die Software „VarFish“ ist frei verfügbar und in der aktuellen Ausgabe der Fachzeitschrift "Nucleic Acids Research" beschrieben.
Berlin Institute of Health (BIH)
Seltene Erkrankungen sind gar nicht so selten: Etwa vier Millionen Menschen in Deutschland sind insgesamt betroffen, doch von jeder einzelnen der geschätzt 8.000 verschiedenen Krankheiten treten meist nur wenige Fälle auf. Das hat zur Folge, dass es oft Jahre dauert, bis die Betroffenen die richtige Diagnose erhalten, was jedoch die Voraussetzung für eine wirksame Therapie darstellt. „Häufig landen die betroffenen Eltern nach einer jahrelangen Odyssee bei uns“, erzählt Dr. Nadja Ehmke, Fachärztin im Institut für Medizinische Genetik und Humangenetik der Charité. „Sie haben ein krankes Kind, das sich geistig oder körperlich nicht richtig entwickelt und wollen nun wissen, warum. Wenn wir ihnen dann aufgrund einer genetischen Analyse erklären können, wo der Fehler liegt, ist das für die Eltern oft eine riesengroße Erleichterung, auch wenn das noch nicht bedeutet, dass man auch therapeutisch etwas für ihr Kind tun kann.“ So können sich die Eltern mit anderen Betroffenen austauschen, Selbsthilfegruppen besuchen oder gründen und auch besser einschätzen, ob ein weiteres Kind ebenfalls erkranken könnte.
Leistungsfähige Software entdeckt Unterschiede
Für die genetische Analyse isolieren die Wissenschaftler das Erbgut aus Blutzellen der Patienten, lesen die Sequenz, also die Abfolge der Buchstaben und vergleichen diese mit dem Erbgut von Eltern, Geschwistern oder den vorhandenen Erbgut-Analysen in großen Datenbanken. „Hier stößt der Mensch naturgemäß an seine Grenzen“, berichtet Dr. Dieter Beule, der Leiter der BIH Core Unit Bioinformatics. „Selbst wenn wir nur die eiweißkodierenden Bereiche des Erbguts analysieren, das so genannte Exom, müssen wir Millionen von Bausteinen vergleichen. Dazu benötigt man leistungsfähige Software, die die Unterschiede entdeckt.“ Doch selbst zwischen zwei gesunden Personen unterscheidet sich das Erbgut in den kodierenden Bereichen an rund 30.000 Positionen. Welche der vielen Abweichungen ist die krankmachende? Genau hier wurden Dieter Beule und sein Team aktiv: Sie entwickelten „VarFish“.
“VarFish“ vergleicht die eingegebene Sequenz des Patienten mit Sequenzen aus weltweit zusammengetragenen Datenbanken. Die Wissenschaftler der Core Unit Bioinformatics griffen dabei auf viele offene und freie Datenressourcen zurück, wie die amerikanischen Datenbanken vom National Center for Biotechnology Information, der Universität von Washington, Seattle, dem European Bioinformatics Institute EBI in Cambridge, UK, aber auch auf Datenbanken und Algorithmen der Charité und des BIH.
Sekundenschneller Abgleich
„Innerhalb weniger Sekunden kann VarFish 29.950 von den 30.000 Unterschieden ausschließen“, sagt Dieter Beule. „Denn viele dieser Abweichungen findet die Software z.B. auch in den Sequenzen der Bevölkerungsdaten und führen dort offenbar nicht zu auffälligen Problemen und sind daher aller Wahrscheinlichkeit nach nicht für die seltene Erkrankung verantwortlich.“ Die übrig gebliebenen 50 Genvarianten vergleichen die Wissenschaftler dann mit bereits bekannten Erbkrankheiten und können so den Kreis der in Frage kommenden, ursächlich verantwortlichen Veränderungen weiter auf etwa zehn eingrenzen.“
In manchen Fällen entdecken auch Wissenschaftler in Forschungsstudien Mutationen in einem bekannten Gen. „VarFish“ ermöglicht eine rasche Prüfung auf Mutationen in bekannten Genen“, erklärt Professorin Ute Scholl, die am BIH eine Johanna-Quandt Professur innehat. „Das ist wichtig, weil es uns Hinweise geben kann, welches Medikament helfen könnte und klinisch weiterverfolgt werden sollte." Wenn keine Mutationen in bekannten Genen vorliegen, kann mit Hilfe von VarFish innerhalb einer größeren Familie oder im Vergleich mehrerer betroffener Familien mit der gleichen Erkrankung ein neues Kandidatengen identifiziert werden. Was genau Mutationen in einem solchen Kandidatengen bewirken, kann anschließend oft im Labor geklärt werden. Die Arbeitsgruppe von Ute Scholl schleust die mutierten Gene in Zellkulturen oder in Mäuse ein und kann so beobachten, welche Auswirkungen die Mutation hat. „Auf diese Weise haben wir kürzlich ein neues Gen für eine seltene angeborene Form von Bluthochdruck charakterisiert", berichtet die Medizinerin. Doch die Medikamentenentwicklung oder gar zielgerichtete Behandlung liegt eher in weiter Ferne. „Wir wollen zunächst einmal verstehen, wie die Krankheit überhaupt entsteht und woher die Beschwerden rühren.“
Schnelle und zielsichere Diagnose
Dr. Manuel Holtgrewe, Bioinformatiker in der Core Unit und Erstautor der Publikation, freut sich darüber, dass die neue Software so viele Anwender findet: „Innerhalb der ersten Wochen wurde VarFish schon hundertfach eingesetzt, Wissenschaftler*innen und Ärzt+innen weltweit haben damit gearbeitet. Intern haben wir schon tausende Datensätze damit verarbeitet.“ Gerade bei der Erforschung und Behandlung von Seltenen Erkrankungen ist die internationale Zusammenarbeit wichtig, weil jede einzelne Mutation ja meist nur wenige Male in jedem Land auftritt. Professor Stefan Mundlos, Direktor des Instituts für Medizinische Genetik und Humangenetik der Charité und der Forschungsgruppe "Development & Disease" am Max-Planck-Institut für Molekulare Genetik Berlin, berichtet: „VarFish hat uns sehr dabei geholfen, Teilnehmer*innen für unsere Klinischen Studien schnell und zielsicher zu einer Diagnose zur verhelfen.“
Manuel Holtgrewe und Dieter Beule planen nun, „VarFish“ dahingehend zu erweitern, dass auch die genomweite Analyse von sogenannten strukturellen Varianten möglich wird. Außerdem sollen weitere Funktionen für die effektive und sichere standortübergreifende Zusammenarbeit von Humangenetikern eingefügt werden. „VarFish unterstützt die Anwender*innen bei der Analyse ihrer molekulargenetischen Daten sowohl in der Grundlagenforschung als auch in der klinischen Anwendung. Die Mission des BIH ist ja die Translation, also die Übertragung von Ergebnissen aus der Forschung in die Klinik und umgekehrt, von Beobachtungen in der Klinik zurück ins Labor. Mit unserer Software „VarFish“ unterstützen wir genau dieses Anliegen“, sagt Dieter Beule.
Originalveröffentlichung
Manuel Holtgrewe, Oliver Stolpe, Mikko Nieminen, Stefan Mundlos, Alexej Knaus, Uwe Kornak, Dominik Seelow, Lara Segebrecht, Malte Spielmann, Björn Fischer-Zirnsak, Felix Boschann, Ute Scholl, Nadja Ehmke, Dieter Beule; "VarFish: comprehensive DNA variant analysis for diagnostics and research"; Nucleic Acids Research; 2020