Röntgenstrahlen vergrößern die Proteinstruktur im 'Herzen' des COVID-19-Virus
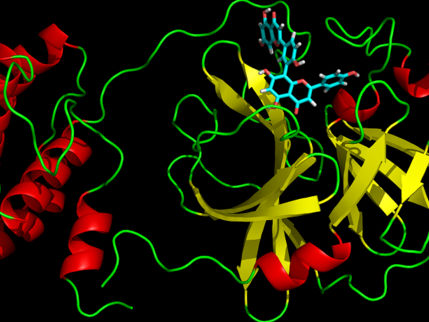
Das Protease-Protein hat sowohl die Form eines Herzens als auch die Funktion eines Herzens, wodurch sich das Virus vermehren und verbreiten kann
Ein Forscherteam in den nationalen Laboratorien Oak Ridge und Argonne des Department of Energy hat die ersten Röntgenmessungen bei Raumtemperatur an der SARS-CoV-2-Hauptprotease - dem Enzym, das die Vermehrung des Virus ermöglicht - durchgeführt.
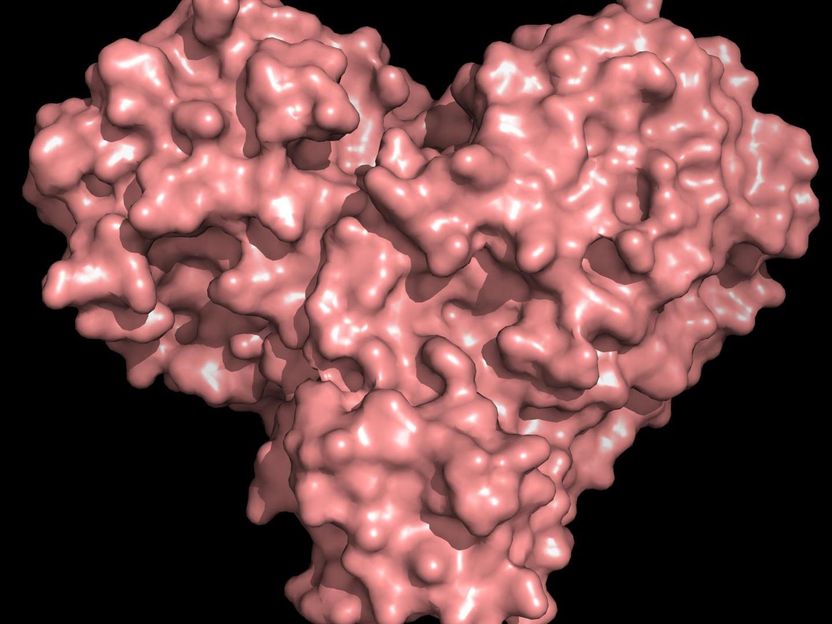
Das Protease-Protein hat sowohl die Form eines Herzens als auch die Funktion eines Herzens, wodurch sich das Virus vermehren und verbreiten kann. Eine Hemmung der Protease würde die Virusvermehrung blockieren.
Andrey Kovalevsky/ORNL, U.S. Dept. of Energy
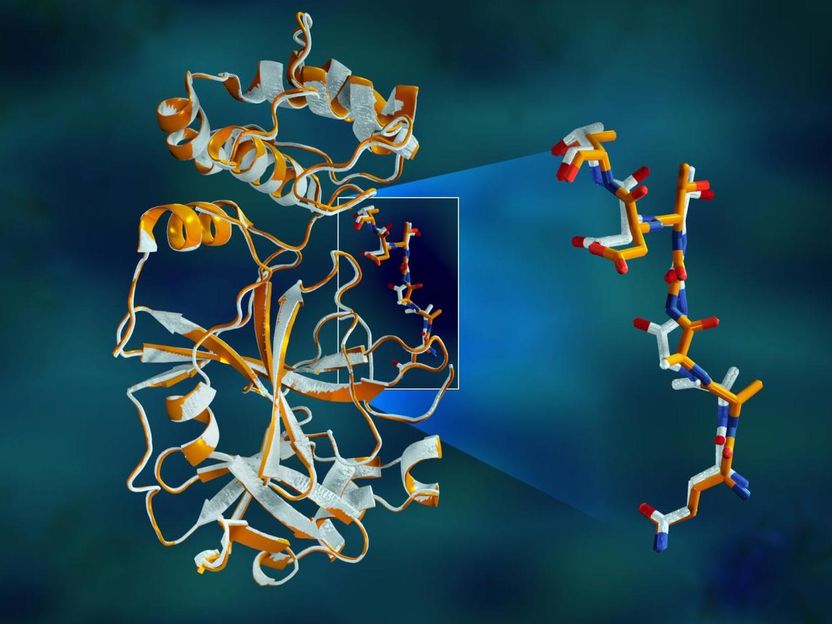
Überlappende Röntgendaten der SARS-CoV-2-Hauptprotease zeigen strukturelle Unterschiede zwischen dem Protein bei Raumtemperatur (orange) und der kryogenisch gefrorenen Struktur (weiß).
Jill Hemman/ORNL, U.S. Dept. of Energy
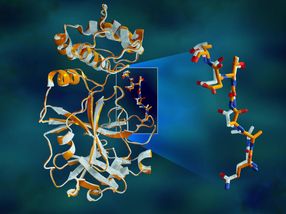
Die Röntgenmessungen markieren einen wichtigen ersten Schritt im Hinblick auf das letztendliche Ziel der Forscher, ein umfassendes 3D-Modell des enzymatischen Proteins zu erstellen. Das Modell wird verwendet, um Supercomputing-Simulationen voranzutreiben, die darauf abzielen, Medikamenteninhibitoren zu finden, die den Replikationsmechanismus des Virus blockieren und dazu beitragen, die COVID-19-Pandemie zu beenden. Ihre Forschungsergebnisse sind öffentlich zugänglich und wurden in der Zeitschrift Nature Communications veröffentlicht.
SARS-CoV-2 ist das Virus, das die Krankheit COVID-19 verursacht. Das Virus vermehrt sich, indem es lange Ketten von Proteinen exprimiert, die durch das Protease-Enzym in kleinere Längen geschnitten werden müssen.
"Die Protease ist für den Lebenszyklus des Virus unverzichtbar. Das Protein hat die Form eines Valentinsherzens, aber es ist wirklich das Herz des Virus, das es ihm ermöglicht, sich zu vermehren und zu verbreiten. Wenn man die Protease hemmt und das Herz stoppt, kann das Virus die Proteine, die für seine Replikation unerlässlich sind, nicht produzieren. Deshalb gilt die Protease als ein so wichtiger Angriffspunkt für Medikamente", sagt Andrej Kowaljewski, korrespondierender Autor am ORNL. Während die Struktur aus kryogenisch konservierten Kristallen bekannt ist, "ist dies das erste Mal, dass die Struktur dieses Enzyms bei Raumtemperatur gemessen wurde, was von Bedeutung ist, weil sie nahe der physiologischen Temperatur liegt, bei der die Zellen arbeiten".
Um ein vollständiges Modell der Proteinstruktur zu erstellen, müssen die einzelnen Elemente innerhalb der Struktur und ihre Anordnung identifiziert werden. Röntgenstrahlen sind ideal für den Nachweis schwerer Elemente wie Kohlenstoff-, Stickstoff- und Sauerstoffatome. Wegen der Intensität der Röntgenstrahlen in den meisten großen Synchrotronanlagen müssen biologische Proben typischerweise bei etwa 100 K oder etwa minus 280 Grad Fahrenheit kryogenisch eingefroren werden, um der Strahlung lange genug standhalten zu können, damit Daten gesammelt werden können.
Um die Lebensdauer der kristallisierten Proteinproben zu verlängern und sie bei Raumtemperatur zu messen, züchteten die Forscher des ORNL Kristalle, die größer waren als für Synchrotron-Kryo-Studien erforderlich, und benutzten ein hauseigenes Röntgengerät mit einem weniger intensiven Strahl.
"Die Züchtung von Proteinkristallen und das Sammeln von Daten ist ein langwieriger und zeitraubender Prozess. In der Zeit, die normalerweise für die Vorbereitung und den Transport der Probe zu einem Synchrotron benötigt wird, konnten wir die Kristalle züchten, die Messungen vornehmen und mit der Analyse der Daten beginnen", sagte Daniel Kneller vom ORNL, der Erstautor der Studie. "Und wenn eine Pandemie ausbricht und viele Wissenschaftler zur Untersuchung dieses Problems mobilisiert werden, bleibt kein Tag zur freien Verfügung".
Das Enzym Protease besteht aus Aminosäureketten mit einem sich wiederholenden Muster von Stickstoff-Kohlenstoff-Kohlenstoff-Atomen, die das Rückgrat des Proteins bilden. Seitengruppen der Aminosäurebausteine oder "Reste" erstrecken sich von jedem der Kohlenstoffatome des zentralen Rückgrats. Das Enzym wird in eine spezifische 3D-Form gefaltet, wodurch spezielle Taschen entstehen, in denen sich ein Wirkstoffmolekül anlagern würde.
Die Studie zeigte signifikante strukturelle Unterschiede zwischen den Orientierungen des Rückgrats und einigen der Rückstände in den bei Raumtemperatur gefrorenen und kryogenen Proben. Die Forschung legt nahe, dass durch das Einfrieren der Kristalle strukturelle Artefakte eingeführt werden könnten, die zu einem weniger genauen Verständnis der Proteasestruktur führen könnten.
Die Ergebnisse des Teams werden den Forschern unter der Leitung des Vorsitzenden des Gouverneurs der ORNL-Universität von Tennessee, Jeremy Smith, mitgeteilt, die mit Hilfe von Summit at ORNL - dem schnellsten Supercomputer der Nation - Simulationen zum Andocken von Medikamenten durchführen.
"Was die Forscher auf dem Gipfeltreffen tun, ist, bekannte Arzneimittelwirkstoffe zu nehmen und zu versuchen, sie rechnerisch an die Haupt-Protease zu binden, um sie für die Neuverwendung von Arzneimitteln zu nutzen, sowie nach neuen Ansatzpunkten für andere potenzielle Arzneimittelkandidaten zu suchen", sagte ORNL-Korrespondenzautor Leighton Coates. "Unsere Daten zur Raumtemperatur werden verwendet, um ein genaueres Modell für diese Simulationen zu erstellen und die Aktivitäten zur Entwicklung von Medikamenten zu verbessern.
Der nächste Schritt der Forscher zur Vervollständigung des 3D-Modells der SARS-CoV-2-Hauptprotease ist die Nutzung der Neutronenstreuung am Hochflussisotopenreaktor des ORNL und an der Spallationsneutronenquelle. Neutronen sind für die Lokalisierung der Wasserstoffatome unerlässlich, die bei vielen katalytischen Funktionen und bei der Entwicklung von Medikamenten eine entscheidende Rolle spielen.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.