Zweifarbtechnik zeigt Röntgenlaser-Schäden in Biomolekülen
Schritt zu umfassenderem Verständnis von Strukturänderungen in Proteinen durch Röntgenblitze
Mit Hilfe von Röntgenlasern lässt sich die räumliche Struktur von Biomolekülen atomgenau erkunden. Dabei können sie auch die Struktur auch von solchen Molekülen bestimmen, die mit anderen Methoden nur schwer zugänglich sind. Die empfindlichen Proben verdampfen allerdings durch die intensiven Röntgenblitze nahezu sofort. Ein internationales Wissenschaftlerteam hat nun in bisher unerreichtem Detail verfolgt, wie sich diese Strahlenschäden in Biomolekülen ausbreiten. Die Ergebnisse sind wichtig für Design und Interpretation solcher Röntgenlasermessungen, wie das Team, zu dem auch DESY-Theoretikerin Beata Ziaja vom Center for Free-Electron Laser Science CFEL gehört, im Fachblatt „Nature Communications“ berichtet.
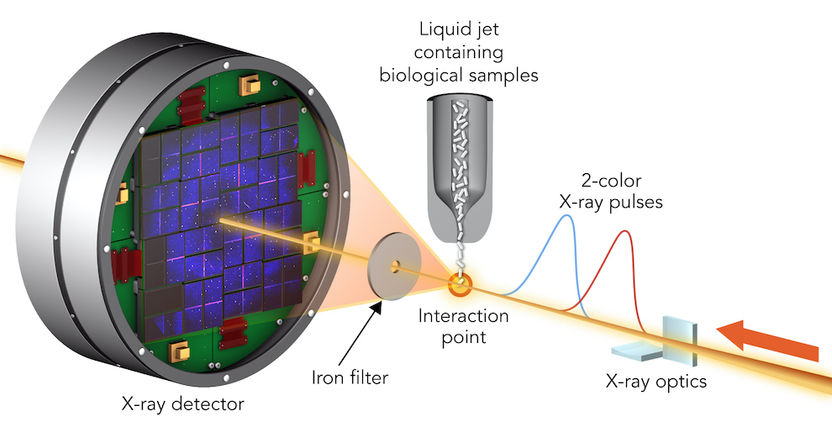
Versuchsaufbau am Röntgenlaser LCLS bei SLAC: Zwei leicht verzögerte Röntgenblitze (blau, rot) durchleuchten Proteinkristalle, die durch eine Düse in den Laserstrahl gesprüht werden. Der erste Blitz löst die Strukturänderungen in den Proteinen aus und wird anschließend von einer Eisenfolie geschluckt. Der zweite Puls erzeugt ein Streubild auf dem Detektor, aus dem die Strukturänderungen berechnet werden können.
SLAC, Greg Stewart
Die räumliche Struktur von Proteinen verrät Wissenschaftlern, wie diese Biomoleküle funktionieren und wie sie sich möglicherweise von Medikamenten beeinflussen lassen. Um die Struktur zu bestimmen, röntgen die Forscher kleine Kristalle, die sie zuvor aus dem zu untersuchenden Protein züchten. Die Kristalle beugen die Röntgenstrahlung auf charakteristische Weise. Aus dem resultierenden Beugungsmuster lässt sich dann die innere Struktur des Kristalls und seiner Bestandteile, der Proteine, berechnen.
Zahlreiche Proteinstrukturen sind auf diese Weise bestimmt worden, aber einige Proteine bilden lediglich Nanokristalle, bei denen extrem helles Röntgenlicht nötig ist, um ein brauchbares Beugungsmuster zu erzeugen. Freie-Elektronen-Röntgenlaser (XFEL) liefern die hellsten Röntgenblitze der Welt und ermöglichen damit selbst die Analyse von Nanokristallen.
Zwar verdampfen die empfindlichen Kristalle durch den Röntgenlaserblitz sofort, frühere Studien haben jedoch gezeigt, dass das Beugungsmuster schneller entsteht als der Kristall zerstört wird. Die Strukturinformation lässt sich daher noch auslesen, bevor der Kristall verdampft. Wie sich der Strahlenschaden allerdings genau in den Proteinen ausbreitet, ist weitgehend unbekannt. Um dies genauer zu untersuchen, hat ein Team unter Leitung von Ilme Schlichting vom Max-Planck-Institut für medizinische Forschung und Sébastien Boutet vom US-Beschleunigerzentrum SLAC nun eine Zweifarben-Röntgentechnik eingesetzt, die an SLACs Röntgenlaser Linac Coherent Light Source LCLS entwickelt worden ist.
Die Wissenschaftler nutzten dafür zwei Sorten kristallisierter Biomoleküle, den natürlichen Süßstoff Thaumatin sowie einen Komplex aus Lysozym (Eiweiß) und der Seltenen Erde Gadolinium. Diese Kristalle beschossen sie jeweils mit zwei Röntgenlaserblitzen von leicht unterschiedlicher Wellenlänge (Farbe), die bis zu 100 Femtosekunden auseinander lagen. Eine Femtosekunde ist ein Millionstel einer Milliardstel Sekunde. Der erste Puls traf jeweils den Kristall, stieß damit die Schädigung an, wurde dann aber von einer Eisenfolie absorbiert. Der zweite Puls hatte eine etwas andere Farbe, die von der Folie nicht absorbiert wurde. Er wurde von der Probe gebeugt und erzeugte das übliche Beugungsmuster auf dem Detektor, so dass sich mit ihm die vom ersten Puls ausgelöste Strukturänderung untersuchen ließ. Durch die variable Verzögerung zwischen beiden Pulsen ließ sich die Fortpflanzung des Strahlenschadens zu verschiedenen Zeiten messen.
„Um einen detaillierten Einblick in die vom ersten Röntgenpuls ausgelösten molekularen Prozesse zu erhalten, war die theoretische Modellierung unverzichtbar“, erläutert Ziaja. Das Team stellte fest, dass jene Teile eines Moleküls, die schwerere Atome als Sauerstoff enthalten, den Hauptteil der Röntgenschäden absorbieren. Auch die Kohlenstoffketten, die das Rückgrat aller Proteine bilden, änderten ihre Struktur mit der Zeit, allerdings in viel geringerem Maße. Diese Veränderungen waren dabei nicht im gesamten Molekül gleich, sondern traten in bestimmten Bereichen stärker auf als in anderen.
Um sicherzustellen, dass die Interpretation der Messdaten nicht durch das theoretische Modell verzerrt wurde, setzte das Team zwei unabhängige theoretische Simulationen ein: Eine vom DESY-Team, bestehend aus Ziaja, Malik Abdullah, Zoltan Jurek und Robin Santra, und eine von der RMIT-Universität in Melbourne und der University of Melbourne. „Beide unabhängigen Simulationen bestätigten die selektiv fortschreitenden Strahlenschäden, die vor allem Schwefel-Ionen betrafen und sie stark elektrisch aufluden“, berichtet Ziaja. „Sie zeigten auch, dass die korrelierte Dynamik der hochgeladenen Schwefel-Ionen viel langsamer verlief als erwartet, was auf den wesentlichen Einfluss der mikroskopischen Umgebung der Ionen zurückzuführen ist.“
Diese Ergebnisse zeigen, dass für zuverlässige Messungen in diesem Bereich die verschiedenen Teile einer Probe modelliert werden müssen, statt eine gleichmäßige Schädigung aller Teile der Moleküls anzunehmen. Die Untersuchung ist ein erster Schritt zu einem umfassenderen Verständnis davon, wie die energiereichen Röntgenlaserblitze die Struktur biologischer Moleküle verändern.
Das Team betont, dass das Prinzip „Beugung vor Zerstörung“ die korrekte räumliche Struktur von Biomolekülen liefert, bei der Auswertung allerdings bedacht werden sollte, ob Intensität und Dauer der Röntgenblitze die Struktur bereits beeinflusst haben könnte. „Unsere Analyse zeigt, dass ein besseres Verständnis der lokalen Strahlenschäden erforderlich ist, sowohl um das Design von XFEL-Untersuchungen solcher Systeme zu planen, als auch eine korrekte Interpretation sicherzustellen, die exakte chemische Informationen liefert“, betont Ziaja.