Neue CRISPR-Screening-Technologie für RNA
Auch für RNA-Viren wie dem Coronavirus
CRISPR-basierte genetische Screens haben den Wissenschaftlern geholfen, Gene zu identifizieren, die bei der Sichelzellanämie, der Krebs-Immuntherapie, der Metastasierung von Lungenkrebs und vielen anderen Krankheiten eine Schlüsselrolle spielen. Diese genetischen Screens sind jedoch in ihrem Umfang begrenzt: Sie können nur die DNA bearbeiten oder auf sie abzielen. Für viele Regionen des menschlichen Genoms ist es möglicherweise nicht möglich, die DNA gezielt zu bearbeiten, und andere Organismen, wie RNA-Viren wie Coronaviren oder Grippe, können mit den bestehenden CRISPR-Screens, die auf die DNA abzielen, überhaupt nicht ins Visier genommen werden.
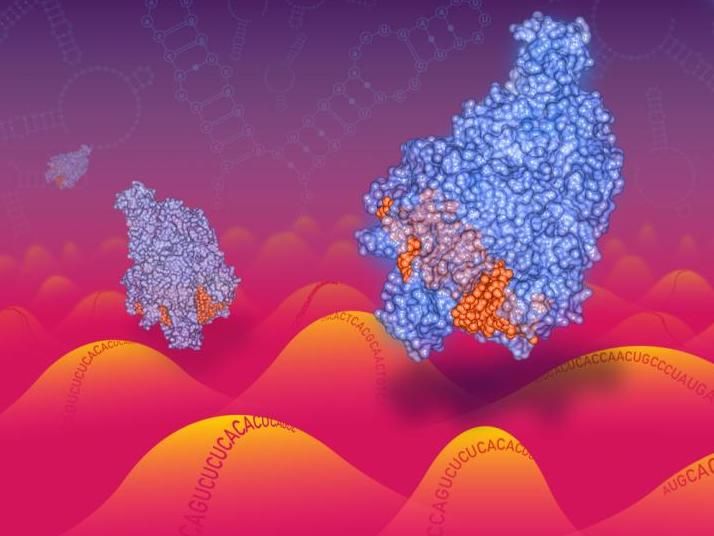
Cas13-Enzyme, die sich entlang einer RNA-Landschaft bewegen.
New York Genome Center
Nun haben Forscher im Labor von Neville Sanjana, PhD, am New Yorker Genomzentrum und der New Yorker Universität eine neue Art von CRISPR-Screening-Technologie entwickelt, die auf RNA abzielt und in Nature Biotechnology veröffentlicht wurde.
Die Forscher machten sich ein kürzlich charakterisiertes CRISPR-Enzym namens Cas13 zunutze, das auf RNA anstelle von DNA abzielt. Mit Cas13 haben sie eine optimierte Plattform für massiv-parallele genetische Screens auf RNA-Ebene in menschlichen Zellen entwickelt. Diese Screening-Technologie kann dazu verwendet werden, viele Aspekte der RNA-Regulierung zu verstehen und die Funktion von nicht-kodierenden RNAs zu identifizieren, d.h. von RNA-Molekülen, die zwar produziert werden, aber nicht für Proteine kodieren.
Indem sie Tausende von verschiedenen Stellen in menschlichen RNA-Transkripten ins Visier nahmen, entwickelten die Forscher ein auf maschinellem Lernen basierendes Vorhersagemodell, um die Identifizierung der effektivsten Cas13-Leit-RNAs zu beschleunigen. Die neue Technologie steht den Forschern über eine interaktive Website und eine Open-Source-Toolbox zur Verfügung, um die Effizienz von Leit-RNAs für kundenspezifische RNA-Ziele vorherzusagen, und bietet vorgefertigte Leit-RNAs für alle menschlichen proteincodierenden Gene.
"Wir gehen davon aus, dass die RNA-targeting Cas13-Enzyme einen großen Einfluss auf die Molekularbiologie und medizinische Anwendungen haben werden, doch ist über das Design der RNA mit hoher Targeting-Effektivität nur wenig bekannt", sagte Dr. Sanjana, Seniorautorin der Studie. "Wir haben uns vorgenommen, dies durch eine eingehende und systematische Studie zu ändern, um Schlüsselprinzipien und prädiktive Modellierung für ein möglichst effektives Leitliniendesign zu entwickeln.
Dr. Sanjana ist Mitglied der Kernfakultät des New Yorker Genomzentrums, Assistenzprofessorin für Biologie an der New Yorker Universität und Assistenzprofessorin für Neurowissenschaften und Physiologie an der medizinischen Fakultät der NYU.
Bei den Cas13-Enzymen handelt es sich um Typ-VI-CRISPR-Enzyme (geclusterte, regelmäßig ineinander greifende kurze palindromische Repeats), die vor kurzem als programmierbare RNA-gesteuerte RNA-Zielproteine mit Nuklease-Aktivität identifiziert wurden, die einen Zielgen-Knockdown ohne Veränderung des Genoms ermöglichen. Diese Eigenschaft macht Cas13 zu einem potenziell bedeutenden Therapeutikum zur Beeinflussung der Genexpression, ohne die Genomsequenz dauerhaft zu verändern.
"Dies ist die Art von technologischer Innovation, die wir am New Yorker Genomzentrum fördern und entwickeln. Diese neueste CRISPR-Technologie aus dem Sanjana-Labor hat aufregende Auswirkungen auf die Bereiche Genomik und Präzisionsmedizin", sagte Tom Maniatis, PhD, wissenschaftlicher Direktor der Evnin Family und Chief Executive Officer des New Yorker Genomzentrums.
Der Postdoktorand Hans-Hermann Wessels und der Doktorand Alejandro Méndez-Mancilla, die Co-Erstautoren der Studie sind, haben eine Reihe neuer Cas13-basierter Werkzeuge entwickelt und ein Transkript-Tiling und Permutationsscreening in Säugetierzellen durchgeführt. Insgesamt sammelten die Forscher Informationen für mehr als 24.000 RNA-Targeting-Guides.
"Wir haben Leit-RNAs über viele verschiedene Transkripte gekachelt, darunter auch mehrere menschliche Gene, bei denen wir den Transkript-Knockdown mittels Antikörperfärbung und Durchflusszytometrie leicht messen konnten", sagte Dr. Wessels. "Auf dem Weg dorthin haben wir einige interessante biologische Erkenntnisse aufgedeckt, die die Anwendung von RNA-targeting Cas13-Enzymen erweitern könnten". Zu den Erkenntnissen des Teams gehören zum Beispiel Erkenntnisse darüber, welche Regionen der Leit-RNA für die Erkennung einer Ziel-RNA wichtiger sind. Unter Verwendung von Tausenden von Leit-RNAs mit 1, 2 oder 3 Einzelbuchstaben-Mismatches zu ihrer Ziel-RNA identifizierten sie eine kritische "Saat"-Region, die besonders empfindlich auf Mismatches zwischen dem CRISPR-Leitfaden und dem Ziel reagiert. Diese Entdeckung wird den Wissenschaftlern bei der Entwicklung von Leit-RNAs helfen, um eine Off-Target-Aktivität auf unbeabsichtigte Ziel-RNAs zu vermeiden. Da eine typische menschliche Zelle etwa 100.000 RNAs exprimiert, ist die genaue Ausrichtung auf Cas13 nur des beabsichtigten Ziels für das Screening und die therapeutische Anwendung von entscheidender Bedeutung.
Zusätzlich zu einem besseren Verständnis der Cas13-Off-Targets könnte die "Saat"-Region für Biosensoren der nächsten Generation verwendet werden, die zwischen eng verwandten RNA-Arten genauer unterscheiden können. Insgesamt erhöht diese Studie die Anzahl der Datenpunkte aus früheren Cas13-Studien in Säugetierzellen um mehr als zwei Größenordnungen.
"Wir freuen uns besonders darüber, das optimierte Cas13-Screening-System zu nutzen, um nicht-kodierende RNAs zu untersuchen", sagte Méndez-Mancilla, Co-Erstautorin. "Dies erweitert die CRISPR-Toolbox für genetische und transkriptomische Vorwärts-Screens erheblich. In der Studie stellten die Forscher einen deutlichen Unterschied im Protein-Knockdown fest, wenn sie auf verschiedene proteincodierende und nicht-codierende Elemente der Boten-RNAs abzielten, und fanden Hinweise darauf, dass Cas13 mit anderen RNA-bindenden Proteinen konkurriert, die an der Transkriptverarbeitung und am Spleißen beteiligt sind.
Das Team hat vor kurzem ihr Leit-RNA-Prädiktionsmodell für eine besonders kritische Analyse genutzt: Der COVID-19-Notfall im Bereich der öffentlichen Gesundheit ist auf ein Coronavirus zurückzuführen, das ein RNA- und nicht ein DNA-Genom enthält. Mit Hilfe des Modells, das sie aus ihren massiv-parallelen Screens abgeleitet haben, haben die Forscher optimale Leit-RNAs identifiziert, die für zukünftige Nachweis- und Therapieanwendungen verwendet werden könnten.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.