VR für die Entwicklung neuer Medikamente nutzen
Mit Hilfe von Virtual Reality in Proteine 'einsteigen'
Forscher an der Universität Bristol leisten Pionierarbeit bei der Verwendung von virtueller Realität (VR) als Werkzeug zur Entwicklung der nächsten Generation von Medikamentenbehandlungen.

Benutzer, der mit einem Protein in VR interagiert.
University of Bristol
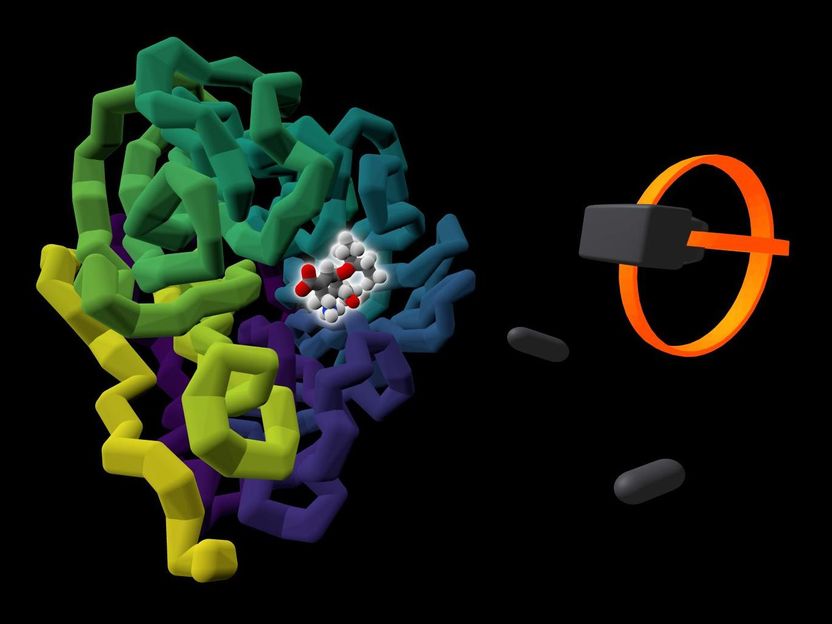
Das im Handel erhältliche Grippemittel Tamiflu (durch ein Leuchten hervorgehoben) ist an das virale Protein, die Neuraminidase (violett bis gelb gefärbt), gebunden. Eine Darstellung des VR-Headsets und der Steuergeräte eines Benutzers wird zusammen mit dem Protein dargestellt.
University of Bristol

Die Ergebnisse, die in der Zeitschrift PLOS ONE veröffentlicht wurden, beschreiben, wie Forscher VR nutzen, um zu verstehen, wie gängige Medikamente auf molekularer Ebene wirken.
Viele Medikamente sind kleine Moleküle, und um neue Medikamente zu entdecken, müssen Moleküle gefunden werden, die sich an biologische Ziele wie Proteine binden.
In der Studie konnten die Anwender mit Hilfe von VR in Proteine 'einsteigen' und sie und die daran bindenden Medikamente mit Hilfe interaktiver Molekulardynamik-Simulationen in VR (iMD-VR) im atomaren Detail manipulieren.
Mit diesem iMD-VR-Ansatz "dockten" die Forscher Wirkstoffmoleküle an Proteine an und konnten genau vorhersagen, wie die Wirkstoffe binden. Zu den untersuchten Systemen gehörten Medikamente gegen Grippe und HIV.
Professor Adrian Mulholland vom Centre for Computational Chemistry der Universität Bristol und Mitleiter der Arbeit, sagte: "Viele Medikamente wirken, indem sie an Proteine binden und diese in ihrer Wirkung stoppen. Durch die Bindung an ein bestimmtes Virusprotein kann ein Medikament beispielsweise die Vermehrung des Virus stoppen.
"Um gut zu binden, muss ein kleinmolekularer Wirkstoff gut in das Protein passen. Ein wichtiger Teil der Arzneimittelentwicklung besteht darin, kleine Moleküle zu finden, die sich eng an bestimmte Proteine binden, und zu verstehen, was diese enge Bindung ausmacht, was zur Entwicklung besserer Medikamente beiträgt.
"Um neue Therapien zu entwickeln, müssen die Forscher verstehen, wie die Moleküle der Medikamente in ihre biologischen Ziele passen. Dazu verwenden wir VR, um sie als vollständig dreidimensionale Objekte darzustellen. Die Anwender können dann ein Medikament in das 'Schlüsselloch' einer Proteinbindungsstelle einpassen, um zu entdecken, wie sie zusammenpassen.
In der Studie wurde den Anwendern die Aufgabe gestellt, Medikamente an Protein-Targets wie die Influenza-Neuraminidase und die HIV-Protease zu binden.
Tests zeigten, dass die Anwender in der Lage waren, korrekt vorherzusagen, wie die Medikamente an ihre Proteintargets binden. Indem sie das Medikament in das Protein hineinziehen, konnten sie Strukturen aufbauen, die den Strukturen der aus Experimenten gefundenen Drogenkomplexe sehr ähnlich sind.
Selbst Nicht-Experten konnten Medikamente effektiv an die Proteine andocken. Dies zeigt, dass die interaktive VR dazu verwendet werden kann, genau vorherzusagen, wie neue potenzielle Medikamente an ihre Targets binden.
Die Studie zeigt, wie VR beim strukturbasierten Arzneimitteldesign auch von Nicht-Experten effektiv eingesetzt werden kann. Sie verwendet eine leicht verfügbare VR-Ausrüstung und ein Open-Source-Software-Framework, so dass sie von jedermann angewendet werden kann.
Professor Mulholland fügte hinzu: "Ein wichtiger Aspekt der Arbeit ist, dass die Drogen und ihre Protein-Targets völlig flexibel sind: Wir modellieren ihre strukturellen Veränderungen und ihre Dynamik, und die Benutzer können sie interaktiv manipulieren, um herauszufinden, wie die Drogen mit ihren biologischen Targets interagieren. Dies ist eine wirklich aufregende und leistungsfähige Art und Weise, die Bindung von Medikamenten zu modellieren. Wir haben in dieser Arbeit gezeigt, dass sie genaue Ergebnisse liefert. Diese Instrumente werden bei der Konzeption und Entwicklung neuer Medikamente nützlich sein.
Dr. David Glowacki, Senior Research Fellow der Royal Society an der School of Chemistry und der Fakultät für Informatik in Bristol, sagte: "Unsere Ergebnisse zeigen, dass es möglich ist, Medikamente von Protein-Targets auf einer Simulationszeitskala zu lösen und wieder zu binden, die deutlich kürzer ist als die Zeitskala ähnlicher Ereignisse, die mit Hilfe nicht-interaktiver Molekulardynamik-Motoren beobachtet werden.
"Es ist auch wichtig zu beachten, dass die mit iMD-VR erzeugten vollständigen Entbindungs- und Wiederbindungsereignisse von den Benutzern in weniger als fünf Minuten Echtzeit erreicht wurden.
"Wo Nicht-Experten Spuren von Atomen hatten, die ihnen die richtige Position zeigten, konnten alle Teilnehmer eine Andockposition festlegen, die nahe genug an der Ausgangsstruktur lag, um wissenschaftlich als neu angedockt betrachtet werden zu können.
"Wo keine Spurenelemente vorhanden waren, waren die Bindungspositionen verständlicherweise variabler, aber die Anwender konnten sich trotzdem in allen drei Systemen in den gleichen Bereich der akzeptierten Bindungsposition bewegen. Diese Ergebnisse wurden innerhalb einer einzigen einstündigen Schulungssitzung mit jedem Teilnehmer erreicht, was die Nutzbarkeit dieses VR-Rahmens demonstriert."
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.
Originalveröffentlichung
H. Deeds, R. Walters, S. Hare, M. O’Connor, A. Mulholland and D. Glowacki; "Interactive molecular dynamics in virtual reality for accurate flexible protein-ligand docking"; PLOS One

























































