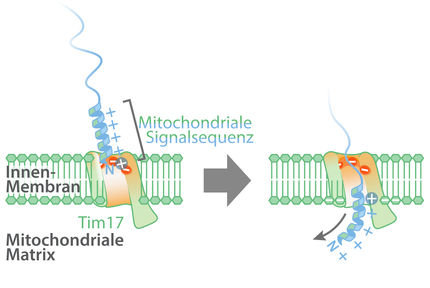
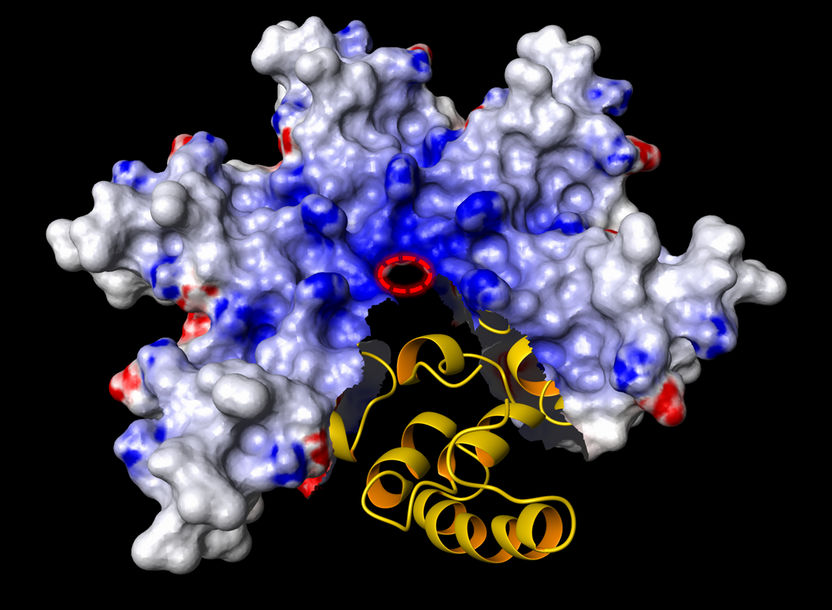
Mitochondrien - Spezialschleuse für Sperrgut
Bereits in ihre 3D-Struktur gefaltete Proteine sind gewissermaßen Sperrgut in der Zelle. LMU-Wissenschaftler haben erstmals die Struktur eines Transportsystems für solche Proteine aufgeklärt und zeigen: In Mitochondrien ähnelt es einer Luftschleuse.
Viele Proteine mit wichtigen Funktionen müssen innerhalb der Zelle durch Membranen geschleust werden, um ihren Einsatzort zu erreichen. In der Regel erfolgt der Transport, solange die Proteine noch nicht in ihre dreidimensionale Struktur gefaltet sind, da dann relativ kleine Poren ausreichen. Müssen gefaltete Proteine transportiert werden, gestaltet sich das schwieriger, da dann ein größeres Loch in der Membran geöffnet werden muss. Vor allem Bakterien und Chloroplasten besitzen hierfür eine besondere Schleuse. In Mitochondrien allerdings ist diese Schleuse verlorengegangen – trotzdem muss ein wichtiges Protein gefaltet durch ihre innere Membran gelangen. Wissenschaftler um Professor Roland Beckmann vom Genzentrum der LMU haben nun den zugehörigen Mechanismus identifiziert und zeigen: Er funktioniert ähnlich wie eine Luftschleuse.
Das sogenannte Rieskeprotein (Rip1) ist ein wichtiger Baustein der Atmungskette, mit der Mitochondrien die universelle Energiewährung der Zelle – das ATP – produzieren. Der Vorläufer dieses Proteins wird aus dem Zellinneren ungefaltet durch die Doppelmembran des Mitochondriums in dessen Innenraum transportiert. Dort wird ein kugelförmiger Proteinabschnitt, eine sogenannte Eisen-Schwefel-Cluster-Domäne, in eine 3D-Struktur gefaltet, die anschließend wieder durch die innere Membran geschleust werden muss.
„Aus früheren Arbeiten unserer Kooperationspartner Walter Neupert und Nikola Wagener war bereits bekannt, dass das sogenannte Bcs1-Enzym an diesem Transport beteiligt ist“, sagt Lukas Kater, der Erstautor der Arbeit. „Nun haben wir im Kryoelektronenmikroskop die 3D-Struktur dieses Enzyms bestimmt – und damit die erste hochaufgelöste Struktur eines eukaryotischen Translokationssystems für gefaltete Proteine überhaupt aufgeklärt.“ Mithilfe dieser Strukturanalysen untersuchten die Wissenschaftler, wie das Enzym es schafft, selektiv den gefalteten Proteinabschnitt durch die Membran zu schleusen, ohne dass dabei größere Mengen anderer Moleküle oder Ionen „mitrutschen“.
Zur Überraschung der Wissenschaftler zeigte sich, dass Bcs1 ein Heptamer ist, also aus sieben Bausteinen besteht, die eine ringförmige Pore bilden. Dadurch kann das Enzym eine deutlich größere zentrale Pore bilden als vergleichbare Enzyme, die nur aus sechs Bausteinen bestehen. Zudem hat Bcs1 eine einzigartige Struktur: Es besitzt zwei Kammern, von denen die eine im Inneren des Mitochondriums liegt und die andere in der inneren Membran. Zwischen den beiden Kammern bilden bestimmte Enzym-Domänen einen Verschluss, der sich vorübergehend öffnen kann, wie die Wissenschaftler zeigten.
„Deshalb schlagen wir einen Mechanismus vor, der dem einer Luftschleuse ähnelt“, sagt Beckmann. Das Rieskeprotein gelangt dabei zuerst durch die große zentrale Pore in die Matrixkammer. Dann kommt es zu einer Konformationsänderung von Bcs1, durch die sich der Verschluss zwischen den beiden Kammern öffnet. Jetzt gelangt das Rieskeprotein in die eigentliche Schleusenkammer, also in die Kammer in der inneren Membran. Im nächsten Schritt schließt sich der Verschluss zwischen beiden Kammern wieder. „Wir vermuten, dass sich anschließend in der Schleusenkammer ein Durchgang öffnet, durch den die gefaltete Eisen-Schwefel-Cluster-Domäne dann in den Zwischenraum der mitochondrialen Doppelmembran gelangt“, sagt Kater.
Mit ihren Ergebnissen haben die Wissenschaftler nicht nur erste Einblicke in die Funktionsweise des Bcs1-Proteins erhalten, es gibt auch eine Reihe von Stoffwechselerkrankungen, die mit dem Protein zusammenhängen, beispielsweise das GRACILE-Syndrom, das zu einem frühen Tod führt, sowie das Björnstad-Syndrom. Deshalb könnte nach Ansicht der Wissenschaftler die Struktur des Proteins verstehen helfen, wie die zugrundeliegenden Mutationen diese Krankheitsbilder verursachen.
Originalveröffentlichung
Lukas Kater, Nikola Wagener, Otto Berninghausen, Thomas Becker, Walter Neupert, Roland Beckmann; "Structure of the Bcs1 AAA-ATPase suggests an airlock-like translocation 3 mechanism for folded proteins"; Nature Structural and Molecular Biology 2020