KI-gesteuerte Klassifizierung einzelner Blutzellen
Neue Methode unterstützt Ärzte bei der Leukämiediagnostik
Erstmals zeigen Forscher des Helmholtz Zentrums München und des Klinikums der Ludwig-Maximilians-Universität München (LMU), dass Deep-Learning-Algorithmen bei der Klassifizierung von Blutproben von Patienten mit akuter myeloischer Leukämie (AML) eine vergleichbare Leistung erbringen wie Fachleute. Die Proof-of-Concept-Studie des Teams ebnet den Weg für eine automatisierte, standardisierte und allgemein verfügbare Analyse von Proben in naher Zukunft.
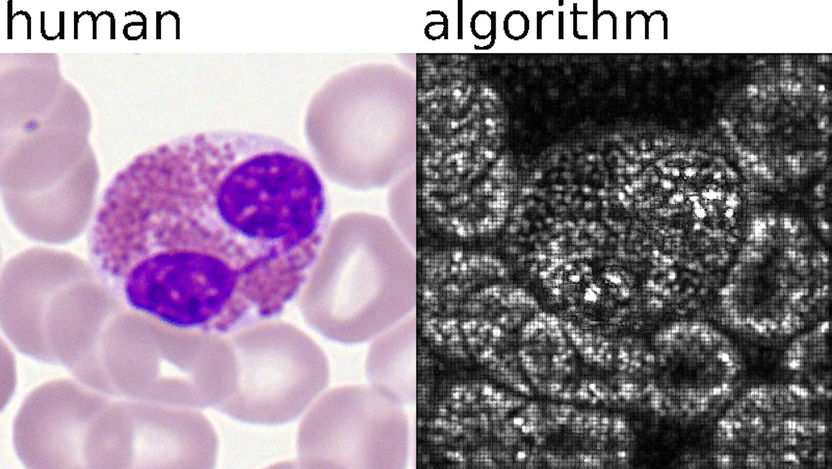
Deep-Learning-Algorithmen der KI analysieren Proben auf automatisierte und standardisierte Weise. Links: Klassifizierung menschlicher Experten. Rechts: Wichtige Pixel für die KI-Analyse.
© Helmholtz Zentrum München / Carsten Marr
Zur Diagnose von Krankheiten werden in medizinischen Labors und Kliniken täglich Millionen einzelner Blutzellen ausgewertet. Der Großteil dieser repetitiven Auswertungen wird noch immer manuell vorgenommen. Ausgebildete Zytologen untersuchen die Zellen in gefärbten Blutausstrichen und teilen diese in etwa 15 verschiedene Kategorien ein. Dieser Klassifizierungsprozess ist anfällig für Qualitätsschwankungen und erfordert die Anwesenheit und das Fachwissen eines Experten.
Um einzelne Blutzellen effizienter auswerten zu können entwickelte ein Team aus Forschern des Helmholtz Zentrums München und des Klinikums der LMU zu diesem Zweck ein neuronales Deep-Learning-Netzwerk. Dieses wurde mit fast 20.000 Einzelbildern trainiert, um Zellen selbstständig klassifizieren zu können. Das Forschungsteam rund um Leiter Dr. Carsten Marr und den Medizindoktoranden Dr. Christian Matek vom Institut für Computational Biology am Helmholtz Zentrum München sowie Prof. Dr. Karsten Spiekermann und Simone Schwarz von der Medizinischen Klinik und Poliklinik III des Klinikums der LMU nutzte dazu Bilder, die aus Blutausstrichen von 100 Patienten mit der aggressiven Blutkrankheit AML und 100 Kontrollen extrahiert wurden. Diese neue, automatisierte Lösung wurde anschließend bewertet, indem ihre Genauigkeit mit der von menschlichen Experten verglichen wurde. Das Ergebnis zeigte, dass das von künstlicher Intelligenz (KI)-gesteuerte Verfahren diagnostisch relevante Blasten (Vorläuferzellen der weißen Blutkörperchen, die normalerweise nur im Knochenmark zu finden sind) mindestens so gut identifiziert wie ein ausgebildeter Zytologe.
Angewandte Forschung durch KI und Big Data
Deep-Learning-Algorithmen für die Bildverarbeitung benötigen zweierlei: eine geeignete neuronale Netzwerkarchitektur mit tausenden von Parametern, sowie eine ausreichend große Menge an Trainingsdaten. Bislang lag kein großer digitalisierter Datensatz von Blutzellen vor, obwohl diese Proben in Kliniken tagtäglich verwendet werden. Die Forschungsgruppe am Helmholtz Zentrum München hat nun den ersten großen Datensatz dieser Art zur Verfügung gestellt. Derzeit arbeiten Marr und sein Team eng mit der Medizinischen Klinik und Poliklinik III des Klinikums der LMU und einem der größten europäischen Leukämie-Labore, dem Münchner Leukämie-Labor (MLL), zusammen, um Hunderte von Blutausstrichen von Patienten zu digitalisieren.
„Um unseren Lösungsansatz in der Praxis umzusetzen, muss die Digitalisierung von Blutausstrichen von Patienten zur Routine werden. Algorithmen müssen mit Proben aus verschiedenen Quellen trainiert werden, um die inhärente Heterogenität bei der Aufbereitung und Färbung der Proben zu lernen“, erklärt Marr. „In unserer Publikation konnten wir beweisen, dass Deep-Learning-Algorithmen eine ähnliche Leistung erzielen können wie Zytologen. In einem nächsten Schritt werden wir untersuchen, wie gut andere Krankheitsbilder, beispielweise genetische Mutationen oder Translokationen, mit dieser neuen KI-gesteuerten Methode vorhergesagt werden können.“
Diese Arbeit ist ein Beispiel dafür, welches Potenzial dem Einsatz künstlicher Intelligenz in der angewandten Forschung zukommt. Sie entstand aus einer Fortführung der Forschungsarbeiten des Helmholtz Zentrums München zur Klassifizierung einzelner Zellen in Blutstammzellen (Buggenthin et al., Nature Methods, 2017), die 2018 mit dem Erwin-Schrödinger-Preis der Helmholtz-Gemeinschaft ausgezeichnet wurden. Unterstützt wurde die Studie vom SFB 1243 der Deutschen Forschungsgemeinschaft (DFG) sowie durch ein Promotionsstipendium der Deutschen José-Carreras-Leukämie-Stiftung an Dr. Christian Matek.