Neue Einblicke in die Evolution von Proteinen
Wie schaffen es Bakterien, sich an synthetische Umweltgifte anzupassen und zum Beispiel in weniger als 70 Jahren ein Pestizid und chemischen Kampfstoff als Nahrung zu erschließen? Die evolutionären Anpassungen, die solchen Prozessen zugrunde liegen, hat jetzt ein internationales Forscherteam im Detail untersucht. Die Wissenschaftler betrachteten ein Enzym, das sie aus Bakterien isoliert hatten, die in der Nähe von Fabriken Pestiziden ausgesetzt gewesen waren. Mithilfe einer neuartigen Kombination von Methoden fanden sie heraus, welche Mutationen im Laufe der Jahre im Erbgut stattgefunden hatten – und wie diese Mutationen zu biochemischen Veränderungen führten, die heute das Enzym dazu befähigen, das Pestizid zu spalten.
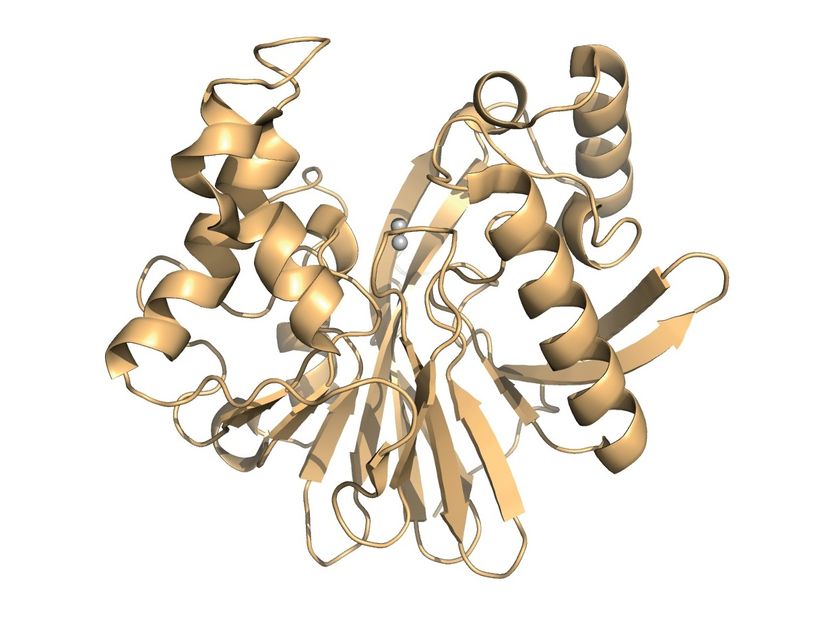
Modell des Enzyms, das die Forscher in ihrer Studie untersuchten. Die zwei grauen Kugeln stellen das aktive Zentrum dar, das sich mit dem Pestizid verbindet, um es zu spalten.
G. Yang et al/Nat Chem Biol
„Mit unserer Studie zeigen wir, dass die Technik der sogenannten ancestralen Rekonstruktion nicht nur genutzt werden kann, um Evolutionsvorgänge zu entschlüsseln, die viele Jahrhunderte zurückliegen, sondern auch, um erst kürzlich stattgefundene Anpassungen mit sehr rascher Evolution, wie sie in Bakterien stattfindet, zu untersuchen“, betont Co-Autor Prof. Dr. Erich Bornberg-Bauer von der Westfälischen Wilhelms-Universität Münster (WWU), dessen Gruppe maßgeblich an der Studie beteiligt war. Sie fand in Kooperation mit der Forschergruppe um Prof. Dr. Nobuhiko Tokuriki von der British Columbia University im kanadischen Vancouver statt. Die Ergebnisse könnten unter anderem dabei helfen, neue Wege für den Abbau und die Entsorgung von Umweltgiften und chemischen Stoffen zu finden.
Hintergrund und Methode
Die Forscher nutzten die Methode der ancestralen Rekonstruktion, bei der mithilfe von Algorithmen nachverfolgt wird, wie Proteine in Vorfahren ausgesehen haben. Die Wissenschaftler „beleben“ dazu eine Art Ur-Gen wieder, das als Ausgangspunkt einer heutigen Genfamilie gilt. Klassischerweise wird diese Methode angewandt, um Proteine, die sich langsam verändern, über mehrere Millionen bis Milliarden Jahre zurückgehend nachzustellen. In diesem Fall nutzten die Forscher die Methode, um ein sich sehr schnell entwickelndes Enzym über einige Jahrzehnte zurückzuverfolgen und molekulare Mechanismen aufzudecken, die zu einer neuen Funktion geführt haben – der Fähigkeit, das Pestizid Methyl-Parathion zu spalten. Dieses wird erst seit rund 70 Jahren industriell erzeugt und ist eng verwandt zu Nervengiften, die auch für die chemische Kriegsführung in Frage kommen. Die Forscher kombinierten die Methode der ancestralen Rekonstruktion mit biochemischen und strukturellen Tests des Vorgänger-Proteins im Labor und identifizierten darin fünf Mutationen, die dafür hauptverantwortlich sind, dass das Protein im Laufe der Zeit seine Funktion ändern konnte.
In einem weiteren Schritt untersuchten die Wissenschaftler, inwiefern diese Mutationen jeweils die sogenannte Fitness, also den Grad der Anpassung der Gene zu bestimmten Zeitpunkten, veränderten. Zudem charakterisierten sie viele verschiedene Kombinationen der Mutationen. Sie setzten neuartige statistische Analysen ein, mit denen sie die Interaktion der Gene und deren unterschiedliche Dominanz erheben konnten. Diese Analysen gaben Aufschluss über die zugrundeliegenden molekularen Interaktionen – sowohl innerhalb des Enzyms als auch zwischen Enzym und Pestizid.
Die Forscher fanden heraus, dass die fünf wichtigsten der identifizierten Mutationen in einem komplexen Netzwerk zusammenspielen und letztendlich gemeinsam beeinflussen, dass sich das sogenannte aktive Zentrum des Enzyms besser mit dem Pestizid verbinden und so neue Funktionen ausbilden kann. „Unser kombiniertes Verfahren kann auch in vielen anderen Fällen eingesetzt werden, um mehr über die molekularen Mechanismen herauszufinden, die der Evolution von Proteinen zugrunde liegen“, sagt Co-Autor Elias Dohmen von der WWU, der im Rahmen seiner Doktorarbeit in der Arbeitsgruppe Molekulare Evolution und Bioinformatik die Rekonstruktion der ancestralen Proteinsequenzen für die Studie durchführte.
In zukünftigen Untersuchungen wollen die Wissenschaftler weitere Proteine mit anderen Funktionen überprüfen, um allgemein verwendbare Regeln aus der Methodik ableiten zu können. Darüber hinaus wollen sie noch mehr Sequenzdaten sammeln, um weitere und bessere ancestrale Rekonstruktionen erstellen zu können.
Das neu gewonnene Wissen ist unter anderem für die sogenannte gerichtete Evolution relevant – ein Verfahren, das die Optimierung von Enzymen und Proteinen zum Ziel hat und vor allem in der industriellen Biotechnologie Anwendung findet. Hierfür gab es 2018 den Chemie-Nobelpreis an Frances Arnold vom California Institute of Technology.
Neben der British Columbia University in Vancouver und der Universität Münster wirkten die Universitäten Canberra in Australien und Uppsala in Schweden an der Studie mit.