KI zur Vorhersage von Proteinstruktur entwickelt
Proteine sind Hochleistungsbiomaschinen: Eiweiße finden sich in jeder Zelle und spielen im menschlichen Körper eine wichtige Rolle, etwa bei der Blutgerinnung oder als Hauptbestandteil von Haaren oder Muskeln. Welche Funktion die molekularen Werkzeuge jeweils erfüllen, lässt sich an ihrer Gestalt erkennen. Forscher des Karlsruher Instituts für Technologie (KIT) haben eine neue Methode entwickelt, diese Proteinstruktur mittels Künstlicher Intelligenz vorherzusagen.
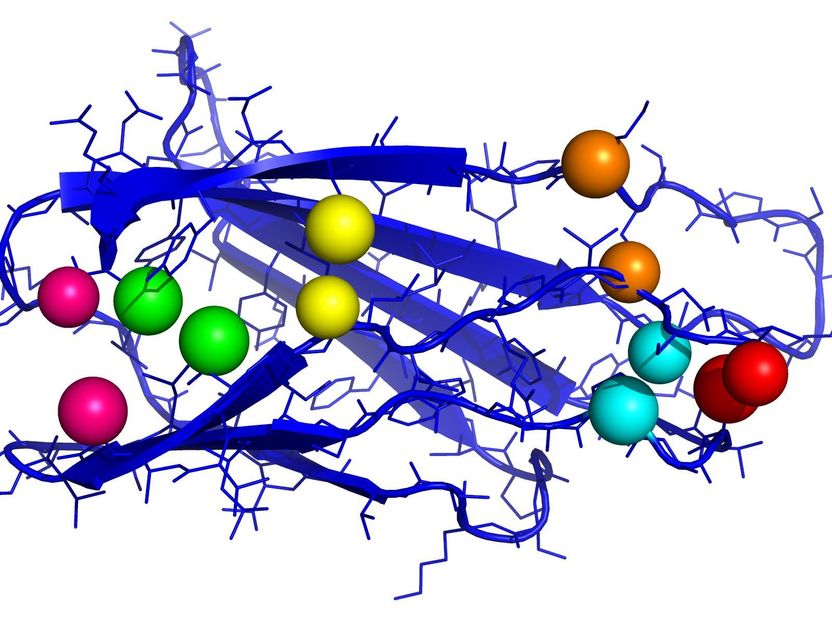
Fibronektin spielt bei der Wundheilung eine bedeutende Rolle. Die Grafik zeigt einen wichtigen Teil des Proteins mit Kontaktpaaren (jeweils gleichfarbige Kugeln).
Ines Reinartz, KIT
Proteine können mit anderen Molekülen durch Eindringen oder Umschließen interagieren, je nach Form. Diese lässt sich – anders als beim Blick in den heimischen Werkzeugkasten – nicht ohne weiteres feststellen, sondern nur mit teuren und aufwendigen Experimenten. Forscher des Steinbuch Centre for Computing (SCC), dem Rechenzentrum des KIT, haben dafür zunächst Datenbanken für Proteinsequenzen durchkämmt und gleiche Proteine unterschiedlicher Spezies verglichen. „Hämoglobin, das in unserem Körper für den Sauerstofftransport zuständig ist, gibt es auch beim Insekt, bei der Feldmaus und beim Schimpansen“, erklärt Markus Götz, Datenanalyst beim SCC. Den Aufbau eines Proteins kann man sich dabei wie eine Perlenkette vorstellen, an der Proteinteile, die Aminosäuren, aufgereiht sind. Seine dreidimensionale Struktur – und damit seine Eigenschaften – erhält es, indem sich manche weit voneinander entfernte „Perlen“ zu Paaren zusammenschließen und das Protein so falten. Bei verschiedenen Organismen können sich diese Paarungen unterscheiden, die Eigenschaften des Proteins bleiben aber dennoch gleich. „Schädliche Mutationen wurden im Laufe der Evolution ausgefiltert“, sagt Götz.
Das Team um Götz hat nun einer Künstlichen Intelligenz (KI) beigebracht, welche Kopplungen in bekannten Proteinsequenzen evolutionär erfolgreich waren. „Wir erwarten, dass das System so auch Rückschlüsse auf den Aufbau unbekannter Proteinsequenzen ziehen kann“, so Götz. Der Nutzen: „Es ist sehr einfach zu bestimmen, aus welchen Aminosäuren eine Proteinkette besteht. Proteinstrukturen direkt experimentell zu bestimmen, ist aber sehr aufwendig und kostet Millionen“, ergänzt Alexander Schug vom SCC.
Der Ansatz, Kontakte in Proteinen von einer KI vorhersagen zu lassen, ist nicht ganz neu. „Aktuell werden dafür vor allem Methoden aus der Bildverarbeitung eingesetzt“, sagt Götz. Solche Neuronalen Netzwerke könnten Muster gut erkennen. Bei der Proteinstruktur spielten aber besonders jene Kontakte von Proteinteilen eine Rolle, die besonders weit auseinander liegen, weil sie beim Falten einen stärkeren Einfluss auf die Form haben als solche, die nahe beieinander liegen. „Daher verfolgen wir stattdessen einen Ansatz aus der automatisierten Sprachübersetzung. Wir betrachten die Aminosäureketten als Sätze, die in eine andere Sprache übersetzt werden.“ Sogenannte „Self-Attention Neural Networks“ kommen in populären Übersetzungsprogrammen zum Einsatz. Sie können erkennen, welche Teile des Satzes miteinander in Beziehung stehen oder – im Proteinkontext – welche Aminosäuren miteinander einen Kontakt bilden.
Meistgelesene News
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

Antibody Stabilizer von CANDOR Bioscience
Protein- und Antikörperstabilisierung leicht gemacht
Langzeitlagerung ohne Einfrieren – Einfache Anwendung, zuverlässiger Schutz

DynaPro NanoStar II von Wyatt Technology
NanoStar II: DLS und SLS mit Touch-Bedienung
Größe, Partikelkonzentration und mehr für Proteine, Viren und andere Biomoleküle

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.