Schnappschüsse der Grippevirus-Replikationsmaschine in Aktion
Forscher des EMBL Grenoble haben erstmals verschiedene Funktionszustände der Grippevirus-Polymerase beobachtet, da sie aktiv transkribiert. Diese in Nature Structural & Molecular Biology veröffentlichten Ergebnisse liefern wertvolle Informationen für die nächste Generation von Grippemittel.
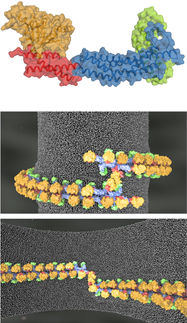
Influenza-Polymerase (hellcyan) bei der Synthese von Messenger-RNA (schieferblau) durch Kopieren der genomischen viralen RNA-Vorlage (gelb).
Stephen Cusack / EMBL
Wenn ein Virus eine Wirtszelle infiziert und in sie eindringt, wird das genomische Material im Virus sowohl repliziert, um mehrere Kopien von sich selbst zu produzieren, als auch in virale Messenger-RNA (mRNA) transkribiert. Die virale mRNA kann von der Proteinproduktionsmaschine der Wirtszelle gelesen werden, wodurch sie dazu gebracht wird, virale Proteine herzustellen. Die viralen Proteine verpacken die Kopien des viralen Genoms zu Nachkommenviren, die aus der Zelle freigesetzt werden, um neue Wirte zu infizieren. Ein Enzym namens Polymerase ist sowohl für die Transkription als auch für die Replikation des viralen Genoms verantwortlich und damit entscheidend für die erfolgreiche Vermehrung des Virus. Das Grippevirus, bei dem das genomische Material nicht aus DNA, sondern aus RNA besteht, bildet keine Ausnahme von diesem Modus operandi.
"Die Erforschung und das Verständnis der einzigartigen Mechanismen der Transkription und Replikation, die das Influenzavirus nutzt, ist unerlässlich, um seine Ausbreitung zu bekämpfen", erklärt EMBL-Gruppenleiter Stephen Cusack.
Eine jahrzehntelange Studie
Stephen Cusack und seine Forschungsgruppe am EMBL Grenoble haben vor mehr als 20 Jahren mit der Arbeit an der Influenza-Polymerase begonnen. Im Jahr 2014 veröffentlichte die Gruppe die ersten Kristallstrukturen der kompletten Polymerase-Maschine. Versuche, die verschiedenen Zustände der aktiv transkribierenden Grippepolymerase strukturell zu charakterisieren, waren jedoch bisher erfolglos.
"Alle bisherigen Studien haben sich mit der Ruhestruktur der Polymerase-Maschine beschäftigt; wir haben noch nie beobachtet, dass sie tatsächlich etwas bewirkt", sagt Tomas Kouba, Postdoc in der Gruppe, der einen Großteil der Arbeit geleistet hat.
Mit Hilfe der Röntgenkristallographie und der Kryoelektronenmikroskopie, die beide auf modernsten Geräten der European Synchrotron Radiation Facility in Grenoble durchgeführt wurden, konnte die Gruppe erstmals Atomauflösungsstrukturen verschiedener Funktionszustände der Influenza-Polymerase bestimmen, die genomische RNA aktiv in mRNA umwandelt. Insbesondere bietet es die erste Charakterisierung der Bewegungen der Polymerase während des so genannten Nukleotid-Additionszyklus, bei dem jedes nachfolgende Nukleotid der wachsenden mRNA-Kette hinzugefügt wird.
Zielpunkt für neue Medikamente
Trotz seiner Vertrautheit ist das Grippevirus - einige Stämme, von denen einige zu den zehn gefährlichsten Viren der Welt gehören - noch lange nicht gut verstanden. Laut der Weltgesundheitsorganisation sterben jedes Jahr bis zu 500.000 Menschen weltweit an Grippe, mit potenziell viel höheren Sterblichkeitsraten, wenn ein neuer pandemischer Grippevirus auftritt. Impfungen sind nicht immer wirksam und es werden Grippemittel als ergänzende Behandlungsmöglichkeit benötigt.
Da die virale Polymerase für die Vermehrung des Grippevirus unerlässlich ist, ist sie ein Hauptziel für die Entwicklung neuer Grippemittel. Die Ergebnisse der EMBL-Gruppe liefern neue Erkenntnisse für die Entwicklung der nächsten Generation von Grippemittel. Der Vorteil von Medikamenten, die die Funktion der Polymerase stoppen, ist, dass es viel unwahrscheinlicher ist, dass das Virus in einer Weise mutiert, die das Medikament nutzlos machen würde.
"Unsere Ergebnisse liefern die ersten Momentaufnahmen eines Films, der den kompletten Transkriptionszyklus der Grippepolymerase von der Initiierung bis zur Beendigung zeigt", erklärt Cusack. Die Gruppe wird weiterhin die leistungsstarke Technik der Kryo-Elektronenmikroskopie nutzen, um die fehlenden Schritte auszufüllen.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.