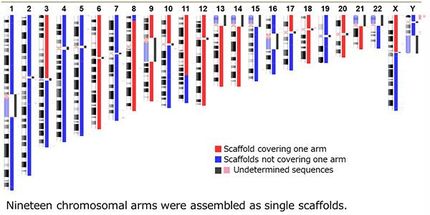
“Vertebrate Genomes Project” veröffentlicht neue Genome
Das internationale „Vertebrate Genomes Project“ (VGP) ist offiziell gestartet und hat 15 neue Referenzgenome veröffentlicht, die alle fünf Klassen von Wirbeltieren repräsentieren: Säugetiere, Vögel, Reptilien, Amphibien und Fische. Diese 15 Genome sind die bisher vollständigsten Genome dieser Spezies. Ziel des VGP ist es, qualitativ hochwertige, nahezu fehlerfreie und vollständige Genome aller 66.000 Wirbeltierarten der Erde zu erstellen. Die Daten dazu werden derzeit hauptsächlich von Teams an drei internationalen Sequenzierungszentren erstellt: der Rockefeller University, USA, dem Wellcome Sanger Institute, Großbritannien, und dem Max-Planck-Institut für molekulare Zellbiologie und Genetik in Dresden. Die Genome einer Fledermaus und eines Fisches wurden in Dresden sequenziert.

Spix's Scheibenflügel-Fledermaus (Thyroptera tricolor) – eine der Wirbeltierarten, deren Erbgut Wissenschaftler hochpräzise entschlüsseln wollen.
© S. Puechmaille
Das ehrgeizige Ziel des VGP ist es, grundlegende Fragen der Biologie, des Artenschutzes und von Krankheiten zu beantworten. Dazu gehört die Bestimmung von Arten, die am ehesten vom Aussterben bedroht sind, und die Erhaltung ihrer genetischen Informationen für zukünftige Generationen. Die hochqualitativen VGP-Genome sollen in Zukunft zu den wichtigsten Referenzquellen für diese Spezies werden. In einer öffentlich zugänglichen, digitalen Bibliothek für Genome, der „Genom-Arche“, werden diese dann gespeichert.
Während der aktuellen Phase 1 des Projektes, werden Referenz-Genome ausgewählter Arten erstellt, die alle Wirbeltierordnungen repräsentieren, die sich seit dem letzten Massenaussterben der Dinosaurier vor 66 Millionen Jahren entwickelt haben. Die Untersuchung dieser Arten auf Ebene der Ordnungen hilft den Wissenschaftlern herauszufinden, welche Arten dieses Aussterben überlebt haben. Diese Studien können zudem Erkenntnisse liefern, wie Arten das gegenwärtige Massenaussterben überleben könnten. Das Aufspüren genetischer Variationen kann dazu beitragen, diese Arten vor dem Aussterben zu bewahren. Zu den 15 neuen Genomen gehören stark gefährdete Arten wie das Schnabeltier und der Kakapo-Papagei. Andere Arten sind der Zebrafink und der Annakolibri, der - wie der Papagei - zu den einzigen drei unter über 40 Vogelordnungen gehört, die Gesang erlernen können. Zwei Fledermausarten, die ebenfalls Laute erlernen können, sind außerdem Teil dieser ersten Genome.
Zur Durchführung des Projekts hat die Dachorganisation Genome10K, aus der das Projekt hervorgegangen ist, über 150 Experten aus Wissenschaft, Industrie und Regierung aus 12 Ländern zusammengebracht, um hochauflösende Sequenzierungsmethoden zu entwickeln, die sowohl Kosten senken als auch die Fehler beseitigen, unter der die aktuellen Referenzgenome leiden. Viele Referenzgenome sind mit Fehlern durchsetzt - Teile von Genen fehlen, einige sind falsch zusammengesetzt, andere Gene fehlen komplett. Folglich arbeiten die Wissenschaftler teilweise mit falschen Gensequenzen und Strukturen, die ihre Forschung behindern. Die neuen VGP-Genome beseitigen die meisten dieser Fehler.
Erbgutanalyse von Fledermäusen und Fischen
Die Forscher am Max-Planck-Institut für molekulare Zellbiologie und Genetik und insbesondere die Bioinformatiker am Zentrum für Systembiologie Dresden (CSBD) sequenzieren in der ersten Phase des Projekts schwerpunktmäßig Fledermäuse und Fische. Die Dresdner Wissenschaftler sind Teil des DRESDEN-concept Genome Center (DCGC) und verfügen über besondere Expertise beim Einsatz verschiedener „long-read“ Sequenzierungstechnologien. Längere Sequenzstücke sind wichtig, da diese die sich wiederholenden Teile im Genom überspannen können und damit eine eindeutige Zuordnung erlauben.
Meistgelesene News
Organisationen
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.