Genregulation schlägt auf den Magen
Max-Planck-Forschern gelingt mit neuer Technik Erfolg im Kampf gegen den Verursacher von Magengeschwüren und Magenkrebs
Ein Durchbruch in der Entschlüsselung der Genregulation von Helicobacter pylori ist einem internationalen Forscherteam um Jörg Vogel vom Max-Planck-Institut für Infektionsbiologie in Berlin gelungen. Mit Hilfe einer neu entwickelten Methode fanden sie im Genom des Bakteriums erstmals hunderte kleine Ribonukleinsäuren - kleine RNA-Partikel, die die Gene des Erregers regulieren. Diese Erkenntnisse sollen den Wissenschaftlern auch bei der Entwicklung neuer Impfstoffe helfen.
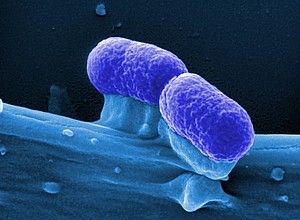
Helicobacter pylori (blau) auf Zellen der Magenschleimhaut (orange)
Brinkmann
Etwa die Hälfte der Weltbevölkerung ist mit Helicobacter pylori infiziert, 30 Prozent der deutschen Bevölkerung tragen den gefährlichen Keim. Das Bakterium beeinflusst neben Krebs auch andere chronische Erkrankungen, unter anderem des Herz-Kreislauf-Systems. Das Genom von Helicobacter pylori wurde bereits 1997 entschlüsselt. Demnach beherbergt es erstaunlich wenig regulatorische Gene. Zwei wichtige Fragen blieben jedoch weitgehend unbeantwortet: Wo genau fangen die Gene von Helicobacter an, und sind wirklich schon alle gefunden worden?
Deshalb suchten Wissenschaftler in den letzten Jahren fieberhaft nach neuartigen Regulatoren, wie zum Beispiel den regulatorischen kleinen Ribonukleinsäuren, auch small RNAs (sRNAs) genannt. Es gibt nämlich wesentlich mehr dieser kleinen Partikel als früher angenommen. Sie können Gene regulieren, indem sie beispielsweise an Sequenzen der Erbinformation binden und so deren Übersetzung in ein Protein verhindern.
Bei Helicobacter pylori war diese Suche seit Jahren erfolglos. Jörg Vogel, Leiter der Arbeitsgruppe RNA Biologie am Max-Planck-Institut für Infektionsbiologie und sein Team haben jetzt die sRNAs im Magenbakterium aufgespürt. Sie entwickelten dafür eine neue Methode, die sich der rapide entwickelnden Hochdurchsatzsequenzierung bedient. Diese Technik ermöglicht die gleichzeitige Entzifferung von Millionen von RNA-Sequenzen, die aktuell in einer Zelle produziert werden. Das überraschte Team fand gleich 60 sRNAs: "Bisher galt H. pylori als Organismus ohne sRNAs", erklärt Vogel.
Magenbakterium als Modell für Genregulation
Die Entdeckung ermöglicht unerwartete Einblicke in die Funktionsweise der sRNA-Regulation: "Wir fanden mindestens genauso viele kleine RNAs wie kürzlich in den Darmbakterien Escherichia coli und Salmonellen", so Vogel. Helicobacter pylori fehlt jedoch ein ganz wichtiges Protein, damit solche RNAs die Genexpression regulieren können. Möglicherweise nutzt es andere, bislang unbekannte Signalwege. Damit könnte das Bakterium ein neuer Modellorganismus für die RNA-Forschung werden. "Wir erhoffen uns völlig neue Erkenntnisse, wie Genregulation funktioniert", erläutert Vogel. Dank der neuen Methode konnten die Forscher außerdem für jedes Gen den Startpunkt ausfindig machen. "Wir können das Genom jetzt ganz anders interpretieren", so Vogel.
Mit dem Forschungserfolg, der in Zusammenarbeit mit Wissenschaftlern aus Leipzig und Frankreich gelang, könnte auch die Entwicklung eines Impfstoffes gegen den Magenkeim erleichtert werden. Die neue Sequenzierungstechnik wird von Vogels Team nun auch auf andere, ebenfalls durch Lebensmittel übertragbare Keime angewendet. Der nächste Kandidat ist Campylobacter jejuni, das neben Salmonellen die häufigste Ursache für infektiöse Durchfallerkrankungen ist.
Originalveröffentlichung: Cynthia M. Sharma, Steve Hoffmann, Fabien Darfeuille, Jérémy Reignier, Sven Findeiß, Alexandra Sittka, Sandrine Chabas, Kristin Reiche, Jörg Hackermüller, Richard Reinhardt, Peter F. Stadler & Jörg Vogel; "The primary transcriptome of the major human pathogen Helicobacter pylori"; Nature, 2010 .
Meistgelesene News
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.
Meistgelesene News
Weitere News von unseren anderen Portalen
Zuletzt betrachtete Inhalte
„Molekularer Klebstoff“ – Forschung an einer neuen Waffe gegen Krebstreiber - Wirkmechanismus einer neu entdeckten zellulären Krebsbremse aufgeklärt
Cargo-Verkehr im Zellfortsatz: IFT-Moleküle transportieren Frachtgut an Ort und Stelle - Mainzer Neurobiologen identifizieren und lokalisieren Transportmoleküle in Zellen der Netzhaut
Darmbein

Tosoh erhöht Produktionskapazität für Trenn- und Aufreinigungsmedien
Covid-19: Neue Energie für schlappe Immunzellen

Die Spritze gegen Krebs - Eine Impfung stoppt das Wachstum von Tumoren bei Nashörnern
Karl_Theodor_Ernst_von_Siebold

Zentrale Biobank für die Medikamentenforschung

Akuter Stress verändert Kontrolle der Genaktivität
SmartCal | Zertifizierte Referenzmaterialien | Mettler-Toledo

Müll von Corona-Patienten trennen - Leitlinien der EU-Kommission