Auf dem Weg den Schafscode zu entschlüsseln
Forscher aus Mecklenburg-Vorpommern im internationalen Projektverbund an der Entschlüsselung des Schaf-Erbgutes beteiligt
Nach der erfolgreichen Entschlüsselung des Rindercodes ist das Leibniz-Institut für Nutztierbiologie (FBN) in Dummerstorf an der Erforschung eines weiteren Meilensteins der Tiergenetik beteiligt. Unter der Führung des College of Agriculture, Utah State University, Logan, USA wollen Wissenschaftler aus Australien, Neuseeland, Großbritannien und Dummerstorf im Verbund die DNA-Sequenz des Schafgenoms komplett aufklären.
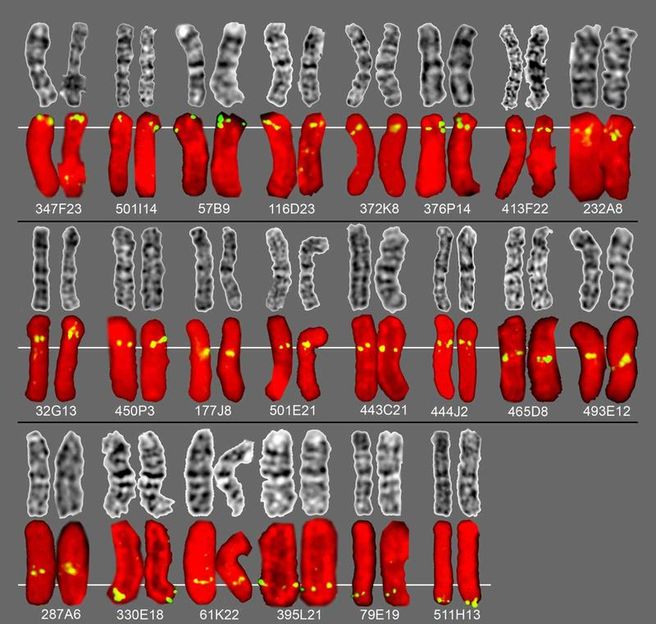
Mit Hilfe von zytogenetischen Chromosomenanalsysen, die das FBN durchführt, können Schafchromosomen identifiziert und die Lage von Genen auf den Chromosomen bestimmt werden.
FBN
Die Arbeitsgruppe um Privatdozent Dr. Tom Goldammer aus dem Forschungsbereich Molekularbiologie ist weltweit eine der wenigen anerkannten Forschergruppen auf dem Gebiet der Genkartierung beim Schaf. Die Kenntnis der genauen Lage von Genen auf den Chromosomen ist eine wichtige Information für die Erstellung der korrekten DNA-Sequenz des Schafgenoms. Das bis Ende 2012 laufende internationale Forschungsprojekt wird vom amerikanischen Landwirtschaftsministerium gefördert.
Dem Schaf kommt im Rahmen der zukünftigen globalen Ernährungssicherung eine besondere Bedeutung zu. Als Wiederkäuer ist es in der Lage, aus vegetativen Pflanzenbestandteilen, die von Nichtwiederkäuern und vom Menschen nicht genutzt werden, Milch und Fleisch zu erzeugen. Weltweit stehen 3,3 Mrd. Hektar Grasland zur Verfügung, die nur von Wiederkäuern genutzt werden können. Darüber hinaus gilt das Schaf aufgrund seiner Rassenvielfalt als ideales Modell zur Gewinnung von Erkenntnissen zu Genomvariationen. Das entschlüsselte Erbgut von Rind und Schaf, soll künftig das Rückgrat für die Analyse der Genstruktur anderer Wiederkäuer, beispielsweise von einer Ziege oder einer Giraffe, bilden. Es wird dann für Wissenschaftler und Forscher frei zugänglich sein.
Um die DNA-Sequenz des Schafgenoms komplett aufklären zu können, müssen die Schafchromosomen zunächst identifiziert und die Lage der Gene auf den Chromosomen bestimmt werden. Die Identifizierung dieser Genorte (Loci) mit Hilfe von zytogentischen Kartierungsmethoden ist Bestandteil der Forschungsarbeit am FBN. Im Ergebnis wird so die Erstellung einer möglichst korrekten Genomsequenz unterstützt. Dass für die Analysen benötigte Probenmaterial wird vom Kooperationspartner in den USA, der Utah State University geliefert. Dabei handelt es sich um DNA-Fragmente, der vor allem in westlichen Nationen in der Zucht verwendeten Schafsrasse Texel.
Durch die vollständige Sequenzierung und Aufklärung der Genstruktur des Schafgenoms wird ein tieferes Verständnis der Biologie und Evolution von Wiederkäuern erwartet. Die Ergebnisse liefern nicht nur die Basis, um das Schafgenom im Detail zu verstehen. Sie bilden auch einen Ansatzpunkt, um neue Strategien für die Zucht aufzuzeigen. So könnten beispielsweise langfristig moderne umweltangepasste Schafstiere entwickelt werden, die mit den lokalen Gegebenheiten einer Region optimal zu Recht kommen und sehr gute Erträge wie Wolle, Fleisch und Milch, liefern. Zudem macht die relativ hohe genetische Ähnlichkeit und damit enge Beziehung zum Menschen, das Schaf zu einem nützlichen Modell für grundsätzliche biomedizinische Fragestellungen.
Meistgelesene News
Organisationen
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.




















































