Una nueva herramienta ayuda a "detectar la diferencia" en la organización del genoma
Esto permite a los científicos rastrear las causas de las enfermedades más rápidamente
La forma en que el ADN está organizado en el núcleo de la célula es muy importante para el desarrollo normal y la función de la célula. Una mutación que afecte al empaquetamiento del material genético en el núcleo de la célula puede conducir a trastornos del desarrollo o enfermedades como el cáncer. Un equipo de científicos del Instituto Max Planck de Münster, el Instituto de Ciencias Médicas de Londres (MRC) y el Centro de Regulación Genómica de Barcelona ha desarrollado un método para el análisis automatizado de la organización del genoma tridimensional en diferentes poblaciones celulares. Esto permite a los científicos rastrear las causas de las enfermedades más rápidamente.
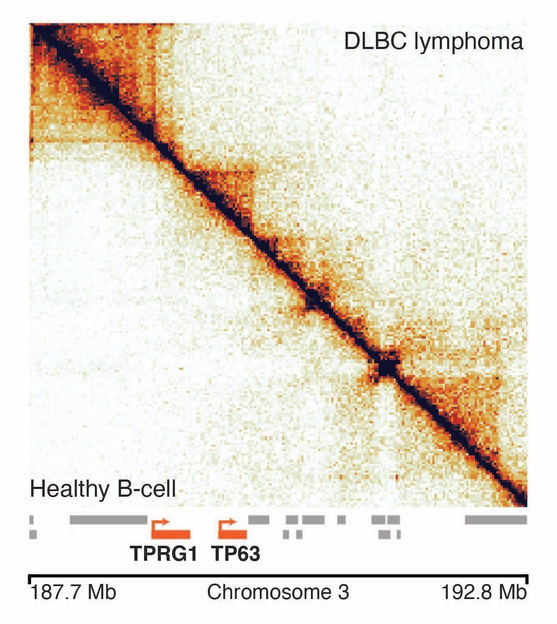
Representación de la estructura del genoma 3D para el cáncer (por encima de la diagonal) y las células B sanas (por debajo de la diagonal). Los genes previamente conocidos relacionados con el cáncer están resaltados en rojo.
Vaquerizas lab / MPI Münster
La mayoría de las células de nuestro cuerpo contienen dos metros de ADN. Toda esta información genética necesita ser empaquetada en el núcleo de la célula, que tiene sólo 0,01 milímetros de ancho. Para comparar, el ancho de un cabello humano es de 0,1 milímetros. Empacar una molécula de ADN en algo con dimensiones diez veces más pequeñas que las de un cabello humano requiere mucha organización.
Dentro del núcleo de la célula, el ADN se organiza en estructuras llamadas cromosomas. Los humanos tienen 23 pares de cromosomas, así que 46 en total. Investigaciones anteriores han demostrado que estas 46 moléculas no se entrelazan dentro del núcleo, como trozos de espagueti en un plato, sino que cada cromosoma tiene su propio espacio.
Entender cómo está estructurado todo este material genético es vital para entender cómo funciona una célula correctamente. Una mutación que afecte al empaquetamiento del ADN puede conducir a condiciones de desarrollo, cánceres, desórdenes neuronales y más. Este conjunto de posibles efectos perjudiciales significa que los investigadores deben ser capaces de controlar cómo se empaqueta el ADN en alta resolución. Esto ha sido posible desde hace unos 10 años, pero hasta ahora la mayoría de los análisis que comparan la organización del genoma en 3D se han llevado a cabo mirando los datos a ojo, o con enfoques demasiado simplificados. Aunque es útil, esto consume mucho tiempo, y podría estar pasando por alto información importante.
La investigación del grupo de Genómica de Desarrollo y Regulación del MRC LMS y del Instituto Max Planck de Biomedicina Molecular de Alemania, publicada el 19 de octubre de 2020 en la revista Nature Genetics, describe una nueva herramienta para ayudar a superar estos problemas. Utilizando una técnica computacional llamada "análisis de similitud estructural", el equipo ha desarrollado un algoritmo que compara los datos de organización del ADN de un individuo sano con los de un paciente con cáncer, por ejemplo. A través del trabajo en colaboración con investigadores del Centro de Regulación del Genoma de Barcelona, el algoritmo puede identificar qué regiones están empaquetadas de forma diferente en el paciente con cáncer, para ayudar a identificar lo que podría haber ido mal.
El algoritmo funciona de manera similar al control de pasaportes en el aeropuerto. La foto de tu pasaporte es la imagen de referencia. La cabina electrónica trata de hacer coincidir esa foto de referencia con tu cara el día que viajas, y si hay suficiente similitud entonces te deja pasar. Aquí, el algoritmo aplica este mismo concepto a los datos del genoma 3D. Utiliza los datos del individuo sano como referencia, y trata de detectar cualquier diferencia en los datos del paciente con cáncer.
Esta herramienta ayuda a los investigadores a escanear toda la longitud del ADN en un proceso automatizado, y mucho más rápido que a simple vista. También permite a los investigadores dar un valor numérico a una región del genoma. Antes, sólo podían decir si una sección determinada del ADN era diferente o no. Ahora, el algoritmo asigna a las regiones del ADN un valor numérico que denota cuán diferente es esa región, por ejemplo, en una persona sana comparada con un paciente con cáncer. La herramienta también puede resaltar las diferencias en las regiones en las que el ojo humano no puede ver ninguna diferencia. También puede ayudar a analizar los datos de un grupo de pacientes mucho más rápidamente.
Juanma Vaquerizas, Jefe del Grupo de Investigación Max Planck del MPI en Münster, Jefe del grupo de Genómica de Desarrollo y Regulación del MRC LMS y autor principal del estudio, discutió las implicaciones para esta investigación: "Esta herramienta se utilizará para la investigación de la biología fundamental y tendrá aplicaciones directas dentro de ella. Por ejemplo, muchos cánceres presentan translocaciones y reordenamientos cromosómicos en los que partes de un cromosoma pueden romperse y volverse a unir al cromosoma equivocado, causando todo tipo de problemas. Esta es una característica de muchas otras enfermedades también".
Otra característica única de esta herramienta, que el equipo denominó CHESS (Comparación de Experimentos de Alta-Conducta usando Similitud Estructural), es que se puede comparar la organización del genoma 3D entre diferentes especies, lo que podría ayudar a identificar áreas que se conservan a través de la evolución. "No hay otra herramienta disponible que pueda realizar este análisis sistemáticamente. Esto realmente abre la posibilidad de tener un enfoque cuantitativo y sistemático para caracterizar cómo la organización del genoma cambia entre diferentes muestras o especies", discuten Silvia Galan, estudiante de doctorado en el laboratorio de Marc Marti-Renom, y Nick Machnik, coautores del estudio.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.