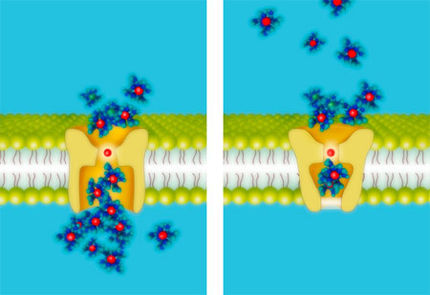
Gene flirten auf Distanz
Geschlossene DNA-Sequenzen erkennen laut einem mathematischen Modell ähnliche Abschnitte in anderen DNA-Doppelsträngen
Anzeigen
Gene können nach Ansicht eines Gastwissenschaftlers am Max-Planck-Institut für Mathematik in den Naturwissenschaften schon von Weitem erkennen, ob ein anderes Gen dem eigenen Aufbau entspricht. "Gene verhalten sich wie Gäste auf einer Party", sagt Alexei Kornyshev, Professor für chemische Physik. "Lernen sich dort zwei Menschen kennen, prüfen sie auch erst aus der Distanz, ob sie füreinander interessant sind." In Leipzig hat Kornyshev nun berechnet, wie weit homologe Abschnitte der DNA, also solche mit sehr ähnlichem Aufbau, voneinander entfernt sein dürfen, um Ähnlichkeiten festzustellen. Homologe Gene tauschen sich etwa aus, wenn Kinder gezeugt werden und sich dabei die Erbinformation der Eltern mischt. "Wüssten wir, wie Gene den richtigen Austauschpartner finden, könnten wir falsche Kombinationen bei künstlicher Befruchtung verhindern", erklärt Kornyshev.
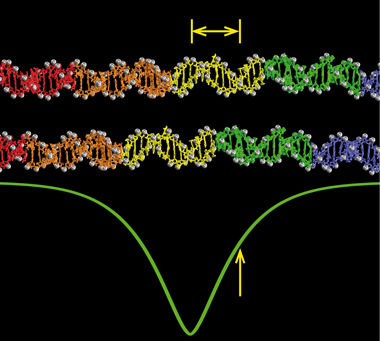
Homologe Sequenzen erkennen sich schon aus gewisser Distanz.
Max-Planck-Institut für Mathematik in den Naturwissenschaften
Trotz der Fülle an menschlichen Genen finden sich im Genom ähnliche Exemplare mit hoher Präzision. Das ist wichtig, denn bei der Vererbung ebenso wie bei der Reparatur defekter Gene dürfen nur Teile ausgetauscht werden, die Informationen für dasselbe Merkmal tragen, die also zu zwei Varianten desselben Gens gehören. Auf die Frage, wie die Gene einander erkennen, wissen die Forscher bislang keine Antwort.
Zwei große Theorien gebe es, erläutert Alexei Kornyshev, der berechnet hat, wie aneinander vorbei gleitende Doppelstränge der DNA wechselwirken. Die eine Theorie geht davon aus, dass ähnliche Sequenzen sich nur finden, wenn der Doppelstrang sich wie ein Reißverschluss geöffnet hat. Kornyshev gehört dagegen zu den Forschern, die meinen, dass homologe Gene, also solche mit vergleichbarer Zusammensetzung, sich schon aus der Ferne identifizieren und ein Auftrennen der Doppelstränge nicht nötig sei.
Möglich ist das - so sein Modell - weil sich zwischen den negativ geladenen Doppelsträngen der DNA, die sich wie eine Wendeltreppe winden, positiv geladene Teilchen festsetzen, etwa Natrium-, Kalium- oder Magnesiumionen. Die unterschiedlich geladenen Bereiche übten anziehende Kräfte aufeinander aus, wenn zwei Doppelstränge aneinander vorbei glitten, schildert der Wissenschaftler.
Nun hat Kornyshevs Team untersucht, wie weit zwei Doppelstränge gegeneinander verschoben werden dürfen, damit sich die ähnlichen Sequenzen noch erkennen. Dazu betrachteten die Forscher, wie durch ein Fenster einen Abschnitt eines Gens und berechneten, wie sich die Energie ändert, wenn ein zweiter DNA-Doppelstrang vorbei gleitet. Dabei stellten sie fest: Ein Gen merkt, dass sich ein ähnliches nähert, bevor die beiden exakt gegenüber stehen. Es "spürt", dass sich eine energetisch günstigere Position bietet.
Solange die homologen Teile um mehr als die Hälfte der Fensterbreite zueinander versetzt sind, entspricht die Wechselwirkung noch der zwischen verschiedenen Genen. Nähern sich die homologen Doppelstränge weiter an, sinkt die Energie. Sie ist am kleinsten, wenn die beiden Doppelstränge einander exakt gegenüber liegen. Die Kurve, die den Energievorteil beschreibt, sieht aus wie die Kontur eines umgedrehten Sombreros. Sie zeigt: Homologe Sequenzen erkennen sich schon aus gewisser Distanz.
Bereits 2001 hatte ein Team um Kornyshev ein Modell aufgestellt, das beschreibt, wie zwei Doppelstränge aufgrund ihrer negativen Phosphate und der zwischen den Strängen angedockten positiven Ionen miteinander in Wechselwirkung treten. Um zu einer mathematischen Formel zu gelangen, modellierten die Wissenschaftler einen Doppelstrang als zwei zueinander parallele Seile, die sich um eine Litfasssäule winden. Die elektrischen Ladungen bildeten sie ab, indem sie die Seile als negativ, die Zwischenräume als positiv geladen ansahen.
Stehen zwei identisch umwickelte Litfasssäulen nebeneinander, dann liegt das geladene Seil der einen Litfasssäule immer exakt den Zwischenräumen der anderen Litfasssäule gegenüber. Das ist energetisch günstig, weil sich unterschiedliche Ladungen anziehen. Diese Position können nur homologe Doppelstränge einnehmen, denn in der DNA windet sich jede Kombination benachbarter Bausteine in einem bevorzugten Winkel. Übertragen auf die Litfasssäule sind die Seile also nicht gleichmäßig aufgewickelt, sondern an manchen Stellen steiler. Stellt man nun zwei Litfasssäulen nebeneinander, die verschiedene DNA-Sequenzen darstellen, liegen nicht immer positive und negative Ladungen gegenüber. Da gleiche Ladungen sich abstoßen, ist diese Situation energetisch von Nachteil. Mit seinen neuen Rechnungen hat Kornyshev nun gezeigt, wie weit der Energievorteil spürbar ist, wenn zwei homologe DNA-Stränge aneinander vorbei gleiten.
"Es ist klar, dass sich Modell und Realität unterscheiden", sagt Kornyshev, "die DNA ist durchaus komplexer als von uns dargestellt". Dennoch hätten erste Versuche im Reagenzglas gezeigt, dass geschlossene homologe DNA-Sequenzen aus mehreren hundert Bausteinen ohne Hilfe von Proteinen zueinander finden. Es sei daher durchaus möglich, dass sich homologe Gene anhand ihrer Struktur erkennen, bevor der komplexe Prozess starte, in dem sich die Stränge auftrennen und austauschen, erläutert der Wissenschaftler.
Originalveröffentlichung: Alexei A. Kornyshev and Aaron Wynveen; "The homology recognition as well as innate property of DNA structure"; PNAS 2009, Online-Vorabpublikation 9. März 2009