Landkarten für das menschliche Erbgut
Saarbrücker Forschungsteam zeigt Krebsforschern, wo sie suchen sollten
Anzeigen
Das Erbgut in Krebszellen unterscheidet sich fundamental von gesunden Zellen. Weite Bereiche sind zerstört oder vervielfältigt, vor allem aber sind die Struktur des Erbguts und seine dreidimensionale "Verpackung" im Zellkern grundlegend gestört. Allerdings ist längst nicht jeder dieser Unterschiede zu gesunden Zellen für die Krebsdiagnose geeignet, und als Ansatz für neue Krebstherapien eignet sich nur ein Bruchteil. Wissenschaftler vom Max-Planck-Institut für Informatik haben in Zusammenarbeit mit Genetikern der Universität des Saarlandes eine "Landkarte des menschlichen Erbguts" berechnet. Damit können Krebsforscher nun gezielt wichtige Veränderungen von Krebszellen identifizieren und analysieren, statt die sprichwörtliche Nadel im Heuhaufen zu suchen. Derartige Landkarten des Erbguts sollen in Zukunft eine zentrale Rolle bei der Identifizierung von krankmachenden Genen und der Suche nach neuen Medikamenten spielen.
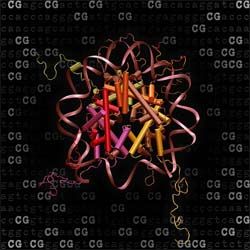
Gen-Schalter: Krebs-relevante Regionen im Genom besitzen oft eine hohe Dichte an CG-Sequenzen (im Hintergrund hell hervorgehoben). Ist die Verpackung der DNA verändert (Vordergrund: DNA wickelt sich spiralförmig um eine Nuklesom-Spule), können wichtige Anti-Krebs-Gene abgeschaltet oder krebsfördernde Gene aktiviert werden.
MPI für Informatik / Bock
Um die Ursachen beispielsweise von Krebs besser zu verstehen, muss man das Zusammenspiel des Erbguts mit seiner "Verpackung" und sogenannten epigenetischen Schaltern untersuchen. Schäden in der Verpackung und fehlerhafte Einstellungen der Schalter können nämlich dazu führen, dass wichtige Gene deaktiviert und gefährliche Gene fälschlicherweise aktiviert werden - die Zellen spielen dann plötzlich "verrückt" und wuchern unkontrolliert in gesundes Gewebe hinein. Sogenannte CpG-Inseln sind wichtige epigenetische Schalter. Hierbei handelt es sich um spezielle Bereiche der DNA, in denen der Anteil von Cytosin-Guanin-Sequenzen (CpGs) deutlich erhöht ist. Mehr als drei Viertel aller menschlichen Gene haben CpG-Inseln in ihren Startbereichen. Eine Veränderung der DNA-Verpackung innerhalb von CpG-Inseln kann dazu führen, dass das zugehörige Gen nicht mehr abgelesen wird (Gene-Silencing).
Christoph Bock aus der Abteilung von Thomas Lengauer am Max-Planck-Institut für Informatik ist es nun erstmals gelungen, epigenetisch kontrollierte und Krebs-relevante Regionen im gesamten Erbgut zu kartieren und ihre Funktion zu klassifizieren. Seine Grundidee: Jede Region im menschlichen Erbgut besitzt einen "natürlichen" epigenetischen Packungszustand, der mit ihrer DNA-Sequenz bereits festgelegt ist. Normale und krankhafte Veränderungen der Verpackung stellen sich als Abweichungen von diesem Grundzustand dar. "Wenn wir die Epigenetik als Software und die DNA-Sequenz als die Hardware des menschlichen Erbgutes betrachten, dann scheint die Hardware ein epigenetisches Standard-Programm zu definieren. Dieses Programm wird solange ausgeführt, bis die epigenetische Software in der Zelle gezielt aktiviert wird und individuelle Veränderungen initiiert", erklärt der Bioinformatiker Bock.
Durch einen bioinformatischen Trick konnte er diesen "natürlichen" Packungszustand des Erbgutes berechnen, der sich in reiner Form in keiner biologisch existierenden Zelle befindet und doch das Grundgerüst für alle Zellen darstellt. Dazu kombinierte er nicht nur Daten von sieben verschiedenen epigenetischen Mechanismen und verschiedenen Zelltypen, sondern führte zusätzlich einen statistischen Abstraktionsschritt ein. Basierend auf Daten für jeden einzelnen Mechanismus wurde ein statistisches Lernverfahren trainiert (eine sogenannte Support-Vektor-Maschine), das die besonderen Merkmale der DNA-Sequenz verschiedener Regionen identifiziert. Durch geschickte Kombination trainierter Modelle konnte dann eine Vorhersage des "natürlichen" Packungszustandes für alle CpG-Inseln im menschlichen Erbgut bestimmt werden.
Wie eine Landkarte die Navigation in unbekanntem Gelände erleichtert, so helfen solche bioinformatische Landkarten sich im Datendschungel des menschlichen Erbgutes zurechtzufinden. "Für zukünftige Ansätze einer epigenetischen Krebstherapie eignen sich möglicherweise jene Regionen, die in Krebszellen nur leicht von ihrem Normalzustand abweichen und die sich durch eine moderate und nebenwirkungsarme Therapie zurücksetzen lassen", erklärt Jörn Walter, der Genetiker im Team. Und da sich derartige Landkarten prinzipiell nicht nur für Krebs-spezifische epigenetische Schalter erstellen lassen, sondern auch für andere biologische Effekte und Krankheiten, werden sie eine wichtige Rolle spielen, wenn es darum geht unser Erbgut zu analysieren und neue Therapiemöglichkeiten zu entwickeln. "Die Suche nach der Nadel im Heuhaufen reduziert sich somit auf die Suche in einer Schachtel Strohhalme", resümiert Christoph Bock schmunzelnd.
Originalveröffentlichung: Christoph Bock, Jörn Walter, Martina Paulsen und Thomas Lengauer; "CpG Island Mapping by Epigenome Prediction"; PLoS Computational Biology, Volume 3, Issue 6, June 2007.