Blick in die Zukunft: Deep Learning sagt Entwicklung von Blutstammzellen voraus
Anzeigen
Autonomes Fahren, automatische Spracherkennung oder das Spiel Go – die Methode des Deep Learning ist momentan in aller Munde. Wissenschaftler am Helmholtz Zentrum München und ihre Partner an der ETH Zürich sowie der Technischen Universität München haben sie nun eingesetzt, um die Entwicklung von Blutstammzellen im Voraus zu berechnen. In Nature Methods beschreiben sie, wie ihre Software anhand von Mikroskopie-Bildern den künftigen Zelltyp vorhersagt.
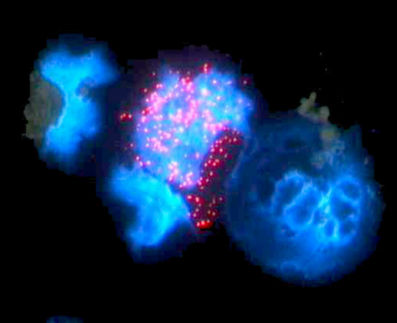
Was wohl aus ihnen wird? Blutstammzellen unter dem Mikroskop: Mit Hilfe neuer Methoden können die Helmholtz-Wissenschaftler voraussagen, wie sie sich entwickeln werden.
Helmholtz Zentrum München
Die Zellbiologie befasst sich heutzutage nicht mehr nur mit statischen Zuständen, sondern versucht auch dynamische Entwicklungen von Zellgruppen zu verstehen. Ein Beispiel dafür ist die Bildung verschiedener Typen von Blutzellen aus ihren Vorläufern, den Blutstammzellen. „Die Entscheidung einer Blutstammzelle für einen Zelltyp lässt sich nicht beobachten. Man kann sie bisher nur nachträglich durch Zelloberflächenmarker nachweisen“, erklärt Dr. Carsten Marr, Leiter der Arbeitsgruppe Quantitative Single Cell Dynamics am Institute of Computational Biology (ICB) des Helmholtz Zentrums München.
Er und sein Team haben nun einen Algorithmus entwickelt, der die Entscheidung bereits im Vorfeld voraussagen kann. Dazu wendeten sie das sogenannte Deep Learning an. „Bei unserer Methode spielen Deep Neural Networks, also künstliche Intelligenz, eine tragende Rolle“, so Marr. „Unser Algorithmus wertet lichtmikroskopische Bilder und Videos einzelner Zellen aus und gleicht diese Daten mit bisherigen Erfahrungen zur Entwicklung derartiger Zellen ab. Der Algorithmus ‚lernt‘ auf diese Weise wie sich bestimmte Zellen verhalten.“
Drei Generationen schneller als bisherige Methoden
Konkret untersuchten die Forscher Blutstammzellen, die im Labor von Timm Schroeder an der ETH Zürich unter dem Mikroskop gefilmt worden waren. Anhand der Informationen über Aussehen und Geschwindigkeit konnte die Software sich die entsprechenden Verhaltensmuster einprägen und anschließend voraussagen. „Verglichen mit herkömmlichen Methoden, etwa fluoreszierenden Antikörpern gegen bestimmte Oberflächenproteine, wissen wir drei Zellgenerationen früher, wie sich die Zellen entscheiden werden“, berichtet ICB-Wissenschaftler Dr. Felix Buggenthin, gemeinsam mit Dr. Florian Büttner Erstautor der Studie.
Doch was bringt den Forscher dieser Blick in die Zukunft? Studienleiter Marr beschreibt es so: „Weil wir nun wissen, welche Zelle sich wie entwickelt, können wir diese früher als bisher isolieren und untersuchen wie sie sich molekular unterscheiden. Dadurch wollen wir aufklären, wie es zur jeweiligen Entscheidung für eine Entwicklungslinie kommt.“
In Zukunft wird es nicht bei den Blutstammzellen bleiben. „Wir verwenden Deep Learning für ganz unterschiedliche Fragestellungen mit ausreichend großen Datensätzen“, erklärt Prof. Dr. Dr. Fabian Theis, ICB-Direktor und Inhaber des Lehrstuhls für Mathematische Modelle biologischer Systeme an der TU München, der zusammen mit Carsten Marr die Studie leitete. „So analysieren wir mit ganz ähnlichen Algorithmen krankheitsassoziierte Muster im Genom und identifizieren Biomarker in klinischen Zell-Screens.“
Originalveröffentlichung
Buggenthin, F. et al.; "Prospective identification of hematopoietic lineage choice by deep learning. Nature Methods"; 2017