Baukasten für Designer-Proteine
Wissenschaftler entwickeln eine Methode, um hochentwickelte Proteine im Großmaßstab herzustellen
Proteine sind einer der wichtigsten Bestandteile eines jeden lebenden Organismus und für mannigfaltige Aufgaben in Biosystemen zuständig: Wie Hebel und Zahnräder bei Maschinen steuern, veranlassen und unterstützen sie biologische Aktivitäten und Funktionen innerhalb der Zelle. In der Medizin werden Proteine als Antikörper in der Impfstoffentwicklung benötigt. Eine der größten Herausforderungen der heutigen Zeit ist es, neuartige Impfstoffe gegen Krebserkrankungen zu entwickeln. Wissenschaftler des Karlsruher Instituts für Technologie (KIT) haben zusammen mit internationalen Forschungspartnern eine Methode etabliert, um genetisch gezielt veränderte Proteine in größerem Maßstab herzustellen. Die schnelle Herstellung der Designer-Eiweißbausteine könnte neue Anwendungen in der Biotechnik und Pharmazie ermöglichen.
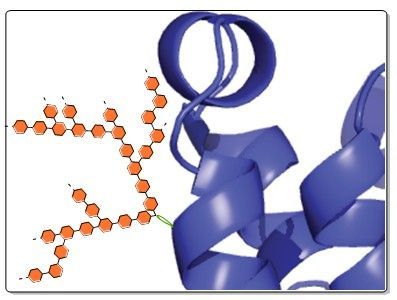
Schematische Darstellung eines genetisch gezielt veränderten Proteins mit Glykanstruktur
KIT
„Proteine sind für alle lebenden Organismen wichtige Bausteine“, sagt Stefan Bräse, Professor am Institut für Organische Chemie und Direktor am Institut für Toxikologie und Genetik des KIT. „Zellen, Muskeln, Organe enthalten Proteine. Komplexe Proteinsysteme (de-)codieren die genetische Information der DNA, um wiederum weitere Proteine mit unterschiedlichsten Aufgaben zu erzeugen.“ Für die Anwendung in der Forschung und Medizin kommen häufig biotechnologisch hergestellte Eiweiße zum Einsatz, deren Eigenschaften im Vergleich zur Ursprungsform verändert und auf das neue System angepasst sind: Diese rekombinanten Proteine werden mithilfe von gentechnisch gezielt veränderten DNA-Viren modifiziert. Die neue Struktur trägt die angestrebte Zielfunktion. Lange fand als Expressionsmedium, sprich als Maschinerie, um neuartige Proteine herzustellen, das Darmbakterium Escherichia coli (E.coli) Anwendung. „E.coli ist ein sehr einfacher Organismus“, erklärt Bräse. Andererseits habe E.coli auch einige Nachteile bezüglich der Biosynthese von Designer-Proteinen.
Deshalb haben sich in den letzten Jahren Insektenzellen, sogenannte Sf21-Zellen, als geeignetes Expressionssystem bewährt, zusammen mit dem „MultiBac-System“. Dieses System nutzt einen Transfer-Vektor, also eine Art „Genfähre“, welche die benötigten Informationen zum Aufbau des rekombinant modifizierten Proteins enthält. „Durch eine Kombination des MultiBac-Systems mit seitenspezifischen Gen-Engineering-Methoden, dem ‚MultiBacTAG‘, können wir nahezu beliebige Proteine mit integrierten Zielfunktionalitäten in großen Mengen und mit hoher Qualität herstellen“, sagt Dr. Edward Lemke, Gruppenleiter für Hochauflösende Untersuchungen am Europäischen Molekularbiologischen Labor (EMBL) in Heidelberg. „Das System benötigt dafür nur einige Wochen. Wir haben es direkt für unsere Glykanstrukturen genutzt“, ergänzt Bräse.
Mit dieser Methode gelang es den internationalen Forschungspartnern ortsspezifisch unnatürliche Aminosäuren in das Zielprotein einzuführen. Ein neu designtes, orthogonales Enzym, das tRNA/ tRNA-Synthetase-Paar, erkennt die unnatürlichen Aminosäuren und kann sie bequem in die gewünschte Zielstruktur einbauen.
Durch die Kombination des MultiBac-Systems mit dem orthogonalem tRNA/ tRNA-Synthetase-Paar ist es Lemke und Bräse mit ihren Gruppen gelungen, Proteinkomplexe mit Zellkerne in Insektenzellen herzustellen, die unnatürliche Aminosäuren tragen und somit eine Vielzahl von Anwendungen erlauben. Die Technologie zur Erweiterung des genetischen Codes ist für die heutige Medizin und Biotechnologie unverzichtbar. „MultiBacTAG ist nutzerfreundlich und leicht nachvollziehbar. Da die Komponenten der Gencode-Veränderung in die Struktur von MultiBacTAG eingesetzt sind, können Nutzer unsere Erweiterung ohne vorherige Erfahrung oder Kenntnisse verwenden“, sagt Bräse.
In Tests konnten die chemischen Biologen an Insekten- und Säugerzellen die Anwendung für eine Vielfalt von Proteinen und Proteinkomplexen aufzeigen. So konnten sie beispielsweise Herceptin herstellen, ein Protein, das als Antikörper bei Brustkrebserkrankungen verwendet wird. „Wir gehen davon aus, dass MultiBacTAG eine breite Palette von Möglichkeiten für das individuelle Proteindesign in biotechnologischen und pharmazeutischen Anwendungen ermöglicht“, fasst Bräse zusammen. „Es könnte vor allem in der Erforschung von Proteinkomplexen und deren funktionellen Wechselwirkungen sehr nützlich sein.“