Wenn Bilder trügen
Bilder aus der Nanoskopie richtig deuten
Was man bisher für Proteincluster auf Zelloberflächen hielt, ist in Wahrheit oft lediglich eine Mehrfachzählung. Eine Methode der TU Wien kann beides nun unterscheiden.
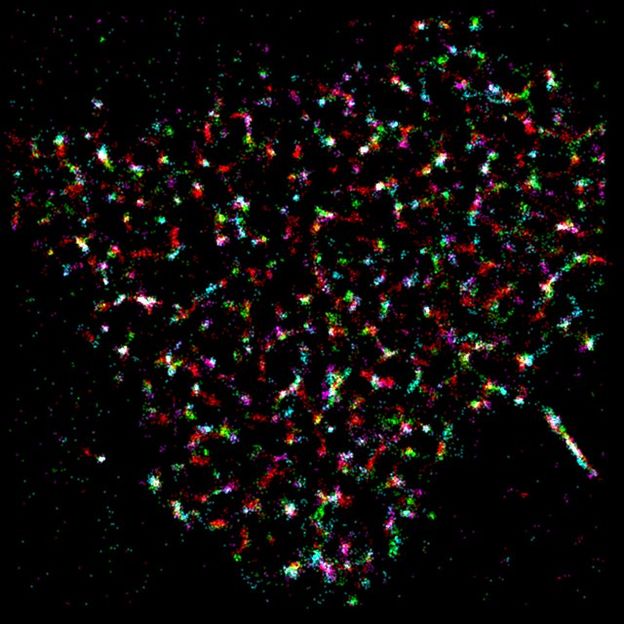
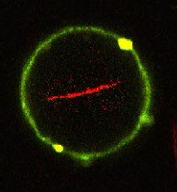
Ein typisches Bild, zusammengesetzt aus mehreren Mikroskop-Aufnahmen.
Copyright: TU Wien
Mit Licht kann man keine Strukturen abbilden, die kleiner sind als die halbe Wellenlänge - zumindest dachte man das für lange Zeit. Die Entwicklung der sogenannten Nanoskopie hat allerdings gezeigt, dass diese Regel gewisse Schlupflöcher offenlässt. Wenn man unterschiedliche Moleküle zu unterschiedlichen Zeitpunkten aufleuchten lässt, kann man sie am Ende zu einem scharfen Bild zusammenfügen. Im Jahr 2014 wurde dafür der Chemie-Nobelpreis vergeben. Inzwischen ist die Nanoskopie eine weltweit angewandte Methode, mit der unter anderem die Struktur der Zellmembran untersucht wird.
Dabei beobachtete man überraschenderweise immer wieder, dass sich Membranproteine in Gruppen zusammenzuballen schienen. Auch an der TU Wien untersuchte man diese Cluster und erkannte, dass es sich bei vermeintlichen Gruppen von Proteinen oftmals um einzelne blinkende Moleküle handelt, die mehrfach gezählt werden. Eine neue Methode, die das Team der TU Wien nun im Fachjournal „Nature Methods“ publizierte, kann zwischen echten Clustern und solchen Artefakten unterscheiden.
Moderne Abbildungsverfahren für die Biologie
„Für viele biologische oder medizinische Fragestellungen ist es entscheidend, die Struktur der Zellmembran genau zu verstehen“, sagt Florian Baumgart von der Biophysik-Forschungsgruppe um Gerhard Schütz am Institut für Angewandte Physik der TU Wien. „Nanoskopie ist ein ideales Werkzeug, um die räumliche Anordnung von Proteinen auf der Zellmembran zu untersuchen. Wir forschen an T-Zellen, die Antigene erkennen und dadurch eine zentrale Rolle in unserem Immunsystem spielen.“
Um Nanoskopie auf biologische Proben anzuwenden, werden Proteine mit fluoreszierenden Molekülen (sogenannten Fluorophoren) markiert. In der klassischen Fluoreszenzmikroskopie ist ein einzelner Fluorophor nicht als klarer Punkt, sondern als leicht verschwommene Scheibe sichtbar. Leuchten alle Moleküle der Probe gleichzeitig, überlappen ihre Abbildungen und man verliert die Information über ihre räumliche Anordnung. Über einen chemischen Trick kann man die Fluorophore allerdings zum Blinken bringen. Man nimmt dann eine Serie von Bildern auf, wobei Jeweils immer nur einige wenige Fluorophore aufleuchten. Am Computer kann man die Position der Moleküle in jedem Einzelbild bestimmen und daraus schließlich ein hochaufgelöstes Bild der Probe rekonstruieren.
Mysteriöse Cluster
„Verschiedenen Arbeitsgruppen haben in der Vergangenheit untersucht, wie die Proteine auf der Zellmembran angeordnet sind. Dabei wurde immer wieder beobachtet, dass sich fast alle untersuchten Proteine nicht zufällig verteilen, sondern an bestimmten Stellen geballt als Cluster vorzukommen scheinen“, erklärt Florian Baumgart.
Das wirkte durchaus glaubwürdig. Man wusste bereits, dass T-Zellen bei der Antigenerkennung in kurzer Zeit stabile Proteincluster ausbilden können, die so groß sind, dass man sie sogar mit klassischer Fluoreszenzmikrokopie nachweisen kann. Nanoskopisch kleine Proteincluster, so dachte man, könnten Vorläufer dieser größeren Strukturen sein – mit entscheidender Bedeutung für die Funktion von T-Zellen.
Auch Baumgart machte sich auf die Suche nach solchen Nanoclustern. Aber Nanoskopie ist eben nicht so einfach wie Urlaubsfotos schießen. Es gibt viele Fehlerquellen, man muss die gewonnenen Daten sorgfältig auswerten, um zuverlässige Ergebnisse zu erhalten. Manche Moleküle können wiederholt aufleuchten, diese mehrfachen Lichtsignale kann man dann leicht als Molekülcluster fehlinterpretieren.
„Wir haben überlegt, wie man Cluster abwechselnd leuchtender Moleküle von einem einzigen, immer wieder blinkenden Molekül unterscheiden kann“, berichtet Florian Baumgart. Gelungen ist das, indem die Konzentration der zur Markierung verwendeten Fluorophore schrittweise verändert wurde. „In den aufgenommen Bildern können zufällig verteilte Moleküle mehrfach abgebildet werden, oder es kann sich tatsächlich um Clusterbildung handeln“, sagt Baumgart. „Doch statistisch unterscheidet sich die Verteilung der Molekülpositionen in den beiden Fällen.“ Versammeln sich die Proteine tatsächlich in Clustern, dann werden dort zwar mehr Molekülpositionen sichtbar, aber dazwischen bleiben dunkle Freiräume. Handelt es sich allerdings um einzelne Moleküle, die mehrfach aufleuchten, dann wird bei steigender Konzentration fluoreszierender Moleküle die gesamte Fläche der Probe mit Molekülpositionen aufgefüllt ohne sich lokal massiv zu häufen.
„Auf diese Weise fanden wir, dass die untersuchten Membranproteine der T-Zellen vor deren Aktivierung gar keine Cluster bilden – diese Theorie muss verworfen werden“, sagt Florian Baumgart. „Es gibt auch Beispiele für Proteine, die nachweislich molekulare Cluster ausbilden. Mit ihnen ergeben sich dann Bilder mit ganz anderen statistischen Eigenschaften als die zufällig verteilten T-Zell Proteine.“
Die Methode, mit der man Proteincluster von mehrfach blinkenden Molekülen unterscheiden kann, wurde nun im Fachjournal „Nature Methods“ publiziert. Sie soll in Zukunft helfen, Bilder aus der Nanoskopie richtig zu deuten und die großen Rätsel der Zellmembran aufzuklären.