Molekulares Daumenkino: Momentaufnahmen aus dem Inneren der Zelle
Forscher beobachten, wie sich die Struktur der RNA-Polymerase bei der Arbeit verändert
Einem Forschungsteam um den Braunschweiger Physikochemiker Prof. Philip Tinnefeld und der Biochemikerin Prof. Dina Grohmann ist es gelungen, Veränderungen in der RNA-Polymerase während ihrer Arbeit sichtbar zu machen. Im Milliardstel Meter Bereich haben sie dafür Farbstoffe auf zwölf Proteinen platziert, deren Helligkeit sich je nach ihrer Entfernung zueinander verändert. Vergleichbar mit einem Daumenkino, konnte das Forschungsteam mit seinen Aufnahmen außerdem nachweisen, dass die Arbeit der RNA-Polymerase durch verschiedene Faktoren gesteuert und beeinflusst wird.
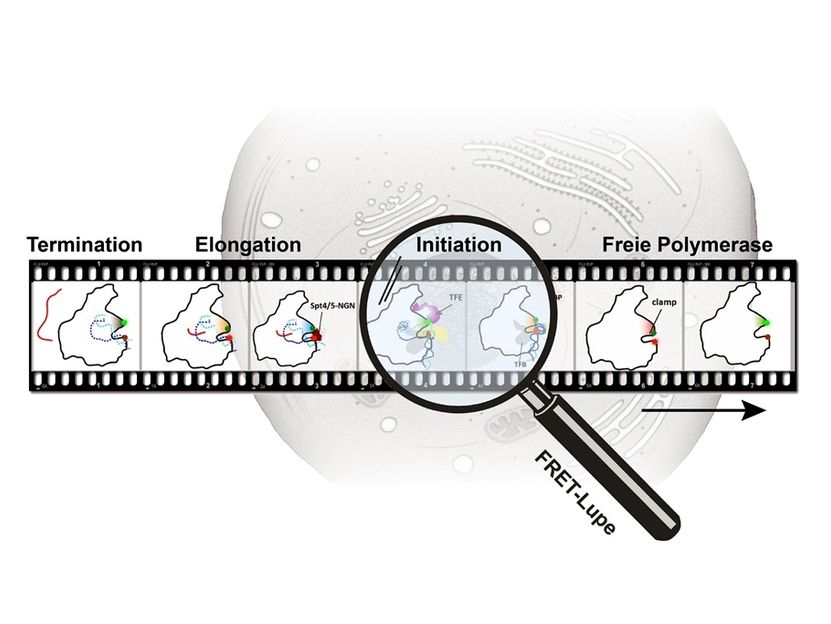
Während ihres Aktivitätszyklus in der Zelle interagiert die RNA Polymerase sowohl mit DNA als auch mit verschiedenen Faktoren. Die damit einhergehenden unterschiedlichen Konformationszustände der RNA-Polymerase konnten über die FRET-Technik mit nanometergenauer Auflösung analysiert werden, sodass ein „molekulares Daumenkino der RNA-Polymerase“ erstellt werden konnte.
PCI/TU Braunschweig
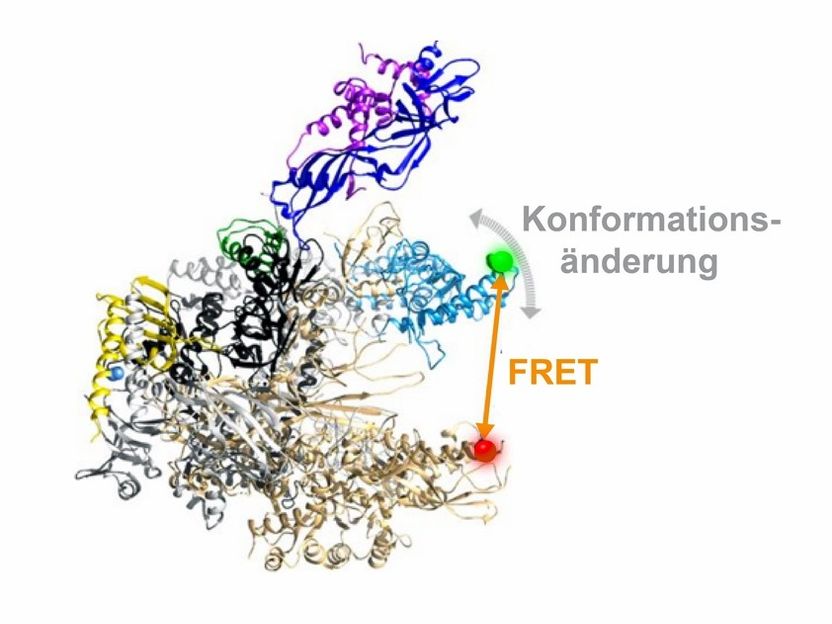
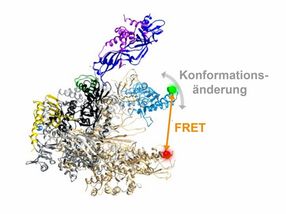
Dreidimensionale Architektur einer RNA Polymerase. Die flexible Einheit der RNA-Polymerase, die verschiedene Konformationen annehmen kann, ist in blau hervorgehoben. Die Position der Fluoreszenzfarbstoffe, über die der mit einem orangen Pfeil angezeigte Abstand in der RNA-Polymerase und damit die Bewegung der flexiblen Einheit gemessen werden kann, ist mit einem grünen und roten Punkt gezeigt.
PCI/TU Braunschweig
Ob Bakterium, Pflanze oder Tier – Nur wenige Enzyme sind baugleich in allen lebenden Organismen wiederzufinden. Unter diesen Molekülen befindet sich eine der zentralen Arbeitseinheiten einer Zelle, die RNA-Polymerase. Ihre Aufgabe ist es, die in der DNA kodierte genetische Information zu lesen und umzuschreiben, die so genannte Transkription. Reguliert und gesteuert wird diese Arbeit durch unterschiedliche Faktoren, deren Einfluss und Wirkung auf die molekulare Übersetzungsmaschine bislang ungeklärt sind. „Es immer noch ein Rätsel, wie dieser hochkonservierte biologische Prozess mit ungeheurer Verlässlichkeit und Kontrolle durch die Transkriptionsfaktoren in den Zellen ablaufen kann“, sagt Prof. Tinnefeld.
Die atomare Aufschlüsselung der RNA-Polymerase-Struktur über Röntgenstrukturanalysen liefert der Wissenschaft sozusagen nur ein Standbild der dynamischen molekularen Maschine. Die so genannten Konformationsänderungen, die einen entscheidenden Schlüssel für die Funktionsweise der hochkomplexen Transkriptionsmaschinerie darstellen, konnten damit jedoch nicht erfasst werden. Prof. Philip Tinnefeld und seiner Arbeitsgruppe „NanoBioScience“ ist es nun gelungen, die Vermutung von der Veränderung der RNA-Polymerase während ihrer Arbeit zu bestätigen und darüber hinaus auch die Faktoren und deren Wirkungsweise aufzuklären, die sie dabei beeinflussen. „Das Verständnis von der Arbeitsweise der RNA-Polymerase, ihrer beeinflussenden Faktoren und deren Wirkungsweise legt die Grundlage für die Aufklärung erblich bedingter Krankheiten sowie die Entwicklung von personalisierter Medizin“, erklärt Prof. Dina Grohmann.
Helligkeit von Farbstoffen zeigte Abstand der Moleküle zueinander
Um einen Einblick in die Funktion der RNA-Polymerase und der sie steuernden Transkriptionsfaktoren unter nahezu unbeeinflussten Bedingungen zu erhalten, hat das Forschungsteam eine Einzelmolekültechnik eingesetzt. Verfolgt haben sie damit bestimmte, relevante Abstände mit nanometergenauer Auflösung durch den gesamten Transkriptionsprozess auf der Polymerase. Dazu wurden der Komplex aus RNA-Polymerase und DNA auf einem Deckglas zusammengesetzt und mit verschiedenen Fluoreszenzfarbstoffen spezifischen Stellen markiert. In einem als FRET (Förster Resonanz-Energietransfer) bezeichneten Prozess übertragen die beiden Farbstoffe abhängig von ihrem Abstand zueinander Energie. Befinden sie sich in räumlicher Nähe, leuchtet der rote Farbstoff, sind sie weiter auseinander positioniert, leuchtet der grüne Farbstoff stärker. Aus dem Verhältnis der Farbintensitäten lassen sich dann exakte Abstände und Abstandsänderungen auf der RNA-Polymerase quantitativ messen. Auf diese Weise hat das Team tausende einzelner Transkriptionskomplexe untersucht, so dass die Veränderung der RNA-Polymerase in jeder Phase ihrer Aktivität bestimmt werden konnte. „Im Gegensatz zum Standbild, haben wir mit unserer Arbeit quasi einen molekularen ‚Stop-Motion‘ Film der aktiven RNA-Polymerase erstellt, der im Detail und mit hoher Präzision die hohe Flexibilität der Transkriptionsmaschinerie zeigt“, fasst Prof. Tinnefeld das Forschungsergebnis zusammen.