Der Schlüssel zur Proteinbindung - ungeordnet, aber ultraschnell
Anzeigen
Die Kommunikation in Zellen zwischen Zellkern und Zellplasma wird durch den dauerhaften Austausch von Tausenden von Signalmolekülen und Proteinen vermittelt. Bisher war unbekannt, warum dieser Proteinverkehr zugleich so schnell und doch exakt genug ist, um unerwünschte Moleküle an der Passage zu hindern. Durch eine Kombination von Computersimulationen und experimentellen Techniken haben Forscher aus Deutschland, Frankreich und England dieses Rätsel gelöst: Ein sehr bewegliches Protein kann an seinen Rezeptor innerhalb von Millardstel Sekunden binden.
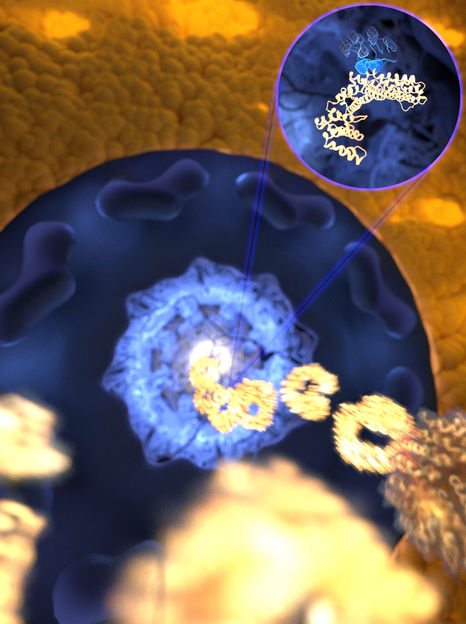
Durch die ultraschnelle, aber zugleich gezielte Bindung rast der Rezeptor (goldfarben) durch die mit ungeordneten Proteinen gefüllte Pore in den Zellkern, während unerwünschte Moleküle ferngehalten werden.
Mercadante /HITS
Proteine können sich gegenseitig erkennen. Jedes einzelne verbindet sich sehr spezifisch nur mit wenigen der vielen verschiedenen Proteine in der lebenden Zelle - wie ein Schlüssel, der in ein Schlüsselloch passt. Aber was passiert, wenn der Schlüssel extrem beweglich ist, wie bei den sogenannten intrinsisch ungeordneten Proteinen (IDPs)? Die Forschungsgruppen unter der Leitung von Edward Lemke am European Molecular Biology Laboratory (EMBL) in Heidelberg, Frauke Gräter am Heidelberger Institut für Theoretische Studien (HITS) und Martin Blackledge am Institut de Biologie Structurale (IBS) in Frankreich gingen dieser Frage in einer stark interdisziplinären Zusammenarbeit nach. Sie verknüpften dabei molekulare Simulationen, Fluoreszenz-Resonanzenergietransfer (FRET) von Einzelmolekülen, Nuklearmagnetresonanzverfahren (NMR), sogenannte „stopped flow“-Spektroskopie und Teilchenverfolgung in der Zelle (in-cell particle tracking) miteinander.
Überraschenderweise fanden sie heraus, dass bewegliche, Spaghetti-artige Proteine von ihrem Bindungspartner gut erkannt werden können, vielleicht sogar besser als steife Proteinblöcke. Dabei binden sie sehr schnell, behalten aber trotzdem die hohe spezifische Genauigkeit bei, die die Zelle benötigt. Die Forscher vermuten, dass diese ungeordneten Moleküle in evolutionär höher entwickelten Organismen darum häufiger vorkommen.
Wissenschaftler hatten angenommen, dass ein IDP-„Schlüssel“, damit er in sein „Schlüsselloch“ passt, sich selbst umbaut, aber die Experimente im Lemke Lab ließen eine andere Erklärung zu. „Die Einzelmolekülexperimente am EMBL zeigten für diese Wechselwirkung zwischen Rezeptor und ungeordnetem Protein quasi nichts: Das Protein blieb in der Bindung mit dem Rezeptor genauso beweglich“, sagt Davide Mercadante (HITS). Dieser Befund regte ihn dazu an, die gleiche Wechselwirkung am Computer zu untersuchen. Das überraschende Ergebnis: Die hohe Flexibilität des IDP hilft tatsächlich, in das „Schlüsselloch“ zu passen – in diesem Fall ein Transportrezeptor, der Proteine in den Zellkern befördert. Die Simulationen legten sogar nah, dass die Bindung ultraschnell sein würde, schneller als alle Verbindungen dieser Art, die bislang gefunden wurden. „Die Computerdaten deuteten darauf hin, dass wir einen neuen, ultraschnellen Bindungsmechanismus identifiziert hatten, aber wir brauchten drei Jahre, bis wir unsere Experimente so eingerichtet hatten, dass wir die Kinetik im Labor beweisen konnten“, erinnert sich Iker Valle Aramburu (EMBL). „Am Ende erzielten wir eine bemerkenswert perfekte Übereinstimmung.“
Die Ergebnisse helfen, ein langjähriges Paradox zu verstehen. „Damit eine Zelle lebensfähig bleibt, müssen sich Moleküle dauerhaft in den Zellkern und wieder hinaus bewegen“, sagt Edward Lemke (EMBL). „Unsere Erkenntnisse erklären das sogenannte Transportparadox – nämlich wie dieser Transport schnell, aber zur gleichen Zeit hochgenau sein kann, damit unerwünschte Moleküle die Schranke nicht überwinden können, die unser Genom schützt.“
Die neue Studie legt nahe, dass es viele sogenannte Bindungsstellen an der Oberfläche gibt, die eine hochreaktive Oberfläche schaffen. Dadurch kann der Schlüssel ultraschnell viele Schlösser erkennen und aufschließen, so dass die Rezeptoren im Eiltempo durch eine Pore gefüllt mit ungeordneten Proteinen rasen.
„Dies könnte ein neues Paradigma für die Erkennung von intrinsisch ungeordneten Proteinen sein“, sagt Frauke Gräter (HITS). Da dreißig bis fünfzig Prozent der Proteine in menschlichen Zellen ungeordnet sind, könnten die Forschungsergebnisse auch eine Erklärung dafür liefern, warum Informationen so schnell weitergeleitet werden – eine unerlässliche Voraussetzung für das Überleben der Zelle.
Originalveröffentlichung
Sigrid Milles, Davide Mercadante, Iker Valle Aramburu, Malene Ringkjøbing Jensen, Niccolò Banterle, Christine Koehler, Swati Tyagi, Jane Clarke, Sarah L Shammas, Martin Blackledge, Frauke Gräter, Edward A Lemke; Plasticity of an ultrafast interaction between nucleoporins and nuclear transport receptors; Cell