"Chamäleon-Proteine" im neuen Licht
Neutronen enthüllen das Kräftespiel des Basischen Myelinproteins
Anzeigen
Intrinsisch ungefaltete Proteine, kurz IDP, sind die Verwandlungskünstler unter den Eiweißmolekülen. In wässriger Lösung nehmen sie keine klar festgelegte Struktur an und können sich sehr flexibel verbiegen. Wo herkömmliche Methoden versagen, konnten Jülicher Neutronenforscher dank eines neuartigen Ansatzes nun neue Erkenntnisse über diese chamäleonhaften Proteine gewinnen. Die Ergebnisse brechen mit gängigen Vorstellungen zur biomolekularen Funktionsweise eines wesentlichen Bausteins des zentralen Nervensystems und wurden in der Fachzeitschrift "JACS" veröffentlicht.
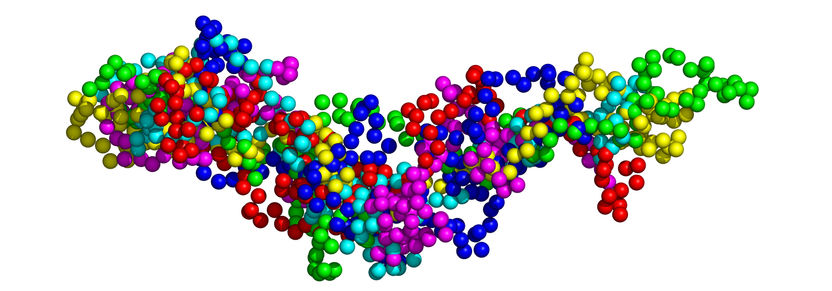
Die Abbildung zeigt eine Überlagerung von sechs möglichen Anordnungen des Basischen Myelinproteins. Jede Kugel steht für eine Aminosäure, die Farbe zeigt die Zugehörigkeit zu jeweils einem Proteinmolekül. Solche vereinfachten Strukturmodelle ermöglichten den Jülicher Forschern, die gemessenen Bewegungen den Bereichen des Proteins zuzuordnen.
Forschungszentrum Jülich

Das Instrument, mit dem die Jülicher Forscher die Bewegungen der Proteine vermessen haben, trägt einen langen Namen: Jülicher Neutronenspinechospektrometer. Dank der besonderen Eigenschaften von Neutronen erkennt es Bewegungen in einer Größenordnung von Nanometern pro Nanosekunde – langsame Bewegungen in der Welt der Physik. In greifbarere Dimensionen umgerechnet entspricht dies einer Geschwindigkeit von Zentimetern bis Metern pro Sekunde.
W. Schürmann /TU München

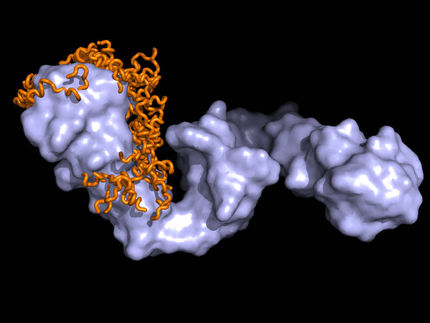
Das Basische Myelinprotein (MBP) ist ein wichtiger Bestandteil des Myelins, das die Nervenzellen im Hirn von Wirbeltieren umhüllt. Sie bilden eine elektrische Isolationsschicht, deren Beschädigung mit zahlreichen schweren Krankheiten wie etwa Multiple Sklerose in Verbindung gebracht wird. Im Gegensatz zu den steiferen globulären Proteinen können sich intrinsisch ungefaltete Proteine wie MBP in wässriger Lösung stark verformen. Dies ist vermutlich der Grund, warum sie an verschiedene Molekülsorten binden und so vielfältige Aufgaben in biologischen Prozessen übernehmen können.
Mit herkömmlichen Methoden sind IDP nur schwer zu erforschen. Diese benötigen üblicherweise kristallisierte Proteine, wobei IDP-Kristalle nur schwer herzustellen sind. Die Forscher um Dr. Andreas Stadler vom Jülich Centre for Neutron Science nutzten nun einen neuen Ansatz, für den keine Kristalle notwendig sind. Dabei fanden sie heraus, dass das Protein einen kompakten zentralen Kern besitzt und nur an den Enden frei beweglich ist. Die dennoch hohe Flexibilität entsteht durch kollektive Streck- und Beugebewegungen großer Teile des Proteins. "Unsere Daten zeigen deutlich, dass die gängigen Modellvorstellungen zur Dynamik von intrinsisch ungefalteten Proteinen falsch sind", betont Stadler. Diese Modelle wurden aus der Polymerphysik abgeleitet und können den kompakten Kern nicht hinreichend abbilden.
Für ihre Messungen nutzten die Wissenschaftler Röntgenkleinwinkelstreuung an der European Synchrotron Radiation Facility (ESRF) im französischen Grenoble und Neutronenstreuung am Heinz Maier-Leibnitz Zentrum in Garching bei München. Die Messergebnisse allein lassen allerdings noch keine direkten Rückschlüsse auf die Moleküldynamik zu. Erst nachdem die Jülicher Forscher die ermittelten Werte über verschiedene Größenbereiche mit am Computer erzeugten Strukturmodellen kombiniert hatten, konnten sie die Bewegungen der einzelnen Molekülbestandteile rekonstruieren.