Krebserkrankungen in frühen Stadien erkennen
„Spektrale Histopathologie“ bei Darmkrebs
Anzeigen
Wissenschaftler der Ruhr-Universität Bochum (RUB) haben eine neue spektroskopische Methode entwickelt, die Pathologen bei der Krebsdiagnose unterstützt. In den Fachzeitschriften „Journal of Biophotonics“ und „Analyst“ verglichen sie die mit der sogenannten Marker-freien „Spektralen Histopathologie“ gewonnenen Ergebnisse mit denen herkömmlicher Diagnoseverfahren am Beispiel Dickdarmkrebs. „Im Gegensatz zu bisherigen Methoden müssen wir das Gewebe nicht mehr färben, um Krebs zu erkennen“, sagt Prof. Klaus Gerwert vom Proteinforschungskonsortium PURE (Protein Research Unit Ruhr within Europe) der RUB. „Das eröffnet die Möglichkeit, Proben in Zukunft automatisch als krankes oder gesundes Gewebe zu klassifizieren.“
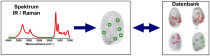
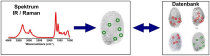
Durch Nutzung der Fourier-Transform-Infrarot-Spektroskopie und des Raman-Imagings gewinnt das RUB-Team einen „spektralen Fingerabdruck“. Dieser spiegelt den momentanen Zustand der Zelle auf Proteinebene wieder. Durch Vergleich mit einer „Kartei“ kann so ein „Fingerabdruck“ dem zugehörigen „Verdächtigen“, zum Beispiel einem Darmtumor, zugeordnet werden.
© RUB, Bild: LS Biophysik
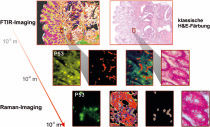
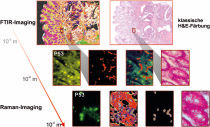
Fourier-Transform-Infrarot (FTIR)- und Raman-Imaging-Analysen von Gewebeschnitten eines kolorektalen Karzinoms (Darmtumor). Die Vergrößerung der Bilder nimmt von oben nach unten zu. Auf der rechten Seite ist der klassisch gefärbte H&E-Schnitt zu sehen (Hämatoxylin-Eosin-Färbung). Mit FTIR-Imaging (links) ist eine Auflösung bis zehn Mikrometer möglich; die spektrale Histopathologie zeigt Veränderungen in den Krypten (rot), also Einstülpungen des Darms, die eine immunohistochemische Fluoreszenzfärbung (P53) bestätigt. Um die Veränderungen einer Krypte detaillierter zu analysieren, setzten die Forscher Raman-Imaging mit höherer räumlicher Auflösung ein (untere Zeile). Die veränderten Zellkerne (grau) in den veränderten Krypten (rot) können hier im Vergleich zu einer Fluoreszenzfärbung (P53, grün) identifiziert werden.
© RUB, Bild: LS Biophysik
Diagnose Dickdarmkrebs
Die Diagnose Dickdarmkrebs stellen Pathologen zurzeit, indem sie gefärbte dünne Gewebeschnitte aus einer Biopsie unter dem Mikroskop begutachten. Das geschieht in der Regel erst in einem fortgeschrittenen Stadium, und das Verfahren liefert keine Informationen über die molekularen Ursachen des Tumors. Die am RUB-Lehrstuhl für Biophysik etablierte Spektrale Histopathologie (SHP)-Methode erfasst hingegen direkt molekulare Veränderungen im Gewebe, insbesondere Proteinveränderungen. Sie funktioniert ohne Marker wie zum Beispiel Fluoreszenzfarbstoffe. Veränderungen detektiert sie schon in frühen Tumorstadien. Da die Analyse mit Lichtstrahlen erfolgt, kann man sie nicht nur auf dünne Gewebeschnitte aus Biopsien anwenden, sondern mit Hilfe von Lichtleitern auch direkt das Gewebe an der zu untersuchenden Stelle analysieren. „In Zukunft wollen wir die Spektrale Histopathologie gemeinsam mit klinischen Partnern endoskopisch, also direkt am Patienten einsetzen“, so Klaus Gerwert.
So funktioniert die Spektrale Histopathologie
Für die SHP zeichnen Forscher ortsaufgelöst Vibrationsspektren des Gewebes mit einem Infrarot- oder Raman-Mikroskop auf. Ein Vibrationsspektrum reflektiert den Zustand aller Proteine im Gewebe an der gemessenen Stelle. Verändern sich die Proteine im Gewebe aufgrund von Krebs, wandelt sich auch das zugehörige Spektrum. Jedes Spektrum ist dabei so charakteristisch für die Proteinveränderung wie ein Fingerabdruck für eine Person. Für ein einzelnes Gewebebild werden insgesamt rund zehn Millionen Infrarot-Spektren aufgenommen. Mit aufwendigen bioinformatischen Bildanalyseverfahren vergleichen die Wissenschaftler diese Spektren mit einer in PURE entwickelten Datenbank von Spektren bereits bekannter Gewebe und Tumore. Jedem Spektrum ordnet das Analyseprogramm einen in der Datenbank hinterlegten Gewebetypen zu, dargestellt durch eine bestimmte Farbe – genauso wie ein Täter durch Abgleich mit einer Datenbank anhand seines Fingerabdrucks identifiziert werden kann. Daraus ergibt sich ein ortsaufgelöstes annotiertes Bild des Darmgewebeschnitts. Die beiden PURE-Mitglieder Prof. Andrea Tannapfel, Direktorin des Instituts für Pathologie der RUB, und Prof. Dr. Axel Mosig, Leiter der Bioinformatik am Lehrstuhl Biophysik, waren maßgeblich daran beteiligt, die Datenbank und den Auswertalgorithmus zu erstellen. Das Auswertprogramm läuft mittlerweile auf einem handelsüblichen Laptop.
Vergleich mit klassischen Methoden zur Tumorerkennung
Um die Sensitivität und Spezifizität der Spektralen Histopathologie zu prüfen, verglich das RUB-Team die SHP-Ergebnisse mit klassischen immunohistochemischen Verfahren, bei denen Tumore durch Fluoreszenzmarker identifiziert werden. „Die Ergebnisse stimmten exzellent überein. Das zeigt eindrucksvoll, dass die Spektrale Histopathologie Änderungen der Gewebezusammensetzung hoch sensitiv und automatisiert nachweisen kann“, sagt Prof. Gerwert. Die Sensitivität und Spezifität der SHP liegen bereits über 95 Prozent und soll möglichst nah an 100 Prozent geführt werden. Durch Erweiterung auf das Raman-Imaging erzielte das RUB-Team eine höhere räumliche Auflösung im Vergleich zum Infrarot-Imaging, allerdings auf Kosten einer längeren Messzeit. „Beide Methoden ergänzen sich hervorragend“, so Klaus Gerwert. „Die Infrarot-Spektroskopie gibt schnell einen Überblick über den gesamten Gewebeschnitt. Mit Raman-Imaging können wir dann verdächtige Regionen genauer analysieren.“ Die Raman-Analyse detektiert etwa veränderte Zellkerne, die für Tumore charakteristisch sind.





















































