Neue Struktur für die selektive Sequenzerkennung
Verbesserte Basenpaarung für DNA
Anzeigen
Der hochspezifische Nachweis von genetischem Material ist für die moderne Medizin, die Forensik und die Biologie eine wichtige Aufgabe. Manche DNA-Sequenzen lassen sich jedoch nur schlecht nachweisen, weil ihre Kernbasen schwache Basenpaare ausbilden. Insbesondere die Kernbase Adenin ("A" im genetischen Alphabet) bindet nur schwach an die komplementäre Base Thymin (T). Marco Minuth und Prof. Clemens Richert vom Institut für Organische Chemie der Universität Stuttgart haben nun einen chemischen Ersatz für das Thymin gefunden, der eine stärkere und selektivere Basenpaarung mit Adenin eingeht. Dadurch gelingt es, ungewollte Basenkombinationen in der Doppelhelix wirkungsvoller zu unterdrücken und Fehlinterpretationen bei genetischen Analysen leichter zu vermeiden.
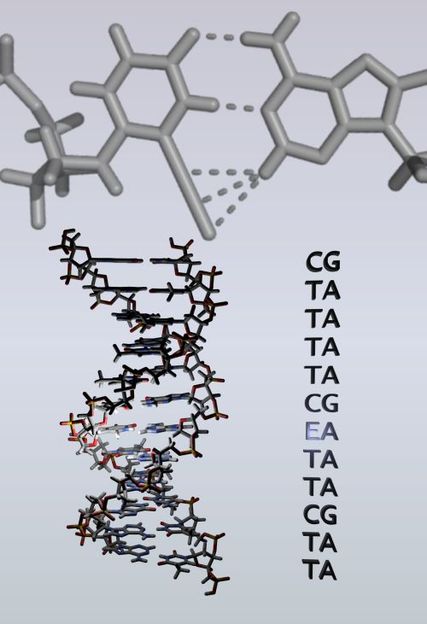
Basenpaarung des neu entwickelten Nucleosids mit Adenin. Die verschiedenen Wechselwirkungen sind durch gestrichelte Linien angedeutet. Links: Doppelhelix, die das neue Basenpaar enthält. Rechts: Sequenz der gezeigten Doppelhelix.
M. Minuth
Man könnte meinen, dass die Natur die Basenpaarung zwischen zwei Strängen der DNA im Laufe der Evolution erschöpfend optimiert hat. Tatsächlich ist es aber so, dass die DNA in der Zelle andere Anforderungen erfüllen muss als bei diagnostischen Tests. So ist es in der Zelle notwendig, dass die Doppelhelix in bestimmten Bereichen besonders leicht zu öffnen ist, damit die genetische Information von diesem Punkt an abgelesen werden kann. Dies ist zum Beispiel unmittelbar vor dem Startcodon eines Gens (d.h. dem Sequenzabschnitt unmittelbar vor einem Leseabschnitt) der Fall, wo die beiden Stränge getrennt werden müssen, bevor einer der beiden Stränge enzymatisch abgelesen werden kann. Hier sind also schwache Basenpaare von Vorteil.
Bei diagnostischen Tests, die ohne Enzyme auskommen, erfolgt die Trennung der Stränge dagegen chemisch oder durch Aufheizen. Der nachfolgende Nachweis wird schwierig, wenn in dem nachzuweisenden Sequenzabschnitt nur schwache Duplexe mit Sondensträngen gebildet werden können. Gerade auf modernen DNA-"Chips", auf denen viele verschiedene Sequenzen gleichzeitig nachgewiesen werden sollten, ergeben sich deshalb viele "falsch negative" Auslese-Ergebnisse.
Die Gruppe von Prof. Richert von der Universität Stuttgart fand nun eine Struktur, die mit der schwer zu bindenden Kernbase Adenin nicht nur die zwei Wasserstoffbrücken bildet, die aus den von Watson und Crick beschriebenen Basenpaaren bekannt sind, sondern auch zusätzliche molekulare Kontakte eingeht. Die Struktur des Adenins wird mit einem Substituenten, der von Acetylen (einem technischen Gas) abgeleitet ist, zusätzlich "abgegriffen". Dadurch wird die Basenpaarung stärker und spezifischer. „Das ist ein wenig so, als hätte man den Bart eines Schlüssel verlängert, um eine höhere Spezifität bei der Erkennung eines Schlosses zu erreichen“, erklären die Stuttgarter Chemiker. „Dadurch gelingt es, falsche Basenkombinationen in der Doppelhelix wirkungsvoller zu unterdrücken.“
Der neue Ersatzstoff für Thymin ist ein sogenanntes "C-Nucleosid", das eine besonders feste Bindung zwischen Kernbase und Zucker besitzt. Eine Synthese für diese Substanz zu entwickeln, erwies sich als eine echte Herausforderung, die insgesamt über zehn Jahre Forschung erforderte. Nachdem die ersten kurzen DNA-Sondenstränge hergestellt werden konnten, ergaben die Messungen, dass sich das verbesserte Basenpaar sowohl beim Binden an DNA-Zielstränge als auch beim Binden an RNA-Zielstränge ausbildet. Damit hat die neue Kernbase, die die Forscher "E" (von Ethinylpyridon) nennen, das Potential, Einzug in viele diagnostische und forensische Tests zu halten.





















































