Einblick in die Kinderstube der Proteine
Anzeigen
Proteine transportieren Sauerstoff, bekämpfen Krankheitserreger oder dienen als Baustoff für Muskeln und Sehnen. Damit sie ihre Funktion erfüllen können, müssen sie eine komplexe räumliche Struktur einnehmen. Dabei werden sie von Helferproteinen, den Chaperons, unterstützt. Einem internationalen Wissenschaftlerteam ist es erstmals gelungen, ein Zwischenstadium der Faltung im Hohlraum des weitverbreiteten Chaperons GroEL zu analysieren.
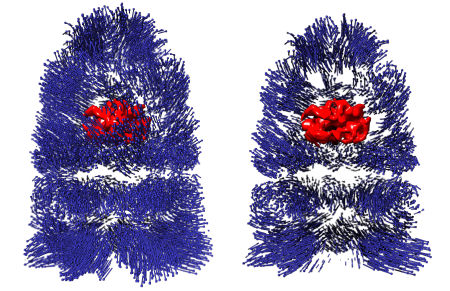
Proteinfaltung im geschlossenen GroEL-GroES-Komplex: Die blauen Pfeile zeigen die Bewegung des umhüllenden Helferproteins während der Faltung des Substratproteins (rot) in seinem Innern an.
Forschungszentrum Jülich
Proteine sind als lange Ketten aufgebaut, die in der Regel aus einigen Hundert aneinandergereihten Aminosäurebausteinen bestehen. Wie sie es schaffen, innerhalb von Sekundenbruchteilen die korrekte Gestalt anzunehmen, ist immer noch ein großes Rätsel. Fehlfaltungen können schwerwiegende Folgen haben, Mediziner führen heute ein breites Spektrum unterschiedlicher Erkrankungen auf missgestaltete Proteine zurück, neben Krebs, Diabetes oder Arterienverkalkung auch viele neurodegenerative Erkrankungen wie Alzheimer oder Parkinson.
Nicht alle Proteine sind in der Lage, sich selbstständig korrekt im Raum zu verknäueln. Helferproteine, Chaperons – so die französischstämmige Bezeichnung für „Anstandsdame“ – unterstützen sie dabei, bestimmte Abschnitte der Aminosäurekette zu charakteristischen, spiralförmigen Helices zusammenzurollen oder in ziehharmonikaförmige Faltblatt-Strukturen zu legen. Auf diese Weise wird ein Großteil der hydrophoben, also der wasserabweisenden, Enden ins Molekülinnere gekehrt. So wird verhindert, dass sich die Proteine über diese hydrophoben Enden vorzeitig miteinander verbinden und zusammenklumpen.
GroEL ist eines der am weitesten verbreiteten Chaperons, das in fast allen bisher analysierten Bakterienarten vorkommt. Nahe Verwandte sorgen auch in Mehrzellern dafür, unfertige Proteine in die richtige Form zu bringen. Das Riesenmolekül besteht aus zwei Ringen, die zusammen eine Art Röhre bilden, in der sich das unfertige Proteinsubstrat ungestört zusammenlegen kann. Um die Aushöhlung während des Faltvorgangs nach außen abzudichten, kommt ein weiteres Helferprotein hinzu, das Co-Chaperon GroES, das sich wie ein Deckel auf die Öffnung legt.
„Bislang war unbekannt, welche molekularen Vorgänge für diese Verkapselung verantwortlich sind und wie das Helferprotein die Proteinfaltung im Innern konkret beeinflusst. Die neuen Erkenntnisse könnten dazu beitragen, diese Fragen besser zu verstehen“, freut sich Prof. Dieter Willbold, Leiter des Jülicher Forschungsbereichs Strukturbiochemie am Institute of Complex Systems (ICS-6), über die Ergebnisse aus seinem Institutsbereich. Die elektronenmikroskopischen Untersuchungen zeigen erstmals, an welcher Stelle ein noch unvollständig gefaltetes Protein andockt, wenn sich der Deckel des GroEL-GroES-Komplexes schließt.
„Wir konnten nachweisen, dass die Anbindung des Proteins die symmetrische Anordnung der Untereinheiten zerstört, aus denen GroEL zusammengesetzt ist“, erläutert Jun.-Prof. Gunnar Schröder, der die in Cell erschienene Studie gemeinsam mit Forschern aus den USA verfasst hat. Wissenschaftler hatten lange nach einem Mechanismus gesucht, der es GroEL ermöglicht, sowohl das unfertige Proteinsubstrat in seinem Innern als auch den „molekularen Deckel“ GroES anzubinden. „Es hat sich gezeigt, dass die Proteinfaltung durch eine Formänderung von GroEL eingeleitet wird“, so Schröder, Leiter der am ICS-6 angesiedelten Jülicher Nachwuchsgruppe Computational Structural Biology. Durch die Verformung vergrößert sich wiederum der Hohlraum im Innern des Helferproteins – möglicherweise, um mehr Platz für die Proteinfaltung zu schaffen.
Zur Untersuchung der Proteinstruktur kam die sogenannte Cryo-Elektronenmikroskopie zum Einsatz. Die Methode bietet den Vorteil, dass Proteine oder Viren nur verhältnismäßig einfach mit flüssigem Ethan heruntergekühlt werden müssen. Nachteil: Die erzielbare Auflösung von etwas weniger als einem Nanometer reicht in vielen Fällen nicht aus, um die atomare Struktur der untersuchten Makromoleküle zuverlässig zu rekonstruieren. Bei der Auswertung der Daten setzten die Wissenschaftler daher auf ein neues Korrekturverfahren.
Das zugrunde liegende Kreuzvalidierungsverfahren ermöglicht es, die mit dem Elektronenmikroskop gewonnenen Daten mit bereits bekannten Informationen über die Molekülstruktur abzugleichen. Das Verfahren ist nicht ganz neu. Es hat sich seit über 20 Jahren in der Röntgenstrukturanalyse bewährt, mit der sich atomare Strukturen von deutlich höherer Qualität bestimmen lassen. Die Untersuchung mit Röntgenstrahlung ist allerdings auf Kristallgitterstrukturen beschränkt. Für die Versuche müssen daher zunächst Kristalle aus Proteinen herangezüchtet werden – ein schwieriges, oft auch unmögliches Unterfangen, das bei der Elektronenmikroskopie entfällt.
Originalveröffentlichung
Dong-Hua Chen, Damian Madan, Jeremy Weaver, Zong Lin, Gunnar F. Schröder, Wah Chiu, Hays S. Rye: Visualizing GroEL/ES in the Act of Encapsulating a Folding Protein. Cell 153, 1354–1365, June 6, 2013.
Benjamin Falkner, Gunnar F. Schröder: Cross-validation in cryo-EM–based structural modeling. PNAS 2013 (published ahead of print: May 14, 2013)