Atomare Struktur eines Staphylokokken-Bakteriophagen mittels Kryo-Elektronenmikroskopie sichtbar gemacht
Forscher der University of Alabama at Birmingham haben mit Hilfe der Kryo-Elektronenmikroskopie die Struktur eines bakteriellen Virus in noch nie dagewesener Detailgenauigkeit aufgeklärt. Dies ist die erste Struktur eines Virus, das Staphylococcus epidermidis infizieren kann, und die hochauflösende Kenntnis der Struktur ist ein wichtiges Bindeglied zwischen der Virusbiologie und dem potenziellen therapeutischen Einsatz des Virus zur Bekämpfung von bakteriellen Infektionen.
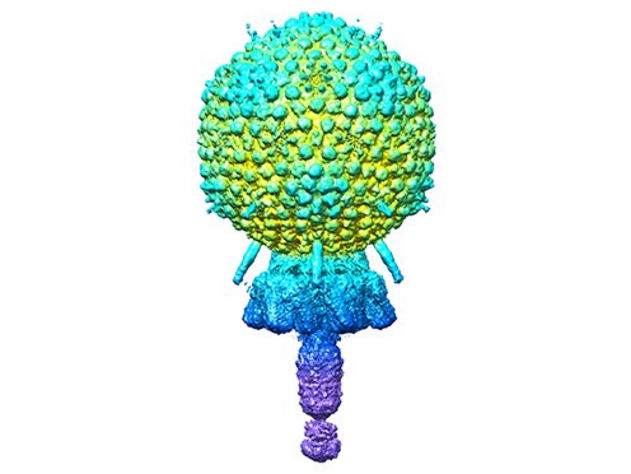
Der Andhra-Phage
UAB, Dokland lab
Bakteriophagen oder "Phagen" ist die Bezeichnung für Viren, die Bakterien infizieren. Die UAB-Forscher unter der Leitung von Terje Dokland, Ph.D., in Zusammenarbeit mit Asma Hatoum-Aslan, Ph.D., von der University of Illinois Urbana-Champaign, haben atomare Modelle für alle oder Teile von 11 verschiedenen Strukturproteinen des Phagen Andhra beschrieben. Die Studie ist in Science Advances veröffentlicht.
Andhra ist ein Mitglied der Gruppe der Picoviren. Sein Wirtsspektrum ist auf S. epidermidis beschränkt. Dieses Hautbakterium ist meist gutartig, aber auch eine der Hauptursachen für Infektionen von medizinischen Verweilgeräten. "Picoviren sind in Phagensammlungen nur selten zu finden und werden für therapeutische Anwendungen nicht ausreichend untersucht und genutzt", so Hatoum-Aslan, Phagenbiologe an der Universität von Illinois.
Mit dem Auftreten von Antibiotikaresistenzen bei S. epidermidis und dem verwandten Erreger Staphylococcus aureus ist das Interesse der Forscher an einer möglichen Verwendung von Bakteriophagen zur Behandlung bakterieller Infektionen wieder gestiegen. Picoviren töten die Zellen, die sie infizieren, immer ab, nachdem sie sich an die bakterielle Zellwand gebunden, diese enzymatisch durchbrochen, die Zellmembran durchdrungen und virale DNA in die Zelle injiziert haben. Sie haben auch andere Eigenschaften, die sie zu attraktiven Kandidaten für den therapeutischen Einsatz machen, darunter ein kleines Genom und die Unfähigkeit, bakterielle Gene zwischen Bakterien zu übertragen.
Die Kenntnis der Proteinstruktur von Andhra und das Verständnis der Art und Weise, wie diese Strukturen es dem Virus ermöglichen, ein Bakterium zu infizieren, wird es ermöglichen, durch genetische Manipulation maßgeschneiderte Phagen für einen bestimmten Zweck herzustellen.
"Die strukturelle Grundlage für die Wirtsspezifität von Phagen, die S. aureus und S. epidermidis infizieren, ist noch wenig bekannt", sagte Dokland, Professor für Mikrobiologie an der UAB und Direktor des UAB Cryo-Elektronenmikroskopiezentrums. "Mit der vorliegenden Studie haben wir ein besseres Verständnis der Strukturen und Funktionen der Andhra-Genprodukte und der Determinanten der Wirtsspezifität gewonnen, was den Weg für ein rationaleres Design von maßgeschneiderten Phagen für therapeutische Anwendungen ebnet. Unsere Ergebnisse geben Aufschluss über kritische Merkmale für den Zusammenbau des Virions, die Erkennung des Wirts und die Penetration".
Staphylokokken-Phagen haben in der Regel ein enges Spektrum an Bakterien, die sie infizieren können, abhängig von den variablen Wandpolymeren der Teichosäure auf der Oberfläche der verschiedenen Bakterienstämme. "Dieser enge Wirtsbereich ist ein zweischneidiges Schwert: Einerseits ermöglicht es den Phagen, nur den spezifischen Krankheitserreger anzugreifen, andererseits bedeutet es, dass der Phage in jedem einzelnen Fall auf den Patienten zugeschnitten werden muss", so Dokland.
Die allgemeine Struktur von Andhra besteht aus einem 20-flächigen, rundlich-ikosaedrischen Kapsidkopf, der das virale Genom enthält. Das Kapsid ist mit einem kurzen Schwanz verbunden. Der Schwanz ist hauptsächlich für die Bindung an S. epidermidis und das enzymatische Aufbrechen der Zellwand verantwortlich. Die virale DNA wird durch den Schwanz in das Bakterium injiziert. Zu den Segmenten des Schwanzes gehören das Portal vom Kapsid zum Schwanz, der Stamm, die Anhängsel, der Knubbel und die Schwanzspitze.
Die 11 verschiedenen Proteine, aus denen jedes Viruspartikel besteht, liegen in mehreren Kopien vor, die sich zusammenfügen. So besteht das Kapsid aus 235 Kopien von jeweils zwei Proteinen, und die anderen neun Virionproteine haben Kopienzahlen von zwei bis 72. Insgesamt besteht das Virion aus 645 Proteinteilen, darunter zwei Kopien eines zwölften Proteins, dessen Struktur mit dem Proteinstrukturvorhersageprogramm AlphaFold vorhergesagt wurde.
Die von Dokland, Hatoum-Aslan und den Co-Erstautoren N'Toia C. Hawkins, Ph.D., und James L. Kizziah, Ph.D., UAB Department of Microbiology, beschriebenen atomaren Modelle zeigen die Strukturen für jedes Protein - wie sie in molekularer Sprache wie Alpha-Helix, Beta-Helix, Beta-Strang, Beta-Fass oder Beta-Prisma beschrieben werden. Die Forscher haben beschrieben, wie jedes Protein an andere Exemplare desselben Proteintyps bindet, um beispielsweise die hexameren und pentameren Flächen des Kapsids zu bilden, und wie jedes Protein mit benachbarten verschiedenen Proteintypen interagiert.
Elektronenmikroskope verwenden einen Strahl beschleunigter Elektronen, um ein Objekt zu beleuchten, und bieten eine viel höhere Auflösung als ein Lichtmikroskop. Die Kryo-Elektronenmikroskopie fügt das Element der superkalten Temperaturen hinzu, was sie besonders nützlich für die Auflösung der nahezu atomaren Struktur von größeren Proteinen, Membranproteinen oder lipidhaltigen Proben wie membrangebundenen Rezeptoren und Komplexen aus mehreren Biomolekülen macht.
In den letzten acht Jahren haben neue Elektronendetektoren der Kryo-Elektronenmikroskopie einen enormen Auflösungssprung gegenüber der normalen Elektronenmikroskopie ermöglicht. Schlüsselelemente dieser so genannten "Auflösungsrevolution" für die Kryo-Elektronenmikroskopie sind:
- Blitzgefrieren wässriger Proben in flüssigem Ethan, das auf unter -256 Grad Celsius gekühlt wird. Anstelle von Eiskristallen, die die Proben stören und den Elektronenstrahl streuen, gefriert das Wasser zu einem fensterartigen "gläsernen Eis".
- Die Probe wird im Mikroskop bei sehr kalten Temperaturen gehalten, und es wird eine geringe Elektronendosis verwendet, um eine Beschädigung der Proteine zu vermeiden.
- Extrem schnelle Direktelektronendetektoren sind in der Lage, einzelne Atome mit Hunderten von Bildern pro Sekunde zu zählen, so dass die Bewegung der Probe während der Aufnahme korrigiert werden kann.
- Moderne Computer fügen Tausende von Bildern zusammen, um dreidimensionale Strukturen in hoher Auflösung zu erzeugen. Grafikverarbeitungseinheiten verarbeiten Terabytes an Daten.
- Der Mikroskoptisch, der die Probe hält, kann während der Aufnahme gekippt werden, so dass ein dreidimensionales tomographisches Bild entsteht, ähnlich wie bei einem CT-Scan im Krankenhaus.
Die Analyse der Struktur des Andhra-Virus durch die UAB-Forscher begann mit 230.714 Partikelbildern. Die molekulare Rekonstruktion von Kapsid, Schwanz, distalem Schwanz und Schwanzspitze begann mit 186.542, 159.489, 159.489 bzw. 159.489 Bildern. Die Auflösung reichte von 3,50 bis 4,90 Angström.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.