Clevere Kombination aus Quantenphysik und Molekularbiologie
Künstliche Intelligenz liefert unerreichte Einblicke in die Funktion von Biomolekülen
Eine neue Analysemethode liefert bislang unerreichbare Einblicke in die extrem schnelle Dynamik von Biomolekülen. Das Entwicklerteam um Abbas Ourmazd von der University of Wisconsin Milwaukee und Robin Santra von DESY stellt die clevere Kombination aus Quantenphysik und Molekularbiologie im Fachblatt „Nature“ vor. Die Forscherinnen und Forscher haben damit verfolgt, wie das photoaktive gelbe Protein (photoactive yellow protein, PYP) in weniger als einer billionstel Sekunde seine Struktur ändert, nachdem es durch Licht angeregt worden ist.
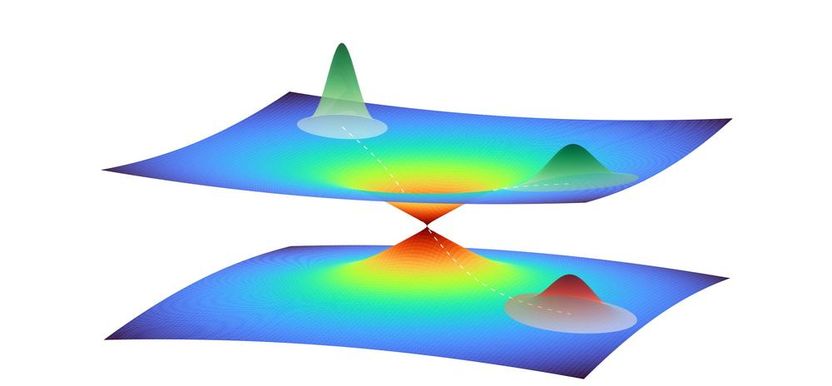
Illustration eines Quantenwellenpakets in unmittelbarer Nähe einer konischen Durchschneidung zweier Quantenzustände. Das Wellenpaket repräsentiert hierbei die kollektive Bewegung mehrerer Atome im photoaktiven gelben Protein. Ein Teil des Wellenpakets bewegt sich über die Durchschneidung von einer Potenzialfläche auf die andere. Der restliche Anteil verbleibt auf der oberen Fläche, und es entsteht eine Überlagerung der beiden Quantenzustände.
DESY, Niels Breckwoldt
„Um biochemische Vorgänge in der Natur wie beispielsweise die Photosynthese in bestimmten Bakterien genau zu verstehen, ist es wichtig, den detaillierten Ablauf zu kennen“, erläutert Santra die Motivation. „Wenn photoaktive Proteine von Licht getroffen werden, ändern sie ihre räumliche Struktur, und diese Strukturänderung bestimmt die Rolle, die ein Protein in der Natur übernimmt.“ Bislang ist es allerdings kaum möglich, den genauen Verlauf solcher Strukturänderungen zu verfolgen: Es lassen sich lediglich Anfangs- und Endzustand eines Moleküls vor und nach einer Reaktion bestimmen und theoretisch deuten. „Aber wie die Energie- und Formänderung dazwischen genau abläuft, wissen wir nicht“, sagt Santra. „Das ist, als könnten Sie sehen, dass jemand seine Hände gefaltet hat, aber Sie können nicht verfolgen, wie er die Finger dafür beugt.“
Während die Hand ausreichend groß und die Bewegung langsam genug ist, damit wir sie mit unseren Augen beobachten können, ist das im Reich der Moleküle nicht so einfach. Der Energiezustand eines Moleküls lässt sich sehr genau mit Hilfe der Spektroskopie bestimmen. Und zur Analyse der Form von Molekülen nutzen Forscherinnen und Forscher helles Röntgenlicht wie von einem Röntgenlaser. Dank seiner sehr kurzen Wellenlänge kann es sehr kleine räumliche Strukturen entschlüsseln, etwa die Positionen der Atome in einem Molekül. Allerdings entsteht dabei kein Abbild wie bei einem Foto, sondern ein charakteristisches Streumuster der Röntgenstrahlen, aus dem sich die räumliche Struktur berechnen lässt.
Kurze, helle Röntgenblitze
Da die Bewegung auf der Molekülebene extrem schnell ist, müssen die Wissenschaftlerinnen und Wissenschaftler äußerst kurze Röntgenblitze verwenden, weil die Aufnahme sonst verschmiert. Erst mit der Erfindung der Röntgenlaser ist es möglich, ausreichend helle und kurze Röntgenblitze zu produzieren, um diese Dynamik festzuhalten. Da sich die Moleküldynamik im Bereich der Quantenphysik abspielt, wo die aus dem Alltag vertrauten Gesetze der Physik nicht mehr gelten, lassen sich die Messungen ohne eine quantenphysikalische Analyse allerdings nicht verstehen.
Dabei gibt es eine Besonderheit der photoaktiven Proteine zu beachten: Durch das eingestrahlte Licht geht ihre Elektronenhülle in einen angeregten Quantenzustand über, der eine erste Formänderung des Moleküls bewirkt. Diese Formänderung wiederum kann zu einer Überschneidung des angeregten und des Grund-Quantenzustands führen. Die Folge ist ein Quantensprung vom angeregten zurück in den Grundzustand, wobei die geänderte Form des Moleküls zunächst bestehen bleibt. Die trichterförmige Überschneidung der Quantenzustände heißt wissenschaftlich konische Durchschneidung und öffnet einen Pfad zu einer neuen räumlichen Struktur des Proteins im quantenmechanischen Grundzustand.
Dem Team um Santra und Ourmazd ist es nun erstmals gelungen, die Strukturdynamik eines photoaktiven Proteins an einer solchen trichterförmigen Überschneidung zu enträtseln. Dabei griffen sie auf Hilfe durch maschinelles Lernen zurück. Denn für eine solche Beschreibung der Dynamik müssten eigentlich alle Bewegungsmöglichkeiten aller beteiligten Teilchen betrachtet werden. Das führt jedoch schnell zu unübersichtlichen, nicht mehr lösbaren Rechnungen.
Rund 6000 Dimensionen
„Das photoaktive gelbe Protein, das wir untersucht haben, besteht aus rund 2000 Atomen“, berichtet Santra, der Leitender Wissenschaftler bei DESY und Physikprofessor an der Universität Hamburg ist. „Da sich jedes Atom grundsätzlich in allen drei Raumdimensionen bewegen kann, gibt es insgesamt 6000 Bewegungsoptionen. Das führt zu einer quantenmechanischen Rechnung mit 6000 Dimensionen – und die ist selbst mit den heute leistungsstärksten Computern nicht lösbar.“
Per Computeranalyse basierend auf maschinellem Lernen ließen sich jedoch kollektive Bewegungsmuster der Atome in dem komplexen Molekül identifizieren. „Das ist wie beim Falten der Hände: Da betrachten wir auch nicht jedes Atom einzeln, sondern deren konzertierte Bewegung“, erklärt Santra. Anders als bei einer Hand, bei der die kollektiven Bewegungsmöglichkeiten offensichtlich sind, lassen sich diese bei den Atomen eines Moleküls nicht so einfach erkennen. Der Computer konnte die rund 6000 Dimensionen auf diese Weise jedoch auf vier reduzieren. Mit der Demonstration dieser neuen Methode kann Santras Team auch erstmals eine konische Durchschneidung von Quantenzuständen in einem komplexen Molekül aus Tausenden von Atomen charakterisieren.
Die detaillierte Rechnung zeigt, wie sich dieser konische Trichter in dem vierdimensionalen Raum bildet und das photoaktive gelbe Protein durch ihn nach einer Anregung mit Licht seine Struktur ändert und dabei wieder in den Ausgangsquantenzustand zurückfällt. Diesen Ablauf können die Wissenschaftlerinnen und Wissenschaftler nun in Schritten von einigen Dutzend Femtosekunden (billiardstel Sekunden) beschreiben und so zum Verständnis der photoaktiven Prozesse beitragen. „So liefert die Quantenphysik neue Einblicke in ein biologisches System, und die Biologie bringt neue Anregungen für die quantenmechanische Methodik“, sagt Santra, der auch Mitglied im Hamburger Exzellenzcluster „CUI: Advanced Imaging of Matter“ ist. „Beide Gebiete befruchten sich dabei gegenseitig.“
Originalveröffentlichung
Diese Produkte könnten Sie interessieren
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Proteinanalytik
Die Proteinanalytik ermöglicht einen tiefen Einblick in diese komplexen Makromoleküle, ihre Struktur, Funktion und Wechselwirkungen. Sie ist unerlässlich für die Entdeckung und Entwicklung von Biopharmazeutika, das Verständnis von Krankheitsmechanismen und die Identifizierung von therapeutischen Zielen. Durch Techniken wie Massenspektrometrie, Western Blot und Immunoassays können Forscher Proteine auf molekularer Ebene charakterisieren, ihre Konzentration bestimmen und mögliche Modifikationen identifizieren.

Themenwelt Proteinanalytik
Die Proteinanalytik ermöglicht einen tiefen Einblick in diese komplexen Makromoleküle, ihre Struktur, Funktion und Wechselwirkungen. Sie ist unerlässlich für die Entdeckung und Entwicklung von Biopharmazeutika, das Verständnis von Krankheitsmechanismen und die Identifizierung von therapeutischen Zielen. Durch Techniken wie Massenspektrometrie, Western Blot und Immunoassays können Forscher Proteine auf molekularer Ebene charakterisieren, ihre Konzentration bestimmen und mögliche Modifikationen identifizieren.