Bahnbrechende Methode offenbart dynamische Struktur bei HIV
Neue Technik kann Moleküle in Echtzeit bei Raumtemperatur verfolgen
Anzeigen
Viren sind beängstigend. Sie dringen wie unsichtbare Armeen in unsere Zellen ein, und jeder Typ bringt seine eigene Angriffsstrategie mit. Während Viren die Gemeinschaften von Menschen und Tieren vernichten, kämpfen die Wissenschaftler auf der Suche nach einem Gegenschlag. Viele setzen die Elektronenmikroskopie ein, ein Werkzeug, mit dem man "sehen" kann, was einzelne Moleküle im Virus tun. Doch selbst die ausgeklügeltste Technologie erfordert, dass die Probe eingefroren und immobilisiert wird, um die höchste Auflösung zu erhalten.
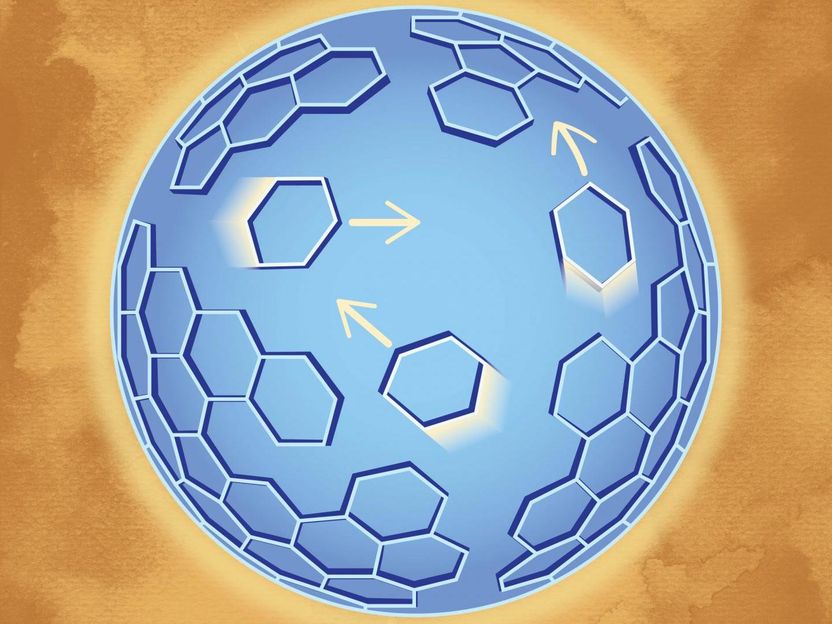
Eine künstlerische Darstellung von Proteinen aus Gag-Molekülen, die eine hexagonale Gitterstruktur bilden und über die virusähnlichen Partikel diffundieren.
Dave Meikle/Saffarian Lab
Nun haben Physiker der Universität von Utah Pionierarbeit geleistet, um virusähnliche Partikel in Echtzeit bei Raumtemperatur mit beeindruckender Auflösung abzubilden. In einer neuen Studie zeigt die Methode, dass das Gitter, das die Hauptstrukturkomponente des menschlichen Immunschwächevirus (HIV) bildet, dynamisch ist. Die Entdeckung eines diffundierenden Gitters aus Gag- und GagPol-Proteinen, das lange Zeit als völlig statisch galt, eröffnet potenzielle neue Therapien.
Wenn HIV-Partikel aus einer infizierten Zelle ausbrechen, erleiden die Viren eine Verzögerungszeit, bevor sie infektiös werden. Protease, ein Enzym, das als Halbmolekül in GagPol-Proteine eingebettet ist, muss sich in einem Prozess, der Dimerisierung genannt wird, an andere ähnliche Moleküle binden. Dies löst die Virusreifung aus, die zu infektiösen Partikeln führt. Niemand weiss, wie diese Halbprotease-Moleküle einander finden und sich dimerisieren, aber es könnte mit der Umordnung des Gitters zu tun haben, das von den Gag- und GagPol-Proteinen gebildet wird, die genau innerhalb der Virushülle liegen. Gag ist das wichtigste Strukturprotein und hat sich als ausreichend erwiesen, um virusähnliche Partikel zusammenzusetzen. Gag-Moleküle bilden eine hexagonale Gitterstruktur, die mit sich selbst verflochten ist, wobei winzige Lücken eingestreut sind. Die neue Methode zeigte, dass das Gag-Proteingitter nicht statisch ist.
"Diese Methode ist einen Schritt voraus, da sie Mikroskopie verwendet, die traditionell nur statische Informationen liefert. Zusätzlich zu den neuen Mikroskopiemethoden verwendeten wir ein mathematisches Modell und biochemische Experimente, um die Gitterdynamik zu verifizieren", sagte Hauptautorin Ipsita Saha, diplomierte Forschungsassistentin an der Fakultät für Physik und Astronomie der Universität. "Abgesehen vom Virus besteht eine wichtige Implikation der Methode darin, dass man sehen kann, wie sich Moleküle in einer Zelle bewegen. Damit kann man jede biomedizinische Struktur untersuchen."
Kartierung einer Nanomaschine
Die Wissenschaftler suchten zunächst nicht nach dynamischen Strukturen - sie wollten nur das Gag-Proteingitter untersuchen. Saha leitete die zweijährigen Bemühungen, Mikroskopietechniken zu "hacken", um Viruspartikel bei Raumtemperatur untersuchen zu können, um ihr Verhalten im wirklichen Leben zu beobachten. Die Größe des Virus ist winzig klein - etwa 120 Nanometer im Durchmesser - deshalb verwendete Saha die interferometrische photoaktivierte Lokalisierungsmikroskopie (iPALM).
Zunächst markierte Saha den Gag mit einem fluoreszierenden Protein namens Dendra2 und produzierte virusähnliche Partikel der resultierenden Gag-Dendra2-Proteine. Diese virusähnlichen Partikel sind die gleichen wie die HIV-Partikel, bestehen aber nur aus der Gag-Dendra2-Proteingitterstruktur. Saha zeigte, dass die resultierenden Gag-Dendra2-Proteine die virusähnlichen Partikel auf die gleiche Weise zusammensetzten, wie virusähnliche Partikel reguläre Gag-Proteine bildeten. Der fluoreszierende Aufsatz ermöglichte es iPALM, den Partikel mit einer Auflösung von 10 Nanometern abzubilden. Die Wissenschaftler fanden heraus, dass jedes immobilisierte virusähnliche Partikel 1400 bis 2400 Gag-Dendra2-Proteine enthielt, die in einem hexagonalen Gitter angeordnet waren. Als sie die iPALM-Daten verwendeten, um ein Zeitrafferbild des Gitters zu rekonstruieren, stellte sich heraus, dass das Gitter von Gag-Dendra2 im Laufe der Zeit nicht statisch war. Um dies sicherzustellen, verifizierten sie es unabhängig voneinander auf zwei Arten: mathematisch und biochemisch.
Zuerst teilten sie das Proteingitter in gleichförmige separate Segmente auf. Mit Hilfe einer Korrelationsanalyse testeten sie, wie jedes Segment mit sich selbst über einen Zeitraum von 10 bis 100 Sekunden korrelierte. Wenn jedes Segment weiterhin mit sich selbst korrelierte, waren die Proteine stationär. Wenn sie die Korrelation verloren, waren die Proteine diffundiert. Sie stellten fest, dass die Proteine im Laufe der Zeit recht dynamisch waren.
Die zweite Art, wie sie das dynamische Gitter verifizierten, war biochemisch. Für dieses Experiment schufen sie virusähnliche Partikel, deren Gitter zu 80% aus Gag-Wildtyp-Proteinen, zu 10% aus mit SNAP markiertem Gag und zu 10% aus mit Halo markiertem Gag bestand. SNAP und Halo sind Proteine, die einen Linker binden können, der sie für immer aneinander bindet. Die Idee war, festzustellen, ob die Moleküle im Proteingitter stationär blieben oder ob sie ihre Positionen wechselten.
"Die Gag-Proteine setzen sich zufällig zusammen. Die SNAP- und Halo-Moleküle können sich überall innerhalb des Gitters befinden - einige können nahe beieinander liegen, und einige werden weit entfernt sein", sagte Saha. "Wenn sich das Gitter verändert, besteht die Möglichkeit, dass sich die Moleküle nahe beieinander befinden", so Saha.
Saha führte ein Molekül namens Haxs8 in die virusähnlichen Partikel ein. Haxs8 ist ein Dimerisierer - ein Molekül, das SNAP- und Halo-Proteine kovalent bindet, wenn sie sich innerhalb eines Bindungsradius voneinander befinden. Wenn sich SNAP- oder Halo-Moleküle nebeneinander bewegen, bilden sie einen dimerisierten Komplex. Sie verfolgte diese Konzentrationen von dimerisierten Komplexen über die Zeit. Wenn sich die Konzentration änderte, würde dies darauf hinweisen, dass neue Molekülpaare einander gefunden haben. Wenn sich die Konzentration verringerte, würde dies darauf hinweisen, dass die Proteine auseinander gebrochen sind. In jedem Fall würde dies darauf hinweisen, dass eine Bewegung stattgefunden hat. Sie stellten fest, dass mit der Zeit der Prozentsatz des dimerisierten Komplexes zunahm; die Proteine HALO und SNAP Gag bewegten sich überall im Gitter und kamen mit der Zeit zusammen.
Ein neues Werkzeug zur Untersuchung von Viren
Dies ist die erste Studie, die zeigt, dass die Protein-Gitterstruktur eines umhüllten Virus dynamisch ist. Dieses neue Werkzeug wird wichtig sein, um die Veränderungen innerhalb des Gitters besser zu verstehen, die auftreten, wenn neue Viruspartikel von unreif bis gefährlich infektiös werden.
"Was sind die molekularen Mechanismen, die zu einer Infektion führen? Damit eröffnet sich eine neue Studienrichtung", sagte Saha. "Wenn Sie diesen Prozess herausfinden können, können Sie vielleicht etwas tun, um zu verhindern, dass sie sich gegenseitig finden, wie eine Art Medikament, das das Virus in seiner Spur stoppen würde.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.























































