Wie gut sind Vorhersageprogramme für Proteinstörungen eigentlich?
Unordnung in Proteinen ist entscheidend für die biologische Funktion, und strukturelle Störungen der Proteine sind allgegenwärtiger, als Sie vielleicht denken. Proteine mit ungeordneten Regionen können klebrig sein und innerhalb und zwischen den Zellen verklumpen. Sie sind direkt an einer Reihe von neurodegenerativen Erkrankungen beteiligt. Daher ist es sehr wichtig, ungeordnete Regionen in Proteinen identifizieren zu können.
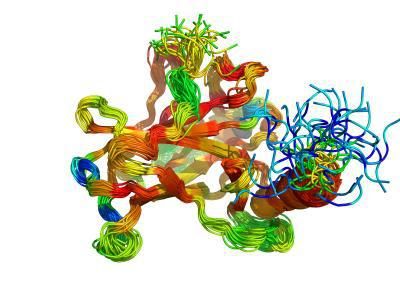
Die AU-Forscher Jakob T. NIelsen und Frans Mulder präsentieren Analysen mit NMR-Daten als Maßstab für den Test von Programmen zur Vorhersage von Proteinstörungen. Die Abbildung zeigt die NMR-Ensemblestruktur für die Kerndomäne des Proteins p53, gefärbt nach CheZOD Z-Scores.
Dr. Jakob Toudahl and Assoc. Prof. Frans Mulder at Department of Chemistry and Interdisciplinary Nanoscience Center, Aarhus University
Leider ist es schwierig und zeitaufwendig, die strukturellen Neigungen von Polypeptiden experimentell zu charakterisieren, und deshalb sind bioinformatische Methoden zur Vorhersage von Proteinstörungen aus der Sequenz unerlässlich.
In den letzten Jahren haben viele Bioinformatiker daher Algorithmen konstruiert, um diejenigen Peptidsequenzen, die sich falten, von denen zu unterscheiden, die sich nicht falten. Diese Algorithmen können auf verschiedenen "Merkmalen" basieren, die sich aus physikalisch-chemischen Parametern (wie Ladung oder Hydrophobie einer Aminosäure) ableiten und die evolutionäre Verwandtschaft betrachten.
Nun, da viele solcher Vorhersageprogramme verfügbar geworden sind, ist es von offensichtlichem Wert, eine Art Benchmark zu haben, um die Vorhersagen zu validieren und zu testen. Um dieses Dilemma zu lösen, generierten und validierten Nielsen und Mulder einen repräsentativen experimentellen Benchmarking-Satz von ortspezifischen und kontinuierlichen Störungen unter Verwendung von hinterlegten chemischen NMR-Shift-Daten für mehr als hundert ausgewählte Proteine. Anschließend analysierten sie die Leistung von 26 weit verbreiteten Methoden zur Vorhersage von Störungen und fanden heraus, dass diese deutlich variieren.
Der in ihrer Forschung vorgestellte Vergleich soll Proteinwissenschaftlern auf der ganzen Welt helfen, besser informierte Entscheidungen darüber zu treffen, welche Programme am besten geeignet sind.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

Antibody Stabilizer von CANDOR Bioscience
Protein- und Antikörperstabilisierung leicht gemacht
Langzeitlagerung ohne Einfrieren – Einfache Anwendung, zuverlässiger Schutz

DynaPro NanoStar II von Wyatt Technology
NanoStar II: DLS und SLS mit Touch-Bedienung
Größe, Partikelkonzentration und mehr für Proteine, Viren und andere Biomoleküle

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.