Mit der Histon-Angel im Protein-Teich fischen – wie entscheiden Zellen über ihre DNA-Reparatur?
Neue Möglichkeiten für Krebstherapien?
Nimmt die DNA im Zellkern Schaden, so können unsere Zellen auf verschiedene Möglichkeiten zurückgreifen, diesen zu reparieren. Auf welcher molekularen Grundlage eine Zelle zwischen den Reparaturwegen auswählt, klärt eine aktuelle Studie in ‚Nature Cell Biology‘, an der Wissenschaftler des Helmholtz Zentrums München maßgeblich beteiligt waren. Der Trick dabei: Sie hatten sich eine molekulare Angel gebaut, um die beteiligten Proteine sprichwörtlich aus dem Zellkern herauszufischen. Darauf aufbauend könnten sich langfristig neue Möglichkeiten für Krebstherapien ergeben, so die Autoren.
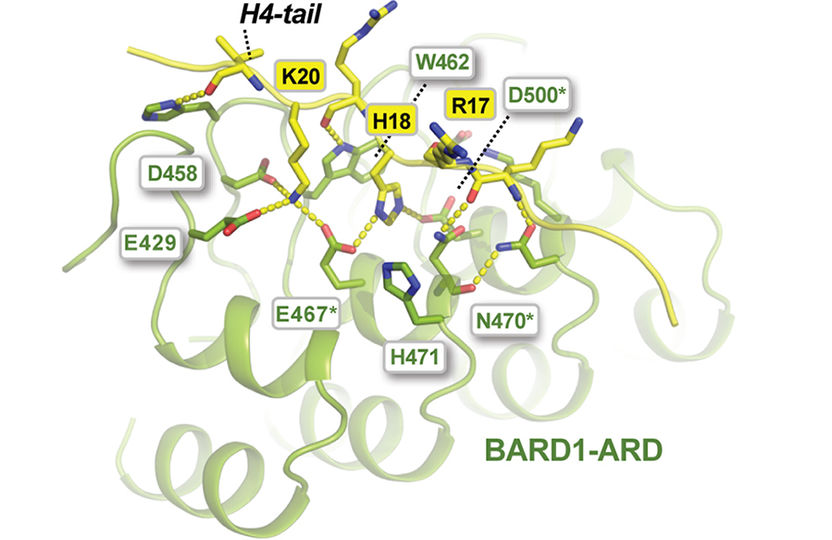
Die Entscheidung über den Reparaturweg fällt am Molekül BARD1
© Helmholtz Zentrum München
Die Abfolge der Basenpaare in der DNA enthält die genetische Information aller Lebewesen. Allerdings sind nicht alle Gene zur selben Zeit aktiv – denn das hängt auch von anderen Faktoren ab, die sich um die DNA herum abspielen. Einen wesentlichen Anteil daran haben die sogenannten Histone. „Histone sind Proteine, um die unsere DNA gewickelt ist und die so das Erbgut verpacken“, erklärt corresponding author Dr. Till Bartke, stellvertretender Direktor des Instituts für Funktionale Epigenetik (IFE) am Helmholtz Zentrum München. „Je nachdem wie die Histone chemisch verändert sind, können sie ganz unterschiedlich auf die DNA einwirken und die Genaktivität steuern.“ Herauszufinden, wie dieser sogenannte ‚Histon-Code’ funktioniert, das ist das große Ziel von Bartke und dem IFE.
Dazu rekonstruieren die Forscher bekannte chemische Histon-Modifikationen im Reagenzglas und untersuchen in einem Zellkernextrakt, welche Proteine jeweils daran binden. „Vereinfach gesagt bauen wir uns eine molekulare Angel mit definierten Histonen als Köder und gehen damit im Zellkern fischen – und dann wird es spannend zu sehen, wer da so alles anbeißt“, so Bartke.
DNA-Reparatur im Blick
In der aktuellen Arbeit suchten Bartke und sein Team mit dieser Methode nach Proteinen, die speziell an neu gebildetes Chromatin binden. „Die Idee war, Proteine zu identifizieren, die zwischen altem und neuem Chromatin unterscheiden können und daher mit der Verdopplung und Reparatur der DNA im Zusammenhang stehen“, so der Molekularbiologe. Dabei fanden die Forscher eine Gruppe von strukturell verwandten Enzymkomplexen, die an der Entscheidung der Zelle beteiligt sind, welchen DNA-Reparaturmechanismus sie auswählt. In weiteren Untersuchungen klärten dänische Kollegen um Anja Groth vom Biotech Research and Innovation Centre (BRIC) der Universität Kopenhagen im Detail, welche Proteine und Strukturelemente zur Entscheidung der Zelle über die unterschiedlichen DNA-Reparaturwege führen.
Ansatz für die Krebstherapie?
Zentrales Element bei der Reparatur bildet demnach ein Enzymkomplex um BRCA1 (abgekürzt für ‚Breast Cancer 1’), ein Gen, welches mittlerweile große Bekanntheit als Risikomarker für Brustkrebs erlangt hat. „Trägerinnen von Mutationen im BRCA1-Gen haben ein erhöhtes Risiko, an Brust- oder Eierstockkrebs zu erkranken, da bei ihnen die Reparatur von DNA-Schäden beeinträchtigt ist und sich dadurch weitere schädliche Mutationen anhäufen können. Krebspatientinnen, die Mutationen im BRCA1-Gen tragen, können jedoch mit sogenannten PARP-Inhibitoren behandelt werden - eine Wirkstoffklasse, die in diesen Patientinnen eine besonders starke Wirkung entfaltet“, erklärt Till Bartke.
Im Rahmen der Publikation hatten daraufhin britische Wissenschaftler um Ross Chapman vom Wellcome Centre For Human Genetics in Oxford gezielt die Auswirkungen von Mutationen im BRCA1-Komplex untersucht. Im Fokus stand dabei der Bereich, der für die Erkennung des neu gebildeten Chromatins verantwortlich ist. Zellen, bei denen dieser Bereich inaktiviert war, wurden darin äußerst effektiv von einem PARP-Inhibitor abgetötet.
„Durch die Zusammenarbeit unserer drei Forschungsgruppen kennen wir nun die genaue Funktionsweise dieser wichtigen DNA-Reparaturproteine und haben mit der Chromatin-Bindestelle des BRCA1-Komplexes bereits eine Zielstruktur ermittelt, an der wir medikamentös eingreifen und Krebspatienten therapieren können. Mithilfe kleiner Moleküle wäre es beispielsweise möglich, die BRCA1-Mutation quasi nachzuahmen und dadurch PARP-Inhibitoren auch für ein breiteres Patientenspektrum ohne diese Mutation nutzbar zu machen.“ Ein vielversprechender Anfang sei gemacht, so Bartke, und Firmen seien schon dabei, solche Moleküle zu entwickeln.