Eindeutig uneindeutig: Eine Hefe bricht mit der wichtigsten Regel des genetischen Codes
In unserer DNA sind die Baupläne des Lebens festgeschrieben. Man spricht vom „genetischen Code“, nach dessen Anweisung lebende Zellen Aminosäuren aneinanderfügen und so Proteine herstellen. Wie jeder andere Code ist der genetische Code eindeutig: Eine bestimmte DNA-Sequenz wird immer in dieselbe Aminosäure übersetzt. So dachte man bisher. Wissenschaftler am Max-Planck-Institut (MPI) für biophysikalische Chemie in Göttingen und an der University of Bath (England) berichten nun erstmals über ein Lebewesen, das mit dieser Regel bricht: Die Hefe Ascoidea asiatica übersetzt eine bestimmte DNA-Sequenz zufällig in zwei verschiedene Aminosäuren.
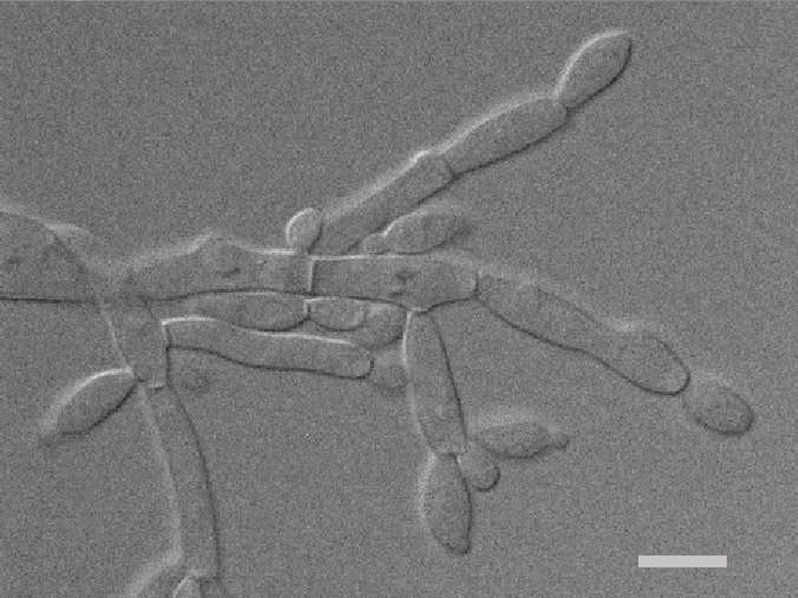
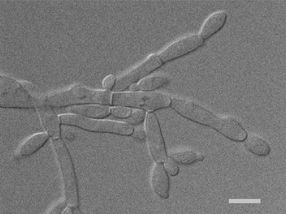
Die Hefe Ascoidea asiatica unter dem Mikroskop. Der Balken im Bild entspricht 5 Mikrometer (Millionstel Meter).
© Hans Dieter Schmitt / Max-Planck-Institut für biophysikalische Chemie
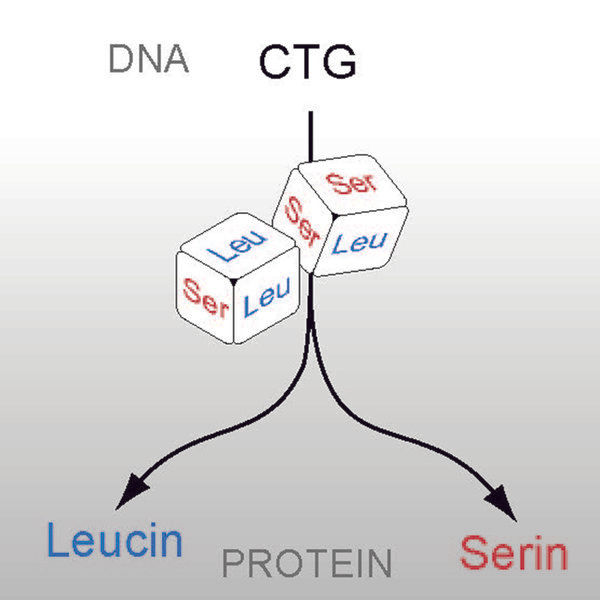
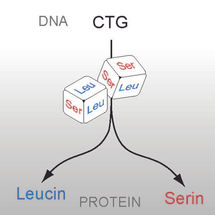
Ascoidea asiatica übersetzt das Codon CTG nach dem Prinzip Zufall in die Aminosäuren Leucin oder Serin.
© Martin Kollmar
Codes sind allgegenwärtig, auch wenn wir uns dessen häufig nicht bewusst sind. Digitale Daten etwa werden in einem Code aus Nullen und Einsen gespeichert, deren Abfolge sich in Ziffern, Zeichen oder Buchstaben übersetzt. Sprache und Schrift lassen sich ebenfalls als Codes betrachten, bei denen Laut- und Zeichenfolgen für bestimmte Bedeutungen stehen.
Allen Codes gemein ist, dass sie eindeutig sind: Einer bestimmten Zeichenfolge ist eine definierte Übersetzung zugeordnet. Ohne diese Regel könnten Codes nicht funktionieren, da sie sonst nicht interpretierbar wären.
Anfang der 1960er-Jahre entdeckten Biochemiker, dass die Abfolge der vier Bausteine A, C, G und T in unserer DNA der Zelle vorschreibt, in welcher Sequenz sie Aminosäuren zu Proteinen zusammenbaut. Damit hatten sie den wohl ältesten Code der Welt geknackt, den genetischen Code. Auch dieser schien der elementaren Regel der Eindeutigkeit zu gehorchen: Eine Sequenz von jeweils drei DNA-Bausteinen – Codon genannt – wird von der Zelle in eine spezifische Aminosäure übersetzt. Das Codon CTG steht beispielsweise in den meisten Lebewesen für die Aminosäure Leucin. Da es mehr unterschiedliche Codone als Aminosäuren gibt, sind den meisten Aminosäuren mehrere Codone zugeordnet. Leucin beispielsweise wird ebenfalls von CTA, CTT und CTC kodiert. Wissenschaftler nennen das Redundanz.
Bei manchen Lebewesen findet sich eine bemerkenswerte Variation des genetischen Codes: Sie übersetzen das Codon CTG in die Aminosäuren Serin oder Alanin. Bei einer Gruppe von Hefen war bislang unklar, welche Aminosäure CTG zugeordnet ist. Ein internationales Forscherteam um den Göttinger Bioinformatiker Martin Kollmar in Zusammenarbeit mit Henning Urlaub, der am MPI für biophysikalische Chemie die Forschungsgruppe Bioanalytische Massenspektrometrie leitet, analysierte daher die Proteinsequenzen von sieben Hefearten dieser Gruppe. Bei sechs der Arten fanden sie eine eindeutige Zuordnung des CTG-Codons zu den Aminosäuren Serin oder Alanin. „Bei Ascoidea asiatica erwartete uns allerdings eine Überraschung: Diese Hefe übersetzt CTG sowohl als Leucin als auch als Serin“, berichtet Kollmar.
Übersetzung nach dem Prinzip Zufall
„Für unsere Beobachtung kamen prinzipiell zwei Erklärungen infrage“, erläutert Stefanie Mühlhausen, Postdoktorandin im Labor von Laurence D. Hurst an der University of Bath und Erstautorin der jetzt im Fachjournal Current Biology erschienenen Arbeit. „Entweder bestimmen die Codone, die CTG auf der DNA umgeben, ob die Zelle es in Leucin oder Serin übersetzt. In diesem Fall müsste in einem Protein an der entsprechenden Position immer Leucin oder immer Serin eingebaut werden. Die Interpretation des genetischen Codes wäre so trotzdem eindeutig. Oder aber die Hefe überlässt es dem Zufall, ob CTG in Leucin oder Serin übersetzt wird.“
Weitere Experimente zeigten: Letzteres trifft zu. „Ascoidea asiatica übersetzt CTG tatsächlich stochastisch je zur Hälfte in Leucin und Serin“, sagt Hans Dieter Schmitt, Projektgruppenleiter am MPI für biophysikalische Chemie. Damit war klar: Ascoidea asiatica bricht mit der Eindeutigkeits-Regel von Codes.
Den Wissenschaftlern drängte sich nun die Frage auf: Wie kommt der Organismus damit zurecht? „Wenn er selbst gar nicht beeinflussen kann, wann welche Aminosäure eingebaut wird, müsste das zu Chaos führen“, gibt Kollmar zu bedenken. „Aber offenbar kommt die Hefe gut zurecht.“
Die Lösung heißt: vermeiden
Auf der Suche nach einer Antwort berechneten die Bioinformatiker, wie häufig Ascoidea asiatica das problematische Codon verwendet. Das Ergebnis war erstaunlich: Die Hefe nutzt CTG nur in etwa einem Prozent aller Fälle, in denen Serin oder Leucin in einem Protein auftaucht. Bei verwandten Arten liegt der Prozentsatz bei zehn bis 30 Prozent. Bei den restlichen 99 Prozent profitiert die Hefe von der Redundanz des genetischen Codes und greift auf die anderen Codone zurück, die eindeutig für die Aminosäuren Leucin und Serin kodieren. Außerdem setzt Ascoidea asiatica das CTG-Codon überwiegend in seltenen Proteinen ein und nie an den „wichtigen“ Stellen eines Proteins, wo sich ein Wechsel der Aminosäure auf die Funktionalität auswirken könnte. „Ascoidea asiatica hat also eine Vermeidungsstrategie entwickelt“, fasst Kollmar zusammen.
Dafür hatte die Hefe reichlich Zeit: Die Forscher schätzen, dass die Uneindeutigkeit des genetischen Codes bei Ascoidea asiatica bereits vor etwa 100 Millionen Jahren entstand. Möglicherweise waren davon auch verwandte Arten betroffen, die im Laufe der Zeit aber zum eindeutigen Code zurückkehrten.
Originalveröffentlichung
Stefanie Mühlhausen, Hans Dieter Schmitt, Kuan-Ting Pan, Uwe Plessmann, Henning Urlaub, Laurence D. Hurst, Martin Kollmar; "Endogenous Stochastic Decoding of the CUG Codon by Competing Ser- and Leu-tRNAs in Ascoidea asiatica"; Current Biology; 2018