Geregelter Nano-Aufbau
Universelle pH-Steuerung der Assemblierung von DNA-Nanostrukturen
Anzeigen
Die DNA, Träger der genetischen Information, hat sich als hochinteressantes Baumaterial für die Nanotechnologie etabliert. Voraussetzung für viele mögliche Anwendungen ist eine kontrolliert schaltbare Zusammenlagerung der Nanostrukturen, etwa durch eine Änderung des pH-Wertes. Wissenschaftler stellen eine neue Strategie für eine solche Steuerung vor. Sie basiert auf Ethylendiamin, das nur in neutralem bis saurem Milieu die Zusammenlagerung von DNA-Bausteinen unterstützt – unabhängig von der Basensequenz und ohne Anwesenheit von Metallionen.
© Wiley-VCH
Ein Satz verschiedener kurzer DNA-Einzelstränge lässt sich zu Bauelementen verknüpfen, die dann selbstgesteuert über eine Kohäsion komplementärer Abschnitte zu einer breiten Palette von Geometrien aggregieren. Diese Assemblierung von DNA-Bausteinen ahmt einen Kristallisationsprozess nach. Sind die richtigen Stränge designt, bildet sich die spezifische Struktur per Selbstorganisation. Man hofft, auf dieser Basis in Zukunft Nanomaterialien oder auch Nanoroboter zu konstruieren, die winzige Eingriffe in erkrankten Organen oder gar Zellen vornehmen. Auch eine zukünftige Nanoelektronik oder Nanokatalyse könnten Einsatzgebiete sein.
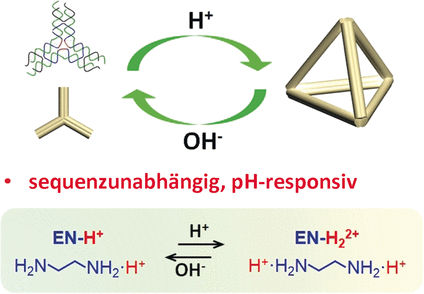
Eine kontrolliert schaltbare Zusammenlagerung der Nanostrukturen gelingt mittels bestimmter DNA-Strukturmotive, die ihre Form pH-abhängig verändern. Sie basiert auf ganz speziellen Basen-Sequenzen. Eine Sequenz-unabhängige Steuerung würde Zugang zu einer universellen Methode für die Selbstassemblierung von DNA eröffnen und damit den Anwendungsbereich der dynamischen DNA-Nanotechnologie erheblich erweitern.
Die Wissenschaftler von der Hefei University of Technology, der University of Science and Technology of China in Hefei, und der Purdue University, West Lafayette (USA) setzen dafür auf ein kleines organisches Molekül: Ethylendiamin H2N—CH2—CH2—NH2. In Wasser binden je nach pH-Wert eine oder beide Aminogruppen reversibel ein Proton (H+). Die Mengenverhältnisse der drei Spezies hängen stark vom pH-Wert ab. Ethylendiamin-Moleküle mit zweifach positiver Ladung sind in der Lage, die negativen Ladungen des DNA-Rückgrats elektrostatisch so abzuschirmen, dass sie sich weniger abstoßen, was eine Zusammenlagerung begünstigt.
In der Tat gelang es dem Team um Yulin Li, Zhaoxiang Deng und Chengde Mao, in einem Ethylendiamin-haltigen Puffer einzelne kreuzförmige DNA-Bausteine in neutralem bis saurem Milieu zu zweidimensionalen Wabenstrukturen zu assemblieren. Im leicht Alkalischen entstanden dagegen keine ausgedehnten Strukturen. Als Beispiel einer dreidimensionalen Struktur stellten sie Tetraeder-förmige Käfige aus Bausteinen in Form dreizackiger Sterne her. Bei pH 6,5 ließen sich die Sterne reversibel in Tetraeder umwandeln, bei pH 8 zurück in Sterne oder einzelne Stränge.
Während zur Ladungsabschirmung der DNA sonst meist zweifach positiv geladene Metallionen wie Mg2+ eingesetzt werden, kommt der Ethylendiamin-Puffer ohne Metallionen aus. Metallionen verstärken die Aktivität vieler Enzyme, vor allem DNA-spaltender Nucleasen. Das neue System ist damit für einen Einsatz in Gegenwart von Enzymen besser geeignet.
Originalveröffentlichung
Yongfei Li, Lei Song, Bang Wang, Prof. Jianbo He, Prof. Yulin Li, Prof. Zhaoxiang Deng, Prof. Chengde Mao; "Universal pH‐Responsive and Metal‐Ion‐Free Self‐Assembly of DNA Nanostructures"; Angew. Chem.; 2018
Yongfei Li, Lei Song, Bang Wang, Prof. Jianbo He, Prof. Yulin Li, Prof. Zhaoxiang Deng, Prof. Chengde Mao; "Universal pH‐Responsive and Metal‐Ion‐Free Self‐Assembly of DNA Nanostructures"; Angew. Chem. Int. Ed.; 2018


























































