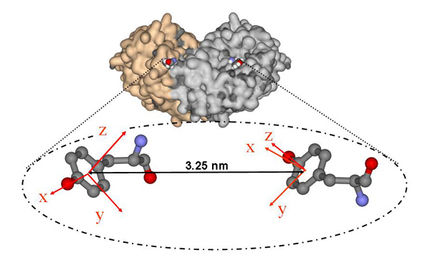
Proteinstruktur des E. coli Bakteriums entschlüsselt
Internationaler Forschergruppe gelingt dank neuer Methodik die strukturelle Aufklärung großer Membranproteine
Mithilfe einer neuen Methodik der NMR-Spektroskopie ist es Wissenschaftlern erstmals gelungen, die dreidimensionale Struktur eines sehr großen Membranproteins des E. coli Bakteriums aufzuklären. In der aktuellen Online-Ausgabe des Wissenschaftsmagazins Science vom 26. Juni veröffentlichen sie die Ergebnisse ihrer fast 10-jährigen Forschungs- und Entwicklungsarbeit.
Neben der technischen Verfeinerung der Methodik sind die Ergebnisse gleichzeitig von großer medizinischer Bedeutung in der Arzneimittelforschung. Sie bilden die Grundlage für die Entwicklung gezielt wirksamer Medikamente etwa in der Krebsforschung.
Die NMR-Spektroskopie (Nuclear Magnetic Resonance) ist eine weit verbreitete Analysemethode. In der Biochemie ermöglicht sie es, Proteine wie zum Beispiel die des E. coli Bakteriums nachzuweisen, sie im Detail zu analysieren und dreidimensional in Lösung darzustellen. Über die dreidimensionalen Strukturen erhalten die Wissenschaftler Auskunft über die Funktionsweise der Proteine.
Je größer ein Protein ist, desto problematischer ist seine Analyse. Besonders schwierig zu bestimmen sind darüber hinaus Proteine, die in Zellmembranen vorkommen. Unter den zurzeit rund 58.000 aufgeklärten Proteinstrukturen befinden sich bisher nur etwa 100-150 kleinere Membranproteine. Für das zelluläre Leben sind sie jedoch essentiell. Sie sind verantwortlich für den Transport von Nährstoffen und Signalen. Das heißt, sie kommunizieren mit der Umgebung. Daher sind sie für die Wissenschaft von großem Interesse.
Dr. Frank Sönnichsen vom Otto Diels-Institut für Organische Chemie der Christian-Albrechts-Universität (CAU) und seinen Kollegen ist es beim E. coli Bakterium gelungen, ein sehr großes Membranprotein zu entschlüsseln. Es besteht aus 360 Aminosäuren mit neun Helixen. E. coli ist ein säurebildendes Bakterium, das im menschlichen und tierischen Darm vorkommt.
"Die neue Methodik ist eine bedeutende und wichtige Erweiterung unseres technischen Arsenals", sagt Sönnichsen, Co-Autor der neuen Science-Studie. "Mit ihr hoffen wir künftig auch die Eigenschaften großer menschlicher Proteine entschlüsseln zu können - etwa aus den Bereichen der Brustkrebs- oder Alzheimerforschung." Eine Kenntnis der dreidimensionalen Struktur sei vor allem für die Entwicklung neuer Heilmittel relevant.
Momentan binden sich etwa 60 Prozent aller Arzneimittel an Membranproteine. Jedoch sind erst wenige in ihrer genauen Struktur und Funktion erforscht: "Die Medikamente wirken zwar, doch wir wissen nicht immer, wie sie es tun", sagt Sönnichsen. "Mit unserer neuen Methodik legen wir die Grundlage für die Wirkstoffforschung, gezielter wirkende Heilmittel zu entwickeln und so auch Nebenwirkungen zu reduzieren."
An der Studie, die Charles R. Sanders, Professor für Biochemie an der Vanderbilt Universität in Nashville, USA, koordiniert hat, sind neun Autoren beteiligt. Neben den Kollegen aus den USA, Korea und China ist der Kieler Chemiker Sönnichsen einziger europäischer Vertreter.
Meistgelesene News
Themen
Organisationen
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

Antibody Stabilizer von CANDOR Bioscience
Protein- und Antikörperstabilisierung leicht gemacht
Langzeitlagerung ohne Einfrieren – Einfache Anwendung, zuverlässiger Schutz

DynaPro NanoStar II von Wyatt Technology
NanoStar II: DLS und SLS mit Touch-Bedienung
Größe, Partikelkonzentration und mehr für Proteine, Viren und andere Biomoleküle

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.