Une coupe de précision pour les ciseaux génétiques CRISPR-Cas
Une nouvelle étude indique comment l'apprentissage profond peut améliorer les thérapies géniques et les médicaments antiviraux
La nucléase Cas13b associée aux systèmes de défense CRISPR - également connus sous le nom de ciseaux génétiques - pourrait être utilisée à l'avenir dans les maladies héréditaires pour réduire au silence les gènes indésirables. Dans la lutte contre les infections, elle fait également l'objet de recherches comme agent antiviral, car Cas13b peut cibler le génome des virus et les rendre inoffensifs. Malgré ces caractéristiques prometteuses, les chercheurs sont à la recherche d'inhibiteurs de nucléases capables de contrôler ou d'arrêter ces activités. L'objectif est d'accroître la sécurité et l'efficacité des futures thérapies et d'aider à prévenir les effets hors cible. Une équipe internationale dirigée par l'Institut Helmholtz de Würzburg, en coopération avec l'Université de Fribourg, vient d'appliquer pour la première fois l'apprentissage profond pour trouver des inhibiteurs de nucléases naturels. L'étude, publiée dans la revue scientifique "Molecular Cell", a identifié le premier inhibiteur de ce type qui bloque l'activité de Cas13b.
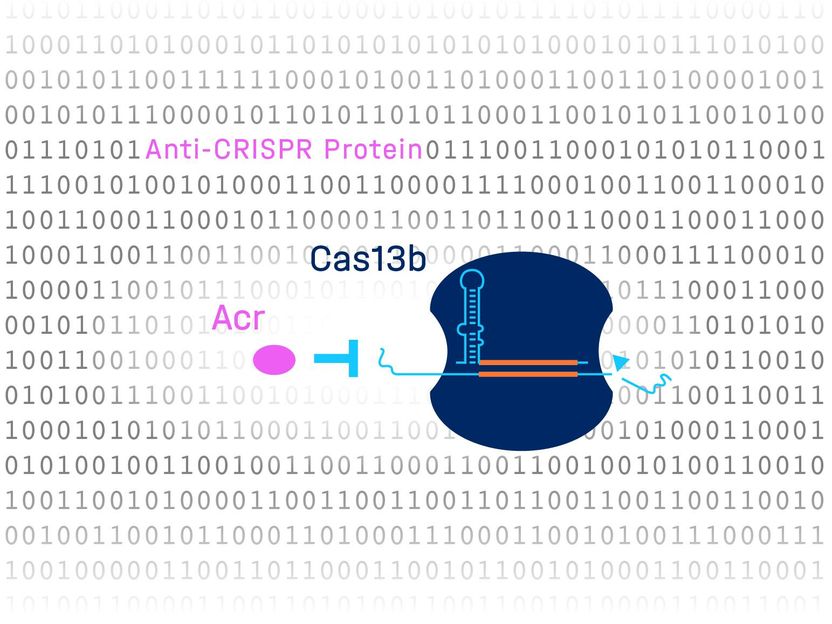
Grâce à un apprentissage computationnel complexe associé à un crible à haut débit, une nouvelle protéine anti-CRISPR a été découverte, qui inhibe Cas13b.
HIRI/Chase Beisel
Les bactéries contiennent souvent des systèmes de défense CRISPR-Cas qui les protègent contre les envahisseurs viraux appelés phages. En réponse, les phages ont développé des protéines capables de bloquer CRISPR. Ces protéines sont appelées protéines anti-CRISPR, ou Acrs. Les chercheurs pensent qu'il existe de nombreuses Acrs qui n'ont pas encore été découvertes. L'un d'entre eux est Chase Beisel, chef du département de biologie synthétique de l'ARN à l'Institut Helmholtz de recherche sur les infections à base d'ARN (HIRI) de Würzburg, un site du Centre Helmholtz de recherche sur les infections (HZI) de Braunschweig en coopération avec la Julius-Maximilians-Universität (JMU) de Würzburg. "Les Acrs peuvent être utilisés pour mieux contrôler les technologies CRISPR", déclare Beisel, tout en reconnaissant que "les identifier revient à trouver une aiguille dans une botte de foin, car ils ne se ressemblent pas du tout." Cela, dit-il, a conduit les études scientifiques du passé à proposer de nouveaux outils de prédiction. Toutefois, le succès de ces méthodes a été limité, selon M. Beisel. Il est à l'origine de l'étude publiée dans la revue "Molecular Cell".
Premier exemple d'utilisation de l'apprentissage profond pour la découverte d'Acr
Par conséquent, une équipe de chercheurs du HIRI de Würzburg, ainsi que des scientifiques des centres de recherche sur la signalisation de l'université de Fribourg et du département des sciences de l'information et de l'informatique de l'université King Fahd en Arabie saoudite, ont été les premiers à utiliser l'intelligence artificielle pour identifier de nouveaux Acr.
"Grâce à un apprentissage informatique complexe combiné à un crible à haut débit, nous avons réussi à découvrir une nouvelle protéine anti-CRISPR qui inhibe la nucléase Cas13b, cliniquement intéressante", explique Katharina Wandera. La doctorante du laboratoire de Chase Beisel travaille sur la caractérisation d'Acrs et est l'un des premiers auteurs de l'article actuel.
"Les millions de prédictions réalisées par notre algorithme ouvrent de toutes nouvelles voies de recherche", assure Rolf Backofen, de l'université de Fribourg. Son groupe a développé la méthode, appelée DeepAcr, pour l'étude.
Les résultats suggèrent également que DeepAcr peut non seulement être utilisé pour découvrir de nouvelles protéines qui inhibent les systèmes CRISPR-Cas dépourvus d'Acrs connus. "La protéine anti-CRISPR identifiée, AcrVIB1, était non seulement inconnue jusqu'à présent, mais elle semble également utiliser un mécanisme d'action totalement nouveau", résume Omer Alkhnbashi, professeur adjoint à l'université King Fahd, qui partage la paternité de l'étude avec Wandera.
La caractérisation de ce mécanisme d'action est l'une des prochaines étapes qui vont maintenant suivre les travaux scientifiques.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.

































