Las estructuras consideradas clave para la expresión de los genes son sorprendentemente fugaces
Un estudio revela que los bucles del genoma no duran mucho tiempo en las células; es posible que haya que revisar las teorías sobre cómo los bucles controlan la expresión de los genes
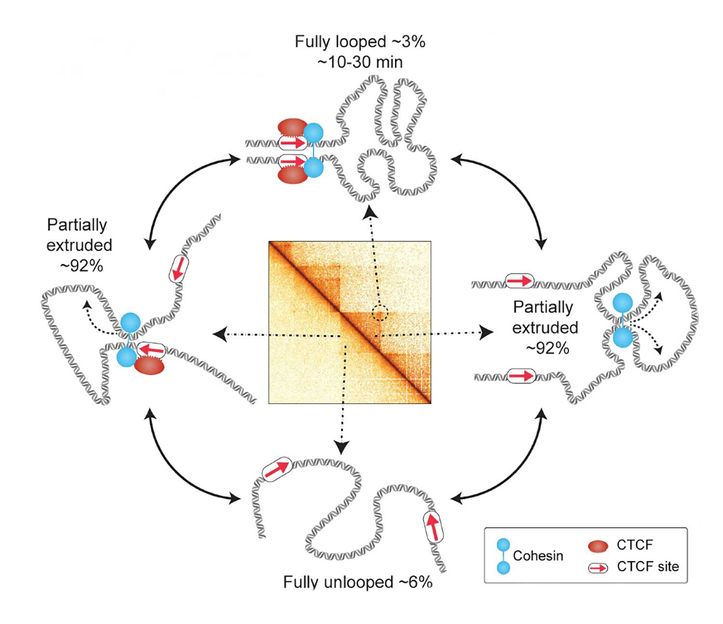
En los cromosomas humanos, el ADN está recubierto por proteínas para formar una cadena de cuentas extremadamente larga. Esta "cuerda" se pliega en numerosos bucles, que se cree que ayudan a las células a controlar la expresión de los genes y facilitar la reparación del ADN, entre otras funciones. Un nuevo estudio del Instituto Tecnológico de Massachusetts (MIT), en colaboración con el Instituto Max Planck de Biología Celular Molecular y Genética (MPI-CBG) de Dresde (Alemania) y el Centro de Biología de Sistemas de Dresde (CSBD), sugiere que estos bucles son muy dinámicos y de vida más corta de lo que se pensaba. En el nuevo estudio, los investigadores pudieron controlar el movimiento de un tramo del genoma en una célula viva durante unas dos horas. Comprobaron que este tramo estaba completamente en bucle sólo entre el 3 y el 6 por ciento del tiempo, y que el bucle sólo duraba entre 10 y 30 minutos. Los hallazgos sugieren que la comprensión actual de los científicos sobre cómo los bucles influyen en la expresión génica puede necesitar ser revisada, dicen los investigadores.
Las imágenes de súper resolución de células vivas de los bucles de cromatina mediados por CTCF/Cohesina revelan que estos bucles son raros y dinámicos.
© Gabriele et al., Science
"Muchos modelos en el campo han sido estas imágenes de bucles estáticos que regulan estos procesos. Lo que nuestro nuevo trabajo muestra es que esta imagen no es realmente correcta", dice Anders Sejr Hansen, profesor asistente de desarrollo de carrera Underwood-Prescott de Ingeniería Biológica en el MIT. "Sugerimos que el estado funcional de estos dominios es mucho más dinámico". Hansen es uno de los autores principales del nuevo estudio, junto con Leonid Mirny, profesor del Instituto de Ingeniería y Ciencias Médicas del MIT y del Departamento de Física, y Christoph Zechner, jefe de grupo del MPI-CBG y del CSBD. Michele Gabriele, postdoctorante del MIT, Hugo Brandão, recién doctorado por la Universidad de Harvard, y Simon Grosse-Holz, estudiante de posgrado del MIT, son los autores principales del artículo, que aparece hoy en Science.
Fuera de onda
Mediante simulaciones informáticas y datos experimentales, los científicos del grupo de Mirny en el MIT han demostrado que los bucles en el genoma se forman mediante un proceso llamado extrusión, en el que un motor molecular promueve el crecimiento de bucles progresivamente mayores. El motor se detiene cada vez que encuentra una "señal de parada" en el ADN. El motor que extruye estos bucles es un complejo proteico llamado cohesina, mientras que la proteína CTCF unida al ADN sirve de señal de parada. Estos bucles mediados por la cohesina entre los sitios de CTCF se observaron en experimentos anteriores. Sin embargo, esos experimentos sólo ofrecían una instantánea de un momento en el tiempo, sin información sobre cómo cambian los bucles a lo largo del tiempo. En su nuevo estudio, los investigadores desarrollaron técnicas que les permitieron marcar con fluorescencia los sitios de ADN de CTCF para poder obtener imágenes de los bucles de ADN durante varias horas. También crearon un nuevo método computacional que permite inferir los bucles a partir de los datos de las imágenes. "Este método fue crucial para distinguir la señal del ruido en nuestros datos experimentales y cuantificar los bucles", dice Zechner. "Creemos que estos enfoques serán cada vez más importantes para la biología a medida que sigamos ampliando los límites de la detección con experimentos".
Los investigadores utilizaron su método para obtener imágenes de un tramo del genoma en células madre embrionarias de ratón. "Si ponemos nuestros datos en el contexto de un ciclo de división celular, que dura unas 12 horas, el bucle completamente formado sólo existe en realidad durante unos 20 a 45 minutos, es decir, entre el 3 y el 6 por ciento del tiempo", afirma Grosse-Holz.
"Si el bucle sólo está presente durante un periodo tan diminuto del ciclo celular y dura muy poco, no deberíamos pensar que este estado de bucle completo es el principal regulador de la expresión génica", afirma Hansen. "Creemos que necesitamos nuevos modelos sobre cómo la estructura 3D del genoma regula la expresión génica, la reparación del ADN y otros procesos funcionales posteriores". Mientras que los bucles completamente formados eran raros, los investigadores encontraron que los bucles parcialmente extruidos estaban presentes alrededor del 92 por ciento de las veces. Estos bucles más pequeños han sido difíciles de observar con los métodos anteriores de detección de bucles en el genoma. "En este estudio, al integrar nuestros datos experimentales con simulaciones de polímeros, hemos podido cuantificar la extensión relativa de los estados de bucle no extruido, parcialmente extruido y totalmente extruido", afirma Brandão.
"Como estas interacciones son muy cortas, pero muy frecuentes, las metodologías anteriores no eran capaces de captar plenamente su dinámica", añade Gabriele. "Con nuestra nueva técnica, podemos empezar a resolver las transiciones entre los estados completamente en bucle y sin bucle".
Los investigadores plantean la hipótesis de que estos bucles parciales pueden desempeñar papeles más importantes en la regulación de los genes que los bucles completamente formados. Las hebras de ADN se entrecruzan cuando los bucles comienzan a formarse y luego se deshacen, y estas interacciones pueden ayudar a que elementos reguladores como los potenciadores y los promotores de los genes se encuentren entre sí. "Más del 90 por ciento de las veces, hay algunos bucles transitorios, y presumiblemente lo importante es tener esos bucles que están siendo extruidos perpetuamente", dice Mirny. "El proceso de extrusión en sí mismo puede ser más importante que el estado de bucle completo que sólo se produce durante un corto período de tiempo".
Más bucles que estudiar
Dado que la mayoría de los demás bucles del genoma son más débiles que el estudiado por los investigadores en este trabajo, sospechan que muchos otros bucles también resultarán ser muy transitorios. Ahora planean utilizar su nueva técnica para estudiar algunos de esos otros bucles, en una variedad de tipos de células. "Hay unos 10.000 bucles de este tipo, y nosotros hemos estudiado uno", dice Hansen. "Tenemos muchas pruebas indirectas que sugieren que los resultados serían generalizables, pero no lo hemos demostrado. Con la plataforma tecnológica que hemos creado, que combina nuevos métodos experimentales y computacionales, podemos empezar a abordar otros bucles del genoma." Los investigadores también planean investigar el papel de bucles específicos en las enfermedades. Muchas enfermedades, incluido un trastorno del neurodesarrollo llamado síndrome FOXG1, podrían estar relacionadas con una dinámica defectuosa de los bucles. Los investigadores están estudiando ahora cómo se ven afectadas por la formación de bucles en el genoma tanto la forma normal como la mutada del gen FOXG1, así como el gen MYC, causante del cáncer.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.